Hongos fitopatógenos foliares asociados al cultivo de Prunus persica L.: Identificación morfológica y molecular, y control biológico con Trichoderma asperelloides
DOI:
https://doi.org/10.17268/sci.agropecu.2025.013Palabras clave:
Análisis molecular, biocontrol, confrontación en planta, hongo, identificación, T. asperelloidesResumen
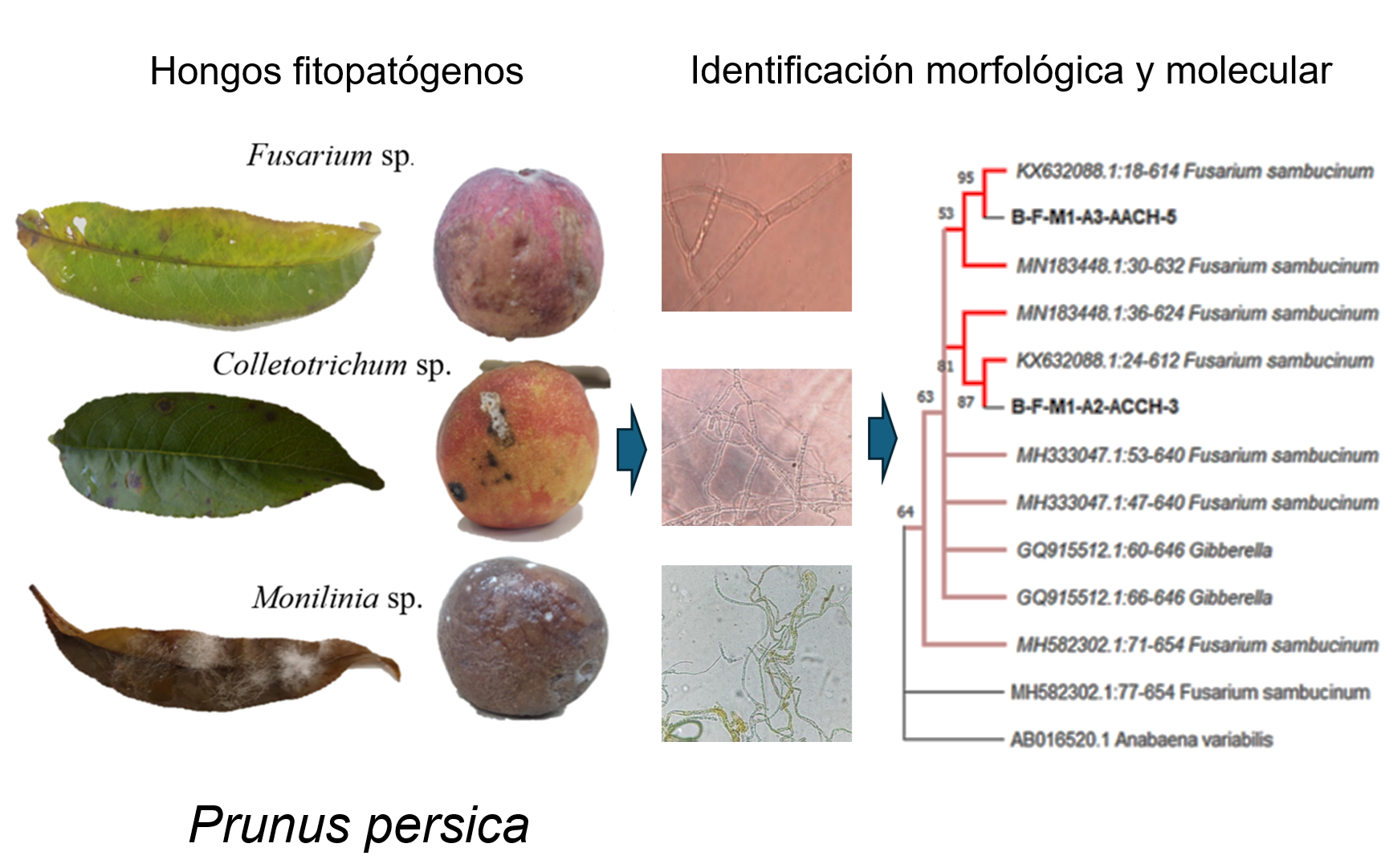
La presencia de hongos fitopatógenos foliares ocasiona severos daños en hojas y frutos de durazno (Prunus persica L.), en zonas productoras del sur de Chihuahua, México, lo que ha ocasionado una disminución en su producción en un 30%. El objetivo de este trabajo fue identificar morfológica y molecularmente los hongos fitopatógenos foliares asociados al cultivo del duraznero evaluando la patogenicidad en plantas de un año contra Trichoderma asperelloides. Se recolectaron hojas con lesiones color café y marrón en frutos momificados en planta de 19 huertos comerciales de durazno distribuidos en tres municipios de producción regional y nacional en el Estado de Chihuahua México. La identificación de hongos de cuatro aislamientos representativos se realizó utilizando métodos morfológicos. caracterización y análisis filogenético basado en la región espaciadora transcrita interna (ITS1 e ITS4) de ADN ribosomal, parte del factor de elongación de traducción 1-alfa (TEF) un segundo cebador secundario por cada uno de los géneros para, Collectotrichum ACT-512F y ACT-583R, Fusarium con EF1. Para las confrontaciones en planta se inoculo una concentración de 1x106 conidios. mL-1 de patógenos como de T. asperelloides, evaluando el diámetro y longitud de hoja, altura, severidad e incidencia. Se logró identificar la presencia de Fusarium sambucinum, Collectotrichum gleosporoides y Monilinia frutícola, además la cepa B-F-M1-A2-ACCH-3 de F. Sambucinum obtuvo los valores más altos en la inhibición de las variables de respuesta y en severidad Monilinia frutícola presentó 61,23% como el valor más alto. Se recomienda usar la cepa 3 como control biológico de fitopatógenos foliares.
Citas
Agu, K. C., & Chidozie, C. P. (2021). An improved slide culture technique for the microscopic identification of fungal species. International Journal of Trend in Scientific Research and Development, 6(1), 243-254.
Almaraz-Sánchez, A., Ayala-Escobar, V., Tlatilpa-Santamaría, I. F., & Nieto-Angel, D. (2018). Fusarium sambucinum Fuckel agente causal de la pudrición de frutos de chile manzano (Capsicum pubescens) en México. Revista Mexicana de Fitopatología, 37(1). https://doi.org/10.18781/r.mex.fit.1810-2
Arafat, K. H., Hassan, M., & Hussein, E. A. (2021). Detection, disease severity and chlorophyll prediction of date palm leaf spot fungal diseases. New Valley Journal of Agricultural Science, 1(2), 98-110.
https://doi.org/10.21608/nvjas.2022.110022.1027
Baltazar, E., Rodrigues, S., Ares, A., Camelo, A., Brandão, I., et al. (2023). Morphological, Molecular and Genomic Identification and Characterisation of Monilinia fructicola in Prunus persica from Portugal. Agronomy, 13(6), 1493. https://doi.org/10.3390/agronomy13061493
Battistini, G., Gazzetti, K., & Collina, M. (2022). A New Approach: Determining cyt b G143A Allele Frequency in Zymoseptoria tritici by Digital Droplet PCR. Biology, 11(2), 240. https://doi.org/10.3390/biology11020240
Boukaew, S., Chumkaew, K., Petlamul, W., Srinuanpan, S., Nooprom, K., & Zhang, Z. (2024). Biocontrol effectiveness of Trichoderma asperelloides SKRU-01 and Trichoderma asperellum NST-009 on postharvest anthracnose in chili pepper. Food Control, 163, 110490. https://doi.org/10.1016/j.foodcont.2024.110490
Da Silva Neto, J. A., De Queiroz Ambrósio, M. M., Araújo, M. B. M., Da Silva, R. M., Pinto, P. S. L., & Holanda, I. S. A. (2022). Morphological, molecular and pathogenic characterization of Colletotrichum gloeosporioides isolated from mango. Revista Caatinga, 35(3), 514-527. https://doi.org/10.1590/1983-21252022v35n302rc
Dini, M., Raseira, M., Scariotto, S., Marchi, P., & Mello-Farias, P. (2021). Research article peach phenological characters: heritability, maternal effect and correlation with brown rot. Genetics and Molecular Research, 20(1). https://doi.org/10.4238/gmr18684
Dong, J., Shi, H., Wu, Y., Yang, L., Zhu, F., & Ji, Z. (2023). Identification and pathogenicity analysis of Fusarium spp. on peach in China. BMC Microbiology, 23, 211. https://doi.org/10.1186/s12866-023-02958-y
Edgar, R. C. (2004). Muscle: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research, 32(5), 1792-1797. https://doi.org/10.1093/nar/gkh340
Gerardo-Lugo, S. S., Tovar-Pedraza, J. M., Maharachchikumbura, S. S., Apodaca-Sánchez, M. A., Correia, K. C., et al. (2020). Characterization of Neopestalotiopsis species associated with mango grey leaf spot disease in Sinaloa, Mexico. Pathogens, 9(10), 788. https://doi.org/10.3390/pathogens9100788
Gununu, P. R., Munhuweyi, K., Obianom, P. C., & Sivakumar, D. (2019). Assessment of eleven South African peach cultivars for susceptibility to brown rot and blue mould. Scientia Horticulturae, 254, 1-6. https://doi.org/10.1016/j.scienta.2019.04.067
Grano-Maldonado, M. I., Ramos-Payan, R., Rivera-Chaparro, F., Aguilar-Medina, M., Romero-Quintana, J. G., Rodríguez-Santiago, A., & Nieves-Soto, M. (2021). First Molecular Characterization of Colletotrichum sp. and Fusarium sp. Isolated from Mangrove in Mexico and the Antagonist Effect of Trichoderma harzianum as an Effective Biocontrol Agent. Plant Pathology Journal, 37(5), 465-475. https://doi.org/10.5423/ppj.oa.03.2021.0048
Hall, T. A. (1999). BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic acids symposium series, 41(49), 95-98.
Heflish, A. A., Abdelkhalek, A., Al-Askar, A. A., & Behiry, S. I. (2021). Protective and Curative Effects of Trichoderma asperelloides Ta41 on Tomato Root Rot Caused by Rhizoctonia solani Rs33. Agronomy, 11(6), 1162. https://doi.org/10.3390/agronomy11061162
Huang, R., Sun, W., Wang, L., Li, Q., Huang, S., et al. (2021). Identification and characterization of Colletotrichum species associated with anthracnose disease of banana. Plant Pathology, 70(8), 1827-1837. https://doi.org/10.1111/ppa.13426
Huilotl-Luna, M. L., Gil-Muñoz, A., Hernández-Romero, E., López, P. A., & Martínez-Carrera, D. C. (2024). Manejo de la pudrición café (Monilinia fructicola) del duraznero por productores de la Sierra Nevada de Puebla. Agricultura Sociedad y Desarrollo, 21(4). https://doi.org/10.22231/asyd.v21i4.1679
Iñiguez-Moreno, M., Sandoval-Contreras, T., Ragazzo-Sánchez, J. A., & Calderón-Santoyo, M. (2023). Modelado del crecimiento de hongos fitopatógenos aislados de frutos de aguacate Hass. Acta de Ciencia en Salud, 20(7), 1-5.
Iqbal, S., Abbas, A., Mubeen, I., Sathish, M., Razaq, Z., Mubeen, M., Kamran, M., Haroon, M., Syed, S., Naqvi, S., & Ahmed, M. A. A. (2022). Taxonomy, distribution, epidemiology, disease cycle and management of brown rot disease of peach (Monilinia spp.). Notulae Botanicae Horti Agrobotanici Cluj-Napoca, 50(1), 12630. https://doi.org/10.15835/nbha50112630
Karlsson, I., Edel-Hermann, V., Gautheron, N., Durling, M. B., Kolseth, A. K., et al. (2016). Genus-specific primers for study of Fusarium communities in field samples. Applied and environmental microbiology, 82(2), 491-501. https://doi.org/10.1128/AEM.02748-15
Lanfear, R., Calcott, B., Ho, S. y. W., & Guindon, S. (2012). PartitionFinder: Combined Selection of Partitioning Schemes and Substitution Models for Phylogenetic Analyses. Molecular Biology And Evolution, 29(6), 1695-1701. https://doi.org/10.1093/molbev/mss020
Luo, C., Schnabel, G., Hu, M., & De Cal, A. (2022). Global distribution and management of peach diseases. Phytopathology Research, 4(1). https://doi.org/10.1186/s42483-022-00134-0
Mannai, S., & Boughalleb-M’Hamdi, N. (2022). In vitro and in planta potential effect of some indigenous antagonists against Fusarium and pythiaceous species associated with peach seedlings decline. Egyptian Journal of Biological Pest Control, 32(1). https://doi.org/10.1186/s41938-022-00540-8
Manganaris, G. A., Minas, I., Cirilli, M., Torres, R., Bassi, D., & Costa, G. (2022). Peach for the future: A specialty crop revisited. Scientia Horticulturae, 305, 111390. https://doi.org/10.1016/j.scienta.2022.111390
Moreno, L. J. P., Prado, D. A. A., & Herrera, S. Y. A. (2018). Hongos fitopatógenos asociados a enfermedades foliares de Cattleya, Miltoniopsis y Oncidium en víveros de fusagasugá (Cundinamarca, Co). Revista de Fitopatología Colombiana, 42(1), 13-16.
Özer, N., Uzun, H. İ., Aktürk, B., Özer, C., Akkurt, M., & Aydın, S. (2021). Resistance assessment of grapevine leaves to downy mildew with sporulation area scoring. European Journal Of Plant Pathology, 160(2), 337-348. https://doi.org/10.1007/s10658-021-02247-2
Ramírez-Cariño, H. F., Guadarrama-Mendoza, P. C., Sánchez-López, V., Cuervo-Parra, J. A., Ramírez-Reyes, T., Dunlap, C. A., & Valadez-Blanco, R. (2020). Biocontrol of Alternaria alternata and Fusarium oxysporum by Trichoderma asperelloides and Bacillus paralicheniformis in tomato plants. Antonie Van Leeuwenhoek, 113(9), 1247-1261. https://doi.org/10.1007/s10482-020-01433-2
Ren, F., Dong, W., & Yan, D. (2019). Organs, cultivars, soil, and fruit properties affect structure of endophytic mycobiota of pinggu peach trees. Microorganisms, 7(9), 322. https://doi.org/10.3390/microorganisms7090322
Rios-Hernández, T. A., Uc-Varguez, A., & Evangelista-Martínez, Z. (2021). Biological control of Fusarium oxysporum causal agent of gladiolus corm rot by streptomycetes. Revista Mexicana de Fitopatología, 39(3). https://doi.org/10.18781/r.mex.fit.2105-3
Silva-Neto, J. A. D., Ambrósio, M. M. D. Q., Araújo, M. B. M., Silva, R., Pinto, P. S. L., & Holanda, I. S. A. (2022). Morphological, molecular and pathogenic characterization of Colletotrichum gloeosporioides isolated from mango. Revista Caatinga, 35, 514-527. https://doi.org/10.1590/1983-21252022v35n302rc
Stamatakis, A. (2006). RAxML-VI-HPC: maximum likelihood-based phylogenetic analyses with thousands of taxa and mixed models. Bioinformatics, 22(21), 2688-2690. https://doi.org/10.1093/bioinformatics/btl446
Statistical Analysis Software. (SAS). (2012). Users’ Guide Statistics Version 9.4. SAS Institute Inc., Cary. https://support.sas.com/software/94/
Tan, Q., Schnabel, G., Chaisiri, C., Yin, L. F., Yin, W. X., & Luo, C. X. (2022). Colletotrichum species associated with peaches in China. Journal of Fungi, 8(3), 313. https://doi.org/10.3390/jof8030313
Udhayakumar, R., Usharani, S., & Muthukumar, A. (2019). Pathogenicity variation, morphological and cultural characteristic of Colletotrichum gloeosporioides isolates. Plant Archives, 19(1), 425-430.
Voglmayr, H, Akulov, O. Y., & Jaklitsch, W. M. (2016). Reassessment of Allantonectria, phylogenetic position of Thyronectroidea, and Thyronectria caraganae sp. nov. Mycological Progress, 15(9), 921–937. https://doi.org/10.1007/s11557-016-1218-4
Wang, Q., Fan, K., Li, D., Han, C., Qu, Y., Qi, Y., & Wu, X. (2020). Identification, Virulence and Fungicide Sensitivity of Colletotrichum gloeosporioides s.s. Responsible for Walnut Anthracnose Disease in China. Plant Disease, 104(5), 1358-1368. https://doi.org/10.1094/pdis-12-19-2569-re
White, T., Bruns, T., Lee, S., & Taylor, J. (1990). Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. En Elsevier eBooks (pp. 315-322). https://doi.org/10.1016/b978-0-12-372180-8.50042-1
Yikilmazsoy, G., & Tosun, N. (2021). Characterization of Fusarium sambucinum isolates associated with potato dry rot and evaluation of cultivar susceptibility and fungicides. Turkish Journal of Agriculture and Forestry, 45(2), 222-233. https://doi.org/10.3906/tar-2006-100
Yin, L., Zhang, S., Du, J., Wang, X., Xu, W., & Luo, C. (2021). Monilinia fructicola on loquat: An old pathogen invading a new host. Journal Of Integrative Agriculture, 20(7), 2009-2014. https://doi.org/10.1016/s2095-3119(20)63375-5
Yin, L. F., Mo, W., Guo, D. Y., & Luo, C. X. (2020). First Report of Brown Rot of Prunus mume Caused by Monilinia fructicola in China. Plant Disease, 104(4), 1253. https://doi.org/10.1094/pdis-09-19-1847-pdn
Zhan, Q., Wang, N., Jin, S., Tan, R., Jiang, Q., & Wang, Y. (2018) ProbPFP: A multiple sequence alignment algorithm combining partition function and hidden markov model with particle swarm optimization. IEEE International conference on bioinformatics and biomedicine (BIBM) 1290-1295. https://doi.org/10.1109/BIBM.2018.8621220
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2025 Scientia Agropecuaria

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Los autores que publican en esta revista aceptan los siguientes términos:
a. Los autores conservan los derechos de autor y conceden a la revista el derecho publicación, simultáneamente licenciada bajo una licencia de Creative Commons que permite a otros compartir el trabajo, pero citando la publicación inicial en esta revista.
b. Los autores pueden celebrar acuerdos contractuales adicionales separados para la distribución no exclusiva de la versión publicada de la obra de la revista (por ejemplo, publicarla en un repositorio institucional o publicarla en un libro), pero citando la publicación inicial en esta revista.
c. Se permite y anima a los autores a publicar su trabajo en línea (por ejemplo, en repositorios institucionales o en su sitio web) antes y durante el proceso de presentación, ya que puede conducir a intercambios productivos, así como una mayor citación del trabajo publicado (ver efecto del acceso abierto).




