1. Introducción
El banano (Musa spp.) en las últimas décadas está amenazado por F. oxysporum f. sp. cubense (Foc), conocido como mal de Panamá (Siamak & Zheng, 2018). Esta enfermedad que ocasionó grandes pérdidas al cultivar Gros Michel Musa acuminata (AAA) a fines del siglo XIX (Gurdaswani et al., 2020). Las esporas ingresan a la planta a través de sus raíces y desarrollan hifas largas septadas, que se extienden hasta el xilema y bloquean el suministro de agua alrededor de la planta y provoca el marchitamiento (Niwas et al., 2022).
Los síntomas internos incluyen decoloración de amarillo a marrón rojizo de los tejidos vasculares (Dita et al., 2010). Los síntomas externos por Fusarium comienzan con el amarillamiento y el marchitamiento de las hojas más viejas y progresan hacia las hojas más jóvenes hasta que la planta muere (Izquierdo-García et al., 2021). El patógeno se puede diseminar a través de estructuras propagativas como microconidios, macroconidios y clamidosporas (Meldrum et al., 2013). Las clamidosporas tienen una pared celular engrosada que permanecen inactivas en suelos infestados durante décadas y permite hacer frente a condiciones ambientales extremas (Were et al., 2023).
Se consideran fuentes de diseminación de Fusarium sp. al material de siembra, afluentes de agua, partículas del suelo, herramientas, calzado y maquinaria (Magdama et al., 2019a). Existen tres razas que afectan al banano como son: Foc raza 1, que causa enfermedades en el cultivar ‘Gros Michel’ (AAA), también ataca a las variedades ‘Lady Finger’ (AAB) y ‘Silk’ (AAB). La raza 2 afecta a los bananos de cocción como 'Bluggoe' (ABB) y la raza 4 es capaz de atacar a ‘Cavendish’ (AAA) así como a las otras variedades de banano afectadas por las razas 1 y 2 (Martín et al., 2021).
Las Rizobacterias Promotoras de Crecimiento Vegetal (por sus siglas en inglés, PGPR) que habitan en el suelo, donde su funcionalidad es colonizar las raíces para mejorar el incremento de biomasa vegetal, crecimiento del sistema radicular, floración, contenido de clorofila y nutrientes del tejido foliar y el peso de brotes por la producción de Ácido Indol-3-Acético (AIA), Ácido Giberélico (AG) y Ácido Salicílico (AS) (Keswani et al., 2022; Macías Holguín et al., 2023a; Prisa et al., 2023; Canchignia-Martínez et al., 2025a). El mecanismo de biocontrol de las PGPR implica la producción enzimas como la quitinasas y proteasa, que degradan la pared celular fúngica a N-acetil D-glucosamina (Verma et al., 2023). Inducción de resistencia sistémica adquirida (SAR), inducción de resistencia sistémica (ISR) y producción de metabolitos antifúngicos como: Cianuro de Hidrógeno (HCN), Ácido Fenazina-1-Carboxílico (PCA), Pioluteorina (Plt), Pirrolnitrina (Prn) y 2,4-diacetilfloroglucinol (2,4 DAPG) que suprimen la germinación de conidias a enfermedades fitopatógenas (Crespo-Clas et al., 2024; Crespo Ávila et al., 2024; Canchignia-Martínez et al., 2025b).
Las herramientas moleculares, como la secuenciación, la reacción en cadena de la polimerasa, la caracterización molecular y la diversidad genética de las poblaciones de bacterianas son fundamentales para la identificación de especies y para comprender la dinámica de la microbiota del suelo (Rouhrazi & Khodakaramian, 2015; Getahun et al., 2020). Un método conocido a la variabilidad genética del microbioma es la técnica de PCR de consenso intergénico repetitivo de enterobacterias (ERIC-PCR) específica que es confiable, reproducible, rápida y altamente discriminatoria para la diferenciación de especies y subespecies (Odori et al., 2020; Auhing Arcos et al., 2021). El fraccionamiento de los productos de la PCR produce un patrón de huellas dactilares complejo con el que se pueden diferenciar las cepas bacterianas (Singh, 2014). Las secuencias ERIC reconocen en una gran cantidad de genomas bacterianos, incluidos miembros de la familia Enterobacteriaceae, Pseudomonadaceae, Moraxellaceae etc (Chen et al., 2011). Esta diversidad evoca los procesos de evolución entre cepas bacterianas dentro de una especie (Ranjbar et al., 2017). El análisis de identificación molecular de bacterias comienza por secuenciación del gen ARNr 16S. El gen codifica el componente de ARN de la subunidad 30S del ribosoma bacteriano que está presente en todas las bacterias se puede obtener información de distintos grupos filogenéticos, taxonómico e identificación a nivel de género y especie de las procariotas del 97% (Drancourt et al., 2000; da Silva et al., 2019; Satilmis et al., 2019).
El marchitamiento causado por F. oxysporum f. sp. cubense genera tres tipos de esporas, que incluyen: macroconidios, microconidios y clamidosporas siendo este último resistente a varios fungicidas sintéticos y generan un nuevo ciclo de infección. Al no existir un método de control químico eficaz para controlar la incidencia del mal de Panamá. Entre las alternativas se encuentra el uso de los agentes de control biológico de origen microbiano las cuales poseen características benéficas como la producción enzimas hidrolíticas y metabolitos antagónicos con distintos efectos antibacteriano, nematicida y antifúngicos. Donde el objetivo de este estudio se basa en el análisis filogenético al gen ARNr 16S y ERIC-PCR e identificación del gen ChiA de las PGPR como potencial empleo a la biosíntesis de fitohormonas (AIA, AG y AS) y actividad antagonista en desarrollo micelial y generación de esporas de FOC-R1.
2. Metodología
La investigación se realizó en el laboratorio de Biotecnología Molecular, ubicados en el Campus Experimental “La María” propiedad de la Universidad Técnica Estatal de Quevedo (UTEQ), ubicada en el km 7,5 de la vía Quevedo- Mocache, provincia de Los Ríos, Ecuador. Ubicada a 01º 04’ 48.6” de latitud sur y 79º 30’04.2” de longitud oeste y, a una altitud de 85 msnm.
Caracterización por PCR al gen ChiA
A la identificación de gen ChiA se seleccionaron 10 rizobacterias PGPR que mostraron antagonismo hacia Phytophthora palmivora y Lasiodiplodia theobromae. Estas bacterias se encuentran crioconservadas en glicerol al 10% a -40 °C del banco de germoplasma del Laboratorio de Microbiología y Biología Molecular de la UTEQ (Tabla 1). El ADNg se obtuvo al emplear PureLink™ Genomic DNA Mini Kit - Thermo Fisher Scientific, según el protocolo descrito por el fabricante. Para la amplificación molecular se empleó los primers específicos chiAF (5'GATATCGACTGGGAGTTCCC3') y chiAR (5'CATAGAAGTCGTAGGTCATC3'), con pro-ducto de amplificación 225 pb (Ramaiah et al., 2000).
Tabla 1
Mecanismo de acción de las rizobacterias
Cepa | Tejido Vegetal | | Mecanismo de acción |
Solubilización | | Antagonismo |
N | P | K | | PR | FLT | SFR |
BF 567 | R | ++ | +++ | ++ | | +++ | ++ | + |
FZ 9-7 | R | ++ | ++ | ++ | | +++ | +++ | + |
LH 5-10 | R | + | + | ++ | | ++ | +++ | + |
MH 18 | F | + | + | + | | ++ | + | + |
W-417 | F | + | + | + | | + | + | + |
MN 5-20 | F | + | + | ++ | | +++ | ++ | + |
MN 5-19 | F | + | + | + | | ++ | + | + |
AC3 | F | + | + | ++ | | ++ | + | + |
PV-25 | F | + | + | + | | + | + | + |
Tejido: R-radicular y F-foliar. Mecanismo de acción. Solubilización: N- nitrógeno, P- fósforo, K- potasio, Antagonismos: PR-actividad proteolítica, FLT-fluorescencia, SFR- producción de sideróforo (Canchignia-Martínez et al., 2024c).
La PCR se la realizo en un volumen de 20 µL con: 2 µL Dream taq Green buffer (1X), 1 µL dNTPs, 1,5 µL/cada primer, 0,2 µL Dream taq DNA polimerasa, 2 µL ADN, 13,3 µL H2O. La reacción de PCR se desarrolló en el termociclador (TECHME®). Las condiciones de la reacción de PCR: a 94 °C por 4 min; 35 ciclos a 92 °C por 1 min; 58 °C por 1 min; 72 °C por 1 min, extensión final de 72 °C por 7 min. Los productos amplificados se verificaron por electroforesis en gel de agarosa al 1,5% teñidos en bromuro de etidio. Los fragmentos se determinaron por comparación con el marcador de peso molecular 100 bp (Invitrogen®).
Identificación molecular por amplificación al ARNr 16S y generación de perfiles moleculares por ERIC-PCR
Se amplificó el gen para el ARN ribosomal 16S empleando partidores universales 8F (5’-AGAGTTTGATCCTGGCTCAG-3’) y 1492R (5’-ACG GCTACCTTGTTACGACTT-3’) que amplifica una región la subunidad ribosomal 16S de 1500 pb (Hernández-García et al., 2008). La PCR se realizó en un volumen de 20 µL con: 2 µL Dream Taq Green buffer (1X), 1 µL dNTPs, 1,5 µL/cada primer, 0,2 µL Dream taq DNA polimerasa, 2 µL ADN, 13,3 µL H2O. La reacción de PCR se desarrolló en el termociclador (TECHME®) con las condiciones térmicas: 94 ºC por 4 min; 40 ciclos a 92 ºC por 30 s; 58 ºC por 60 s; 72 ºC por 60 s; extensión final de 72 ºC por 10 min. Los productos amplificados se verificaron por electroforesis en gel de agarosa al 1,5% teñidos en bromuro de etidio. Los fragmentos se determinaron por comparación con el marcador de peso molecular 100 bp (Invitrogen®). Los productos amplificados se purificaron empleando el kit Invitrogen PureLink™ (GERMANY), siguiendo el protocolo descrito por el fabricante. Los fragmentos de 1500 pb, al ARNr 16S se secuenció por MACROGEN empleando los partidores universales 518F (5´-CCAGCAGCCGCGGTAATAC-´3) y 800R (3´-TACCAGGGTATCTAATCC-´5). por (Macrogen, Seúl, Korea). Las secuencias se compararon con la base de datos de GenBank de NCBI (https://www.ncbi.nlm.nih.gov/), empleando Basic Local Alignment Search Tool (BLAST). El árbol filogenético al gen ARNr 16s, se construyó empleando el método Maximun Likelihood y alineados con múltiples secuencias bacterianas disponible en NCBI. El porcentaje de la réplica del árbol se obtuvo con ‘bootstrap’ de valores de 1000 (Felsenstein, 1985). Se eligió el mejor modelo de acuerdo al criterio de información Bayesian (BIC), Kimura-2-parámetros con el programa MEGA 6.06 (Tamura et al., 2013).
El protocolo de ERIC-PCR se realizó a lo descrito por Louws et al. (1994) con algunas modificaciones. Con el empleo de los partidores ERIC-1R (5′ATGTAAGCTCCTGGGGATTCAC-3′) y ERIC-2F (5′AAGTAAGTGACTGGGGTGAGCG-3′). La PCR se realizó en un volumen de 20 µL con: 2 µL Dream taq Green buffer (1X), 1 µL dNTPs, 1,5 µL/cada primer, 0,2 µL Dream taq DNA polimerasa, 2 µL ADN, 13,3 µL H2O. La reacción de PCR se desarrolló en el termociclador (TECHME®). Las condiciones de la reacción de PCR: 94 °C por 3 min; 40 ciclos a 94 oC por 1 min; 37 oC por 2 min; 72 oC por 2 min; extensión final de 72 oC por 10 min. Los productos amplificados se verificaron por electroforesis en gel de agarosa al 1,5% teñidos en bromuro de etidio. Los fragmentos se determinaron por comparación con el marcador de peso molecular 100 bp (Invitrogen®). Los perfiles generados por ERIC-PCR, generó la información binaria de (0 y 1), reemplazando por las letras (A y T). La historia evolutiva se infirió empleando el método Maximun Likelihood, el árbol filogenético se construyó empleando la distancia evolutiva de Tamura 3-parametro con el programa MEGA 6.06. El porcentaje de la réplica del árbol se obtuvo con ‘bootstrap’ de valores de 1000 (Felsenstein, 1985). Se eligió el mejor modelo de acuerdo al criterio de información Bayesian (BIC), Kimura-2-parámetros con el programa MEGA 6.06 (Tamura et al., 2013).
Producción de ácido indol-3-acético (AIA)
Para el análisis de síntesis de AIA las rizobacterias se inocularon en medio de cultivo líquido King B descrito por King et al. (1954), [(g L-1): peptona, 20,0 g; glicerol, 15 mL; K2HPO4, 1,5 g; MgSO4 x 7H2O, 1,5 g;)] suplementado con 5 mM de L-triptófano (Trp) (Thermo Fisher Scientific) se incubaron a 150 rpm por 24 y 72 h a 28 °C para su muestreo. Para la cuantificación a producción de AIA, del cultivo bacteriano se cosechó 750 μL de sobrenadante centrifugadas a 10000 rpm durante 5 minutos. Se añadió 250 μL de la solución de Salkowski (3:1) y mantuvieron en oscuridad por 30 min (Gang et al., 2019). El cambio de coloración de rojo a rojo intenso muestra rasgo positivo de producción de AIA. La cantidad AIA (µg mL-1) fue cuantificada mediante espectrofotómetro "UNICO" modelo 1205 a 530 nm (Gordon & Weber, 1951). Se estableció una curva estándar con una hormona AIA comercial (Sigma-Aldrich) a 5, 10 15, 20, 25 y 30 µg mL-1 (Saker & Al-Rashid, 2013).
Producción de ácido giberélico (AG)
Las rizobacterias se cultivaron en caldo nutritivo [(g L-1): peptona 5 g; cloruro de sodio 5 g, extracto de carne 1,5 g, extracto de levadura 1,5 g, pH 7,4] suplementado con 3 mM de L-metionina (Merck) incubadas a 150 rpm por 24 y 72 h a 28 °C para su muestreo (Jaroszuk-Ściseł et al., 2019). Para la producción de giberelina se empleó el método estándar de Holbrook et al. (1961). Del cultivo bacteriano, se recuperó 5 mL del sobrenadante acidificado pH 2 con HCl 1N centrifugado por 5 min a 8000 rpm. Se agregó 2 mL de Zn(C₄H₆O₄) y 2 mL C₆FeK₄N₆ e incubadas a 27 °C durante 75 minutos. La cuantificación de AG se determinó a 254 nm utilizando el espectrofotómetro UV-Vis Genesys™ 10S y comparadas con una curva estándar de calibración a concentraciones de 10, 20, 40, 60, 80 100 μg mL-1 de ácido giberélico (Sigma-Aldrich) (Kesaulya et al., 2015).
Producción de ácido salicílico (AS)
Las bacterias se cultivaron en medio de cultivo casaminoácidos tamponados con Tris-HCl [(g L-1: Tris-HCl, 12 g; MgSO4x7H2O, 0,25 g; de K2HPO4, 0,9 g; casaminoácidos, 5 g; pH 7,5)] suplementado con 0.1 mM de FeCl3 e incubadas a 150 rpm por 24 y 72 h a 28 °C para su muestreo Mishra & Baek (2021). Del cultivo de células se centrifugó a 10000 rpm durante 10 min y se recuperaron 2 mL de sobrenadante acidificado pH 2. Se agregó 2 mL CHCl3 para la extracción de ácido salicílico en relación (1:1). A los extractos de cloroformo se le añadió 4 mL de H2O y 5 mL de FeCl3 a 2M (Meyer & Abdallah, 1978a). Las soluciones ácido salicílico-hierro púrpura se determinaron por espectro-fotometría "UNICO" modelo 1205 a 527 nm y una curva estándar con ácido salicílico (Thermo Scientific Chemical) a 10, 20, 40, 60, 80 100 μg mL-1 (Cook, 1993).
Identificación de F. oxysporum f. sp. cubense raza 1
Se recolectaron muestras de tejido necrótico vascular de Gros Michel (AAA) mismas que se llevaron al Laboratorio de Microbiología de la UTEQ. Las muestras se desinfectaron de forma superficial con etanol al 70% durante 30 s, NaClO al 5% por 1 min, lavadas con H2O estéril y segmentada en 1 cm2. Se colocaron 5 segmento de tejido vascular en placa Petri con PDA que contenían 120 mg mL-1 de estreptomicina y 250 mg mL-1 de cloranfenicol e incubadas a 28 °C durante cinco días para la obtención de cultivo monospórico de hongo del agente causal (Magdama et al., 2020b). Se observó la morfología de la colonia por su pigmentación y textura. Los caracteres morfológicos se visualizaron por microscopía óptica (ocular 8X y objetivo 100X) como la forma de las esporas microconidios, macroconidios y clamidosporas de Fusarium spp. descrito por Mostert et al. (2017).
Para la extracción de ADNg se tomaron fragmentos de micelio mismo que fueron pulverizados en N2 líquido siguiendo el protocolo descripto por el fabricante DNeasy Plant Mini Kit de (QIAGEN-Start EE.UU.). La identificación de Foc-R1 se realizó por PCR, se empleó los oligonucleótidos específicos W1805F (5’-GTTGAGTCTCGATAAACAGCAAT-3’) y W1805R (5’-GACGAGGGGAGATATGGTC-3’) (Li et al., 2012). Para la PCR preparó un volumen de 20 µL con: 2 µL Dream taq Green buffer (1X), 1 µL dNTPs, 1,5 µL/cada primer, 0,2 µL Dream taq DNA polimerasa, 2 µL ADN, 13,3 µL H2O. La reacción de PCR se desarrolló en el termociclador (TECHME®). Las condiciones térmicas: 94 ºC por 1 min; 35 ciclos a 94 ºC de 1 min; 94 ºC por 30 s; 55 °C por 1 min, extensión final de 72 °C por 10 min. Los productos amplificados se verificaron por electroforesis en gel de agarosa al 1,5% teñidos en bromuro de etidio. Los fragmentos se determinaron por comparación con el marcador de peso molecular 100 bp (Invitrogen®).
Obtención de los extractos celulares antagónicos
La rizobacterias se incubaron en 50 mL de King B líquido a 150 rpm durante 28 °C por 72 h en un agitador. Se recuperaron 15 mL en tubos falcón del inóculo bacteriano centrifugado a 6000 rpm por 5 min, este proceso se realizó por triplicado para separar el sedimento bacteriano. Los extractos celulares se filtraron empleando Nalgene Syringe de 0,2 µm y sometidas a choque térmico de 15 min a 90 °C a baño María y 15 min a – 40 °C en el ultra congelador por triplicado y almacenados a 4 °C.
Inhibición de las estructuras miceliales y de prolife-ración de FOC-R1 por metabolitos antagonistas de PGPR
Se inocularon 0,2 mL de los extractos celulares y esparcidos en 11,8 mL de medio PDA y distribuidas en cajas Petri por triplicado. Se añadió un disco (Ø 7 mm) de micelio en el centro de la placa y se adicionó una placa control sin inoculación de sobrenadantes. Se evaluó el porcentaje de inhibición de los enfrentamientos sobrenadante-patógeno desde el extremo del tarugo hasta el crecimiento fúngico a los 5, 10, 15 y 20 después de la inoculación (dpi), utilizando la formula % inhibición = (crecimiento del control – crecimiento del tratamiento / crecimiento del control) x 100.
Para la generación de esporas se esparcieron 10 mL de agua destilada estéril en placas Petri para la suspensión de esporas y filtradas con papel filtro para la separación de fragmentos de micelio. Se recuperó 1,48 mL de suspensión mezclada con 20 µL de azul de tripán. Se colocó 10 µL de la solución para su visualización en cámara Neubauer, para el conteo en cinco de los cuadrantes de 0,20 mm2, para determinar la concentración de microconidios = (número de células x 10.000) / (número de cuadros).
En todos los experimentos los tratamientos contemplan 5 réplicas con 3 unidades experimen-tales cada una. Los valores a cada condición están representados con la desviación estándar promedio individual (±), los tratamientos fueron sujetos al análisis de varianza por ANOVA, y separados por procedimiento de comparación múltiple de Tukey SD, al nivel de significancia de (p ≤ 0,05), empleando el programa estadístico Statgraphics CenturionTM V.18 (Statgraphics Technologies, 2019).
3. Resultados y discusión
Caracterización molecular del gen chiA por PCR
Se determinó la presencia del gen chiA que codifica el dominio catalítico de las quitinasas bacterianas con un producto de amplificación de 225 pb en las rizobacterias (PV-25, AC-3, BF-567, MH-18, MN 5-19, MN 5-20, CH-1, FZ 9-7, W-417, LH 5-10) (Figura 1). Donde los agentes de biocontrol de P. protegens CHA0 y P. flourescens Pf5 han recibido especial atención debido a que sintetiza metabolitos de actividad antifúngica y antibacteriana como 2,4 DAPG, Prn, Plt y HCN de amplio espectro que influyen a la actividad antagonista a problemas fitosanitarios y estimula la resistencia sistémica inducida en plantas al interaccionar con las raíces de la planta (Maurhofer et al., 2004). Al efecto inhibitorio en crecimiento micelial de Sclerotinia sclerotiorum, Rhizoctonia solani y antibiosis a Rasltonia solanacearum se debe a la producción de 2,4 DAPG, Plt y Prn por P. protegens RS-9 (Rai et al., 2017).
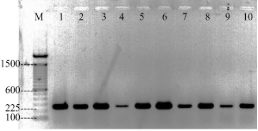
Figura 1. Amplificación del gen ChiA de 225 pb por cepas bacterianas productoras de quitinasas. M, marcador de masa molecular (Lader 100 pb Invitrogen). Carriles 1-10 (PV-25, AC-3, BF-567, MH-18, MN 5-19, MN 5-20, CH-1, FZ 9-7, W-417, LH 5-10).
Relación filogenética de las rizobacterias
La caracterización molecular al gen ARNr 16S exhibe que la cepa PV-25 mantiene una homología del 100% a Klebsiella pneumoniae NF21. La bacteria MN 5-20 identidad con Enterobacter hormaechei del 100%. Las cepas AC-3 y MN 5-19 con alta identidad con Enterobacter cloacae del 100%. Tres bacterias BF-567, MH-18 y LH 5-10 poseen alta identidad con Pseudomonas putida del 91% y 100%. Las bacterias FZ 9-7 y W-417 muestra una alta identidad con Pseudomonas chlororaphis y Pseudomonas simiae del 99% y 100%. La bacteria CH-1 posee una alta identidad con Klebsiella variicola del 100%. Los datos genotípicos presentados aquí dan como resultado sustancialmente la identificación y selección de nuevas rizobacterias PGPR (Tabla 2). El análisis filogenético por gen ARNr 16S generó dos grandes grupos. El Grupo I reúne los sub-grupo (A-B) de mayor homología cinco bacterias K. pneumoniae (PV-25), E. cloacae (AC-3), E. cloacae (MN 5-20), E. hormaechei (MN 5-20) y K. variicola (CH-1) y la formación del Grupo II reúne al sub-grupo (C-D) las bacterias P. putida (BF-567), P. putida (MH-18), P. putida (W-417), P. putida (LH 5-10) y P. chlororaphis (FZ 9-7) (Figura 3). Estas bacterias guardan una estrecha relación filogenética del gen ChiA que codifica a la producción de la enzima quitinasas al biocontrol de enfermedades fitopatógenas (Oktavioni et al., 2020). La quitinasa provoca la lisis de la pared celular de los hongos patógenos, al realizar cambios en las formas morfológicas de los hongos (esporulación, germinación de esporas y crecimiento de hifas) y la inhibición en eclosión de huevos de Meloidogyne incognita (Lee & Kim, 2015; Asril & Supriyadi, 2024).
Estas se agrupan en dos familias, donde cinco cepas bacterianas pertenecen a la familia Pseudomona-daceae al género Pseudomonas (MH-18, BF-567, W-417, LH 5-10, FZ 9-7) (Figura 2) con una secuencia de similitud del 99% a 100% a P. putida KT 2440 de capacidad de tolerar estrés salino y proceso de biorremediación (Fan et al., 2024).
Lo anterior se corrobora en el estudio de Guato-Molina et al. (2019) donde los metabolitos PR, HCN, Prn y 2,4-DAPG presentes en B. subtilis ATCC 55405, P. protegens CHA0, K. variicola BO 3-4 reduce significativamente al crecimiento micelial de F. oxysporum f. sp. Lycopersici, M. roreri y M. fijiensis e interrumpe el desarrollo de tubo germinativo de las esporas presentando anomalías en los caracteres morfológicos en presencia de extractos celular de E. asburiae PM 3-14 que contiene un amplia gama de metabolitos antagónicos (Chávez Arteaga et al., 2020; Macías Holguín et al., 2023).
4. Conclusiones
Las bacterias provenientes de suelo rizosférico muestra mayor diversidad sobre la capacidad de biosíntesis de ácido indol acético, ácido giberélico y ácido salicílico hasta las 72 h de muestreo. Estas bacterias presentaron actividad quiniolítica al gen ChiA. La secuenciación al gen ARNr 16S presenta niveles de similitud del 91% a 100% para las bacterias de K. pneumoniae (PV-25), P. chlororaphis (FZ 9-7), K. variicola (CH-1) E. hormaechei (MN 5-20), E. cloacae (AC-3; MN 5-19), P. simiae (W-417), P. putida (BF-567; MH-18; LH 5-10). El proceso de inhibición antagónico de micelio a FOC-R1 in vitro por FZ 9-7 alcanzaron los promedios superiores al 70% y disminuye la generación de esporas destacándose la actividad antifúngica.
Las musáceas se encuentran amenazadas por enfer-medades vasculares como Fusarium oxysporum f. sp. cubense raza 4 tropical (FOC R4T) y Ralstonia solanacearum raza 2 que afectan a cultivares comerciales de banano (Musa acuminata AAA) y plátano (Musa balbisiana AAB) agentes capaces de provocar pérdidas devastadoras en la producción en su totalidad. En el Ecuador aún no se ha reportado la presencia de FOC R4T, la amenaza latente exige medidas de bioseguridad y desarrollo de estrategias agrobiotecnológicas para el manejo de enfermedades. El uso de FOC-R1 como modelo experimental se presenta como una herramienta estratégica para evaluar interacciones con sobrenadantes antagónicos por constituir un insumo valioso para la formulación de bio-inoculantes con aplicaciones en condiciones de invernadero y sistemas abiertos.
Las interacciones planta-patógeno-microbioma en el Ecuador siguen siendo una incógnita sobre su funcionalidad y mecanismos de defensa molecular que genera al entrar en contacto con enfer-medades vasculares. Como línea de investigación se propone la validación de consorcios microbianos en cultivares de Musa acuminata AAA evaluando su eficacia en la supresión de FOC-R1 en plántulas de cv Gros Michel y extendiendo su aplicación hacia otros fitopatógenos de relevancia como Ralstonia solanacearum raza 2 en plántulas de cv Williams y adicionar un análisis metagenómica y transcrip-tómica para estudiar los mecanismos moleculares involucrados en la inducción de resistencia sisté-mica y la producción de metabolitos antifúngicos y antibacterianos.
ORCID
H. F. Canchignia-Martínez http://orcid.org/0000-0003-1195-5446
C. J. Macías Holguín https://orcid.org/0000-0003-2068-8503
S. G. Saucedo Aguiar https://orcid.org/0000-0002-8707-2175
H. G. Ortiz Almea https://orcid.org/0009-0005-4484-7128
L. Cansing Arichabala https://orcid.org/0009-0002-1944-9353
B. J. Lahuathe Mendoza https://orcid.org/0000-0001-7985-1999
Referencias bibliográficas
Aguilar-Hawod, K. G. I., de la Cueva, F. M., & Cumagun, C. J. R. (2019). Genetic diversity of Fusarium oxysporum f. sp. cubense causing Panama wilt of banana in the Philippines. Pathogens, 9(1), 32. https://doi.org/10.3390/pathogens9010032
Arrebola, E., Aprile, F. R., Calderón, C. E., De Vicente, A., & Cazorla, F. M. (2022). Insecticidal features displayed by the beneficial rhizobacterium Pseudomonas chlororaphis PCL1606. International Microbiology, 25(4), 679-689. https://doi.org/10.1007/s10123-022-00253-w
Asril, M., & Supriyadi, D. (2024). Characterization of Extracellular Chitinase from Bacillus cereus SAHA 12.13 and Its Potency as a Biocontrol of Curvularia affinis. Journal of Multidisciplinary Applied Natural Science, 4(1), 165-175. https://doi.org/10.47352/jmans.2774-3047.203
Auhing Arcos, J. A., Cedeño Moreira, Á. V., Saucedo Aguiar, S., Vera Benites, L. F., Macías Holguín, C. J., & Martínez, H. F. C. (2021). Biodiversidad de ecotipos y rangos de agresividad de Moniliophthora perniciosa, en Theobroma cacao L. nacional del Litoral Ecuatoriano. Scientia Agropecuaria, 12(4), 599-609. http://dx.doi.org/10.17268/sci.agropecu.2021.064
Baldy-Chudzik, K., & Stosik, M. (2005). Specific genomic fingerprints of Escherichia coli strains with repetitive sequences and PCR as an effective tool for monitoring freshwater environments. Polish Journal of Environmental Studies, 14(5).
Beale, M. H., Bearder, J. R., Down, G. H., Hutchison, M., MacMillan, J., & Phinney, B. O. (1982). The biosynthesis of kaurenolide diterpenoids by Gibberella fujikuroi. Phytochemistry, 21(6), 1279-1287. https://doi.org/10.1016/0031-9422(82)80126-X
Bharucha, U., Patel, K., & Trivedi, U. B. (2013). Optimization of indole acetic acid production by Pseudomonas putida UB1 and its effect as plant growth-promoting rhizobacteria on mustard (Brassica nigra). Agricultural research, 2, 215-221. https://doi.org/10.1007/s40003-013-0065-7.
Canchignia-Martínez, H. F., Macías-Holguin, C. J., Tapia-Quintana, D. N., Manzo-Campos, T., Saltos-Avilés, J. D., & Vera-Benites, L. F. (2025a). Bacterias Productoras de Ácido Indol-3-acético y Solubilizadoras de Fósforo y Potasio como Promotoras de Crecimiento en Oryza sativa L. Revista Terra Latinoamericana, 43. https://doi.org/10.28940/terralatinoamericana.v43i.1969
Canchignia-Martínez, H. F., Macías-Holguín, C. J., Zurita Segovia, R. D., Vera-Benites, L. F., Ortiz-Almea, H. G., & Tapia-Quintana, D. N. (2025b). Rhizobacteria and Cell Extracts with Antagonistic Potential on Phytophthora palmivora in Theobroma cacao CCN-51. Revista Terra Latinoamericana, 43. https://doi.org/10.28940/terralatinoamericana.v43i.2009
Canchignia-Martínez, H. F., Vera-Benites, L. F., Tapia-Quintana, D. N., Cedeño-Moreira, Á. V., García-Intriago, E., & Macías-Holguín, C. J. (2024c). Caracterización Bioquímica de Rizobacterias Endófitas con Actividad Biocontroladora Contra Phytophthora palmivora y Lasiodiplodia theobromae. Revista Terra Latinoamericana, 42. https://doi.org/10.28940/terra.v42i0.1807
Cedeño Moreira, Á. V., Romero Meza, R. F., Auhing Arcos, J. A., Mendoza León, A. F., Abasolo Pacheco, F., & Canchignia Martínez, H. F. (2020). Caracterización de Phytophthora spp. y aplicación de rizobacterias con potencial en biocontrol de la enfermedad de la mazorca negra en Theobroma cacao variedad CCN-51. Scientia Agropecuaria, 11(4), 503-512. https://dx.doi.org/10.17268/sci.agropecu.2020.04.05
Chávez-Arteaga, K., Guato-Molina, J., Peñafiel-Jaramillo, M., Mestanza-Uquillas, C., & Canchignia-Martínez, H. F. (2018). Bacterias fluorescentes productoras de metabolitos antagónicos de cultivares nativos de Musa sp. y su diversidad filogenética al gen ARNr 16S. Ciencia y Tecnología, 11, 17-29.
Chávez-Arteaga, K. T., Guato Molina, J. J., Rodríguez Acosta, J. L., Cedeño Moreira, Ángel V., Romero Meza, R. F., Canchignia Martínez, H. F. (2020). Rizobacterias con potencial antagonista in vitro a Mycosphaerella fijiensis Morelet. Ciencia y Tecnología, 13(2), 9–16. https://doi.org/10.18779/cyt.v13i2.387
Chen, Q., Qi, P., Xu, R., Tambong, J. T., Djama, Z. R., & Li, W. (2011). Comparison of three typing methods for evaluating the diversity of Pseudomonas fluorescens in the rhizosphere. Journal of Plant Sciences, 6(2), 52. https://dx.doi.org/10.3923/jps.2011.52.65
Cook, R. J. (1993). Making greater use of introduced microorganisms for biological control of plant pathogens. Annual review of phytopathology, 31(1), 53-80. https://doi.org/10.1146/annurev.py.31.090193.000413
Crespo Ávila, J. A., Carranza Cárdenas, C. C., Cedeño Moreira, A. V., Vera Benites, L. F., & Chevez Villanueva, M. S. (2024). Actividad antagonista de PGPR en nematodo fitoparásito Pratylenchus spp. en Musa paradisiaca (Musa acuminata× M. balbisiana) vc cavendish. Alfa Revista de Investigación en Ciencias Agronómicas y Veterinaria, 8(24), 717-728. https://doi.org/10.33996/revistaalfa.v8i24.297
Crespo-Clas, Á. M., Cedeño-Moreira, Á. V., Canchignia-Martínez, H. F., & Garcés-Fiallos, F. R. (2024). Rhizobacterial consortium differently affects black leaf spot, physiological, morpho-logical, and productive components in two generations of banana plants. Rhizosphere, 31, 100932. https://doi.org/10.1016/j.rhisph.2024.100932
da Silva, C. B., Dos Santos, H. R. M., Marbach, P. A. S., de Souza, J. T., Cruz-Magalhães, V., Argôlo-Filho, R. C., & Loguercio, L. L. (2019). First-tier detection of intragenomic 16S rRNA gene variation in culturable endophytic bacteria from cacao seeds. PeerJ, 7, e7452. https://doi.org/10.7717/peerj.7452
Dita, M. A., Waalwijk, C., Buddenhagen, I. W., Souza Jr, M. T., & Kema, G. H. J. (2010). A molecular diagnostic for tropical race 4 of the banana fusarium wilt pathogen. Plant pathology, 59(2), 348-357. https://doi.org/10.1111/j.1365-3059.2009.02221.x
Djeugap, J. F., Abireche, H. U., Donfack, C. P., Sonkoue, A. M., Ndogho, A., & Nouteka, J. N. (2023). Cultural characterization of five isolates of Fusarium oxysporum f. sp. cubense (banana fusarium wilt) and antifungal activity of plant extracts. Pakistan Journal of Phytopathology, 35(1), 43-53. https://doi.org/10.33866/phytopathol.035.01.0844
Drancourt, M., Bollet, C., & Raoult, D. (2000). 16 S ribosomal DNA sequence analysis of a large collection of enviromental and clinical unidentiable bacterial isolates. Journal of Clinical Microbiol, 38: 362-363. https://doi.org/10.1128/jcm.38.10.3623-3630.2000
El-Sapagh, S., Allam, N. G., El-Sayed, M. N. E. D., El-Hefnawy, A. A., Korbecka-Glinka, G., & Shala, A. Y. (2023). Effects of Silybum marianum L. seed extracts on Multi Drug Resistant (MDR) Bacteria. Molecules, 29(1), 64. https://doi.org/10.3390/molecules29010064
Fan, M., Tan, S., Wang, W., & Zhang, X. (2024). Improvement in Salt Tolerance Ability of Pseudomonas putida KT2440. Biology, 13(6), 404. https://doi.org/10.3390/biology13060404
Felsenstein, J. (1985). Confidence limits on phylogenies: an approach using the bootstrap. evolution, 39(4), 783-791. https://doi.org/10.1111/j.1558-5646.1985.tb00420.x
Flury, P., Vesga, P., Péchy-Tarr, M., Aellen, N., Dennert, F., et al. (2017). Antimicrobial and insecticidal: cyclic lipopeptides and hydrogen cyanide produced by plant-beneficial Pseudomonas strains CHA0, CMR12a, and PCL1391 contribute to insect killing. Frontiers in microbiology, 8, 100. https://doi.org/10.3389/fmicb.2017.00100.
Gang, S., Sharma, S., Saraf, M., Buck, M., & Schumacher, J. (2019). Analysis of indole-3-acetic acid (IAA) production in Klebsiella by LC-MS/MS and the Salkowski method. Bio-protocol, 9(9), e3230-e3230. https://doi.org/10.21769/BIOPROTOC.3230
Getahun, A., Kiros, S., Muleta, D., & Assefa, F. (2020). Genetic and metabolic diversities of rhizobacteria isolated from degraded soil of Ethiopia. Heliyon, 6(12). https://doi.org/10.1016/j.heliyon.2020.e05697
Gordon, S. A., & Weber, R. P. (1951). Colorimetric estimation of indoleacetic acid. Plant physiology, 26(1), https://doi.org/192. 10.1104/pp.26.1.192
Guato-Molina, J. J., Auhing-Arcos, J. A., Crespo-Ávila, J. A., Esmeraldas-García, G. A., Mendoza-León, A. F., & Canchignia-Martínez, H. F. (2019). Bacterias promotoras del crecimiento en plantas con potencial agente biocontrolador a Fusarium oxysporum f. sp. Lycopersici, y Moniliophthora roreri. Scientia Agropecuaria, 10(3), 393-402. http://dx.doi.org/10.17268/sci.agropecu.2019.03.10
Gupta, N., Balomajumder, C., & Agarwal, V. K. (2010). Enzymatic mechanism and biochemistry for cyanide degradation: a review. Journal of hazardous materials, 176(1-3), 1-13. https://doi.org/10.1016/j.jhazmat.2009.11.038
Gurdaswani, V., Ghag, S. B., & Ganapathi, T. R. (2020). FocSge1 in Fusarium oxysporum f. sp. cubense race 1 is essential for full virulence. BMC microbiology, 20, 1-15. https://doi.org/10.4067/S0717-34582008000500005
Gusmiaty, Restu, M., Bachtiar, B., & Larekeng, S. H. (2019). Gibberellin and IAA production by rhizobacteria from various private forest. IOP Conference Series: Earth and Environmental Science, 270(1), 012018. https://doi.org/10.1088/1755-1315/270/1/012018.
Hernández García, M., Morgante, V., Avila Perez, M., Villalobos Biaggini, P., Miralles Noé, P., González Vergara, M., & Seeger Pfeiffer, M. (2008). Novel s-triazine-degrading bacteria isolated from agricultural soils of central Chile for herbicide bioremediation. Electronic Journal of Biotechnology, 11(5), 5-6. http://dx.doi.org/10.4067/S0717-34582008000500005
Hernández, A., Rives, N., Caballero, A., Hernández, A. N., & Heydrich, M. (2004). Caracterización de rizobacterias asociadas al cultivo del maíz en la producción de metabolitos del tipo AIA, sideróforos y ácido salicílico. Revista Colombiana de biotecnología, 6(1), 6-13.
Holbrook, A. A., Edge, W. J. W., & Bailey, F. (1961). Spectrophotometric method for determination of gibberellic acid. https://doi.org/10.1021/ba-1961-0028.ch018
Izquierdo-García, L. F., Carmona, S. L., Zuluaga, P., Rodríguez, G., Dita, M., Betancourt, M., & Soto-Suárez, M. (2021). Efficacy of disinfectants against Fusarium oxysporum f. sp. cubense tropical race 4 isolated from La Guajira, Colombia. Journal of Fungi, 7(4), 297. https://doi.org/10.3390/jof7040297
Jaroszuk-Ściseł, J., Tyśkiewicz, R., Nowak, A., Ozimek, E., Majewska, M., Hanaka, A., ... & Janusz, G. (2019). Phytohormones (auxin, gibberellin) and ACC deaminase in vitro synthesized by the mycoparasitic Trichoderma DEMTkZ3A0 strain and changes in the level of auxin and plant resistance markers in wheat seedlings inoculated with this strain conidia. International Journal of Molecular Sciences, 20(19), 4923. https://doi.org/10.3390/ijms20194923
Kalimuthu, R., Suresh, P., Varatharaju, G., Balasubramanian, N., Rajasekaran, K. M., & Shanmugaiah, V. (2019). Isolation and characterization of Indole acetic acid [IAA] producing tomato Rhizobacterium pseudomonas sp VSMKU4050 and its potential for plant growth promotion. International Journal of Current Microbiology and Applied Sciences, 8(06), 443-455. https://doi.org/10.20546/ijcmas.2019.806.050
Kang, S. M., Adhikari, A., Lee, K. E., & Park, Y. G. (2019). Gibberellin producing rhizobacteria Pseudomonas koreensis MU2 enhance growth of lettuce (Lactuca sativa) and Chinese cabbage (Brassica rapa, chinensis). Journal of Microbiology, Biotechnology & Food Sciences, 9(2). https://doi.org/10.15414/jmbfs.2019.9.2.166-170
Kesaulya, H., Zakaria, B., & Syaiful, S. A. (2015). Isolation and physiological characterization of PGPR from potato plant rhizosphere in medium land of Buru Island. Procedia Food Science, 3, 190-199. https://doi.org/10.1016/j.profoo.2015.01.021
Keswani, C., Singh, S. P., García‐Estrada, C., Mezaache‐Aichour, S., Glare, T. R., Borriss, R., ... & Sansinenea, E. (2022). Biosynthesis and beneficial effects of microbial gibberellins on crops for sustainable agriculture. Journal of applied microbiology, 132(3), 1597-1615. https://doi.org/10.1111/JAM.15348
King, E. O., Ward, M. K., & Raney, D. E. (1954). Two simple media for the demonstration of pyocyanin and fluorescin. The Journal of laboratory and clinical medicine, 44(2), 301-307. https://doi.org/10.5555/URI:PII:002221435490222X
Kochar, M., Upadhyay, A., & Srivastava, S. (2011). Indole-3-acetic acid biosynthesis in the biocontrol strain Pseudomonas fluorescens Psd and plant growth regulation by hormone overexpression. Research in microbiology, 162(4), 426-435. https://doi.org/10.1016/j.resmic.2011.03.006
Lee, Y. S., & Kim, K. Y. (2015). Statistical optimization of medium components for chitinase production by Pseudomonas fluorescens strain HN1205: role of chitinase on egg hatching inhibition of root-knot nematode. Biotechnology & Biotechnological Equipment, 29(3), 470-478. https://doi.org/10.1080/13102818.2015.1010702
Lenin, G., & Jayanthi, M. (2012). Indole acetic acid, gibberellic acid and siderophore production by PGPR isolates from rhizospheric soils of Catharanthus roseus. International Journal of Pharmaceutical Biological Archive, 3(4), 933-938.
Li, M. H., Yu, X. T., Wang, H. F., Zhou, J. N., Xi, P. G., & Jiang, Z. D. (2012). Rapid detection and identification of Fusarium oxysporum f. sp. cubense race 1 and race 4. Scientia Agricultura Sinica, 45(19), 3971-3979. http://10.3864/j.issn.0578-1752.2012.19.008
López-Zapata, S. P., & Castaño-Zapata, J. (2019). Integrated management of Panama disease Fusarium oxysporum Schlechtend.: Fr. f. sp. cubense (EF SM.) WC Snyder & HN Hansen: a review. Revista UDCA Actualidad & Divulgación Científica, 22(2). https://doi.org/10.31910/rudca.v22.n2.2019.1240
Louws, F. J., Fulbright, D. W., Stephens, C. T., & De Bruijn, F. J. (1994). Specific genomic fingerprints of phytopathogenic Xanthomonas and Pseudomonas pathovars and strains generated with repetitive sequences and PCR. Applied and environmental microbiology, 60(7), 2286-2295. https://doi.org/10.1128/aem.60.7.2286-2295.1994
Macías-Holguín, C. J., Canchignia-Martínez, H. F., Delgado-Basurto, V. D., Paucar-Nieto, F. P., Arellano-Ibarra, K. V., & Cedeño-Moreira, Á. V. (2023a). Efectos de la co-inoculación de Bioformulados (PGPR´ s) sobre el porcentaje de germinación y promover el crecimiento en plántula de papaya (Carica papaya L.). Manglar, 20(2), 149-155. http://dx.doi.org/10.57188/manglar.2023.017
Macías Holguín, C. J., Valarezo Padilla, F. C., Tapia Quintana, D. N., Canchignia Martínez, H. F., Cedeño Moreira, Ángel V., & García Intriago, E. (2023b). Efecto de bioformulados bacterianos como controladores de Radopholus similis y potenciadores del desarrollo de plántulas de banano (Musa acuminata) cultivar Williams. Ciencia y Tecnología, 16(2), 9–16. https://doi.org/10.18779/cyt.v16i2.705
Magdama, F., Monserrate-Maggi, L., Serrano, L., Sosa, D., Geiser, D. M., & Jiménez-Gasco, M. D. M. (2019a). Comparative analysis uncovers the limitations of current molecular detection methods for Fusarium oxysporum f. sp. cubense race 4 strains. PLoS One, 14(9), e0222727. https://doi.org/10.1371/JOURNAL.PONE.0222727
Magdama, F., Monserrate-Maggi, L., Serrano, L., García Onofre, J., & Jiménez-Gasco, M. D. M. (2020b). Genetic Diversity of Fusarium oxysporum f. sp. cubense, the Fusarium Wilt Pathogen of Banana, in Ecuador. Plants, 9(9), 1133. https://doi.org/10.3390/plants9091133
Martín, M. C., Leyva, L., Suárez, M. A., Pichardo, T., Caraballoso, I. B., & Capó, Y. A. (2021). Antifungal activity of Bacillus amyloliquefaciens against Fusarium oxysporum f. sp. cubense race 1. Agronomía Mesoamericana, 466-478. https://doi.org/10.15517/am.v32i2.39720
Maurhofer, M., Baehler, E., Notz, R., Martinez, V., & Keel, C. (2004). Cross talk between 2, 4-diacetylphloroglucinol-producing biocontrol pseudomonads on wheat roots. Applied and Environmental Microbiology, 70(4), 1990-1998. https://doi.org/10.1128/AEM.70.4.1990-1998.2004
Meldrum, R. A., Daly, A. M., Tran-Nguyen, L. T. T., & Aitken, E. A. B. (2013). The effect of surface sterilants on spore germination of Fusarium oxysporum f. sp. cubense tropical race 4. Crop protection, 54, 194-198. https://doi.org/10.1016/J.CROPRO.2013.08.014
Meyer, J. A., & Abdallah, M. A. (1978a). The fluorescent pigment of Pseudomonas fluorescens: biosynthesis, purification and physicochemical properties. Microbiology, 107(2), 319-328. https://doi.org/10.1099/00221287-107-2-319.
Meyer, J. M., Azelvandre, P., & Georges, C. (1992b). Iron metabolism in Pseudomonas: salicylic acid, a siderophore of Pseudomonas fluorescens CHA0. BioFactors (Oxford, England), 4(1), 23-27.
Mishra, A. K., & Baek, K. H. (2021). Salicylic acid biosynthesis and metabolism: a divergent pathway for plants and bacteria. Biomolecules, 11(5), 705. https://doi.org/10.3390/biom11050705
Mostert, D., Molina, A. B., Daniells, J., Fourie, G., Hermanto, C., Chao, C. P., ... & Viljoen, A. (2017). The distribution and host range of the banana Fusarium wilt fungus, Fusarium oxysporum f. sp. cubense, in Asia. PLoS One, 12(7), e0181630. https://doi.org/10.1371/journal.pone.0181630
Naik, P. R., Sahoo, N., Goswami, D., Ayyadurai, N., & Sakthivel, N. (2008). Genetic and functional diversity among fluorescent pseudomonads isolated from the rhizosphere of banana. Microbial ecology, 56, 492-504. https://doi.org/10.1007/s00248-008-9368-9
Niwas, R., Chand, G., & Gupta, R. N. (2022). Fusarium wilt: a destructive disease of banana and their sustainable management. In Fusarium-An Overview of the Genus. IntechOpen. https://doi.org/10.5772/intechopen.101496
Odori, C., Ngaira, J., Kinyua, J., & Nyaboga, E. N. (2020). Morphological, genetic diversity and symbiotic functioning of rhizobia isolates nodulating cowpea (Vigna unguiculata L. Walp) in soils of Western Kenya and their tolerance to abiotic stress. Cogent Food & Agriculture, 6(1), 1853009. https://doi.org/10.1080/23311932.2020.1853009
Oktavioni, M., Winata, S. R., Syafriani, E., Syukriani, L., & Jamsari, J. (2020, April). Isolation of Chitinase B [ChiB] gene from Serratia plymutica strain UBCF_13. IOP Conference Series: Earth and Environmental Science, 497(1), 012020. https://doi.org/10.1088/1755-1315/497/1/012020
Parvin, W., Rahman, M. M., Govender, N. T., & Wong, M. Y. (2020). Identification, determination and quantification of indole-3-acetic acid produced by Pseudomonas aeruginosa UPMP3 and its effect on the growth of oil palm (Elaeis guineensis Jacq). World Journal of Agricultural Research, 8(3), 75-83. https://doi.org/10.12691/wjar-8-3-2
Patten, C. L., & Glick, B. R. (1996a). Bacterial biosynthesis of indole-3-acetic acid. Canadian journal of microbiology, 42(3), 207-220. https://doi.org/10.1139/m96-032
Patten, C. L., & Glick, B. R. (2002b). Role of Pseudomonas putida indoleacetic acid in development of the host plant root system. Applied and environmental microbiology, 68(8), 3795-3801. https://doi.org/10.1128/AEM.68.8.3795-3801.2002
Patten, C. L., Blakney, A. J., & Coulson, T. J. (2013c). Activity, distribution and function of indole-3-acetic acid biosynthetic pathways in bacteria. Critical reviews in microbiology, 39(4), 395-415. https://doi.org/10.3109/1040841X.2012.716819
Peng, H., Zhang, P., Bilal, M., Wang, W., Hu, H., & Zhang, X. (2018). Enhanced biosynthesis of phenazine-1-carboxamide by engineered Pseudomonas chlororaphis HT66. Microbial cell factories, 17, 1-12.
Peñafiel-Jaramillo, M., Barrera-Álvarez, A. E., Torres-Navarrete, E. D., Canchignia-Martínez, H. F., Prieto-Encalada, H., & Morante-Carriel, J. (2016). Producción de ácido indol-3-acético por Pseudomonas veronii R4 y formación de raíces en hojas de vid “Thompson seedless” in vitro. Ciencia y Tecnología, 9, 31-36. https://doi.org/10.18779/cyt.v9i1.158
Prisa, D., Fresco, R., & Spagnuolo, D. (2023). Microbial biofertilisers in plant production and resistance: A review. Agriculture, 13(9), 1666. https://doi.org/10.3390/AGRICULTURE13091666
Qin, Y., Xie, X. Q., Khan, Q., Wei, J. L., Sun, A. N., Su, Y. M., ... & Xing, Y. X. (2022). Endophytic nitrogen-fixing bacteria DX120E inoculation altered the carbon and nitrogen metabolism in sugarcane. Frontiers in Microbiology, 13, 1000033. https://doi.org/10.3389/fmicb.2022.1000033
Rai, R., Srinivasamurthy, R., Dash, P. K., & Gupta, P. (2017). Isolation, characterization and evaluation of the biocontrol potential of Pseudomonas protegens RS-9 against Ralstonia solanacearum in Tomato. Indian Jorunal of Experimental Biology, 55, 595-603.
Ramaiah, N., Hill, R. T., Chun, J., Ravel, J., Matte, M. H., Straube, W. L., & Colwell, R. R. (2000). Use of a chiA probe for detection of chitinase genes in bacteria from the Chesapeake Bay. FEMS Microbiology Ecology, 34(1), 63-71. https://doi.org/10.1111/j.1574-6941.2000.tb00755.x
Ramette, A., Moënne-Loccoz, Y., & Défago, G. (2001). Polymorphism of the polyketide synthase gene phlD in biocontrol fluorescent pseudomonads producing 2, 4-diacetylphloroglucinol and comparison of PhlD with plant polyketide synthases. Molecular plant-microbe interactions, 14(5), 639-652. https://doi.org/10.1094/MPMI.2001.14.5.639
Ranjbar, R., Tabatabaee, A., Behzadi, P., & Kheiri, R. (2017). Enterobacterial repetitive intergenic consensus polymerase chain reaction (ERIC-PCR) genotyping of Escherichia coli strains isolated from different animal stool specimens. Iranian journal of pathology, 12(1), 25. https://doi.org/10.30699/ijp.2017.21506
Rijavec, T., & Lapanje, A. (2016). Hydrogen cyanide in the rhizosphere: not suppressing plant pathogens, but rather regulating availability of phosphate. Frontiers in microbiology, 7, 216209. https://doi.org/10.3389/fmicb.2016.01785
Rouhrazi, K., & Khodakaramian, G. (2015). Phenotypic and genotypic diversity of root-nodulating bacteria isolated from chickpea (Cicer arietinum L.) in Iran. Annals of microbiology, 65, 2219-2227. https://doi.org/10.1007/s13213-015-1062-9
Sarker, A., & Al-Rashid, J. (2013). Analytical protocol for determination of Indole 3 acetic acid (IAA) production by Plant Growth Promoting Bacteria (PGPB). Technical report of Quantification of IAA by microbes September: 3–5.
Satilmis, S., Toprak, N. U., Ilgın, C., & Soyletir, G. (2019). Evaluation of direct 16S rRNA PCR from clinical samples for bacterial detection in normally sterile body sites. The Journal of Infection in Developing Countries, 13(11), 978-983. https://doi.org/10.3855/jidc.11732
Shrivastava, U. P. (2017). Molecular diversity assessment of plant growth promoting rhizobacteria using denaturing gradient gel electrophoresis (DGGE) of 16s rRNA gene. International Journal of Applied Sciences and Biotechnology, 5(1), 72-80. https://doi.org/10.3126/ijasbt.v5i1.17029
Siamak, S. B., & Zheng, S. (2018). Banana Fusarium wilt (Fusarium oxysporum f. sp. cubense) control and resistance, in the context of developing wilt-resistant bananas within sustainable production systems. Horticultural Plant Journal, 4(5), 208-218. https://doi.org/10.1016/J.HPJ.2018.08.001
Singh, B. P. (2014). Genetic fingerprinting of antimicrobial fluorescent Pseudomonads associated with banana rhizosphere. Austin Journal of Biotechnology & Bioengineering, 1(2), 1-6.
Statgraphics Technologies (2019). Statgraphics Centurion Version 18 User ́s Manual. The Plains, VA, USA: Statgraphics Inc.
Suresh, P., Varathraju, G., Shanmugaiah, V., Almaary, K. S., Elbadawi, Y. B., & Mubarak, A. (2021). Partial purification and characterization of 2, 4-diacetylphloroglucinol producing Pseudomonas fluorescens VSMKU3054 against bacterial wilt disease of tomato. Saudi Journal of Biological Sciences, 28(4), 2155-2167. https://doi.org/10.1016/j.sjbs.2021.02.073
Tamura, K., Stecher, G., Peterson, D., Filipski, A., & Kumar, S. (2013). MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0. Molecular Biology and Evolution, 30(12), 2725-29. https://doi.org/10.1093/molbev/mst197
Thangavelu, R., Gopi, M., Pushpakanth, P., Loganathan, M., Edwin Raj, E., Marimuthu, N., ... & Uma, S. (2021). First Report of Fusarium oxysporum f. sp. cubense VCG 0125 and VCG 01220 of Race 1 Infecting Cavendish Bananas (Musa sp. AAA) in India. Plant Disease, 105(4), 1215-1215. https://doi.org/10.1094/PDIS-09-20-2052-PDN
Vega-Celedón, P., Canchignia Martínez, H., González, M., & Seeger, M. (2016). Biosíntesis de ácido indol-3-acético y promoción del crecimiento de plantas por bacterias. Cultivos tropicales, 37, 33-39. https://doi.org/10.13140/RG.2.1.5158.3609
Verma, P., Chandra, P., Rai, A. K., Basak, N., Sundha, P., Sehrawat, A., ... & Yadav, R. K. (2023). Isolation, Screening and Evaluation of Biocontrol Potential of Rhizobacteria isolated from different agro-ecologies. Journal of Soil Salinity and Water Quality, 15(2), 229-241.
Versalovic, J. (1994). Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction. Methods in Molecular and Cellular Biology, 5(1), 25-40.
Visca, P., Ciervo, A., Sanfilippo, V., & Orsi, N. (1993). Iron-regulated salicylate synthesis by Pseudomonas spp. Microbiology, 139(9), 1995-2001. https://doi.org/10.1099/00221287-139-9-1995
Were, E., Viljoen, A., & Rasche, F. (2023). Iron necessity for chlamydospore germination in Fusarium oxysporum f. sp. cubense TR4. BioMetals, 36(6), 1295-1306. https://doi.org/10.1007/s10534-023-00519-4.