RESEARCH ARTICLE
Automatic counting of fish larvae using computer vision based on neural networks
Conteo automático de larvas de peces utilizando visión por computadora basado en redes neuronales
Jhordani Guélac1, * ; Jeison Sánchez1 ; Miguel Valles-Coral2 ; Nixon Nakagawa3 ;
Ariel Kedy Chichipe-Puscan3
1 Universidad Peruana Unión, ft Tarapoto, Jr. Los Mártires Nro. 340, Morales, San Martín. Peru.
2 Universidad Nacional de San Martín, Jr. Maynas N° 177, Tarapoto. Peru.
3 Instituto de Investigaciones de la Amazonia Peruana, Av. Abelardo Quiñones km 2.5, Iquitos, Loreto. Peru.
* Corresponding author: jhordani.gomez@upeu.edu.pe (J. Guélac).
Received: 24 December 2021. Accepted: 30 April 2022. Published: 29 June 2022.
Abstract
Fish larvae counting is a technique applied in aquaculture to determine the efficiency of the induction stage and to know the number of fertilized larvae. For this reason, the research aims to improve the count of larvae under 3 fundamental pillars: precision, error and time. For this, we carried out an experimental investigation under a completely randomized design with two counting systems: traditional and artificial vision; each one with 10 repetitions, with 2000 larvae; Later, we carried out the count by means of artificial vision using a camera that captured images of a fish tank with moving fish. The results show that the proposed method is feasible for counting larvae, with 92.65% accuracy, 7.41% error and an average time of 61 seconds per repetition in relation to the traditional counting system: accuracy 64.44%, error 35.61% and time 2009.3 s. The developed system can be replicated in the aquaculture sector due to its effectiveness and cost.
Keywords: aquaculture; automation; automatic counter; ornamental fish; computer vision.
Resumen
El conteo de larvas de peces es una técnica aplicada en la acuicultura para determinar la eficiencia de la etapa de inducción y conocer la cantidad de larvas fecundadas. Por esta razón, el objetivo de este trabajo fue mejorar el conteo de larvas bajo 3 pilares fundamentales: precisión, error y tiempo. Para esto realizamos una investigación experimental bajo un diseño completo al azar con dos sistemas de conteo: tradicional y de visión artificial; cada una con 10 repeticiones, con 2000 larvas; posteriormente realizamos el conteo mediante visión artificial utilizando una cámara que capturaba las imágenes de una pecera con peces en movimiento. Los resultados muestran que el método propuesto es factible para el conteo de larvas, con un 92,65% de precisión, 7,41% de error y un tiempo promedio de 61 segundos por repetición en relación al sistema de conteo tradicional: precisión 64,44%, error 35,61% y tiempo 2009,3 s. El sistema desarrollado puede ser replicado en el sector acuícola por su efectividad y costo.
Palabras clave: acuicultura; automatización; contador automático; peces ornamentales; visión por computadora.
DOI: https://dx.doi.org/10.17268/sci.agropecu.2022.014
Cite this article:
Guélac, J., Sánchez, J. E., Valles-Coral, M., Nakagawa, N., & Chichipe-Puscan, A. K. (2022). Conteo automático de larvas de peces utilizando visión por computadora basado en redes neuronales. Scientia Agropecuaria, 13(2), 159-166.
1. Introducción
El conteo de larvas de boquichico (Prochilodus nigricans) es una técnica aplicada en la acuicultura con el objetivo de determinar la eficiencia de la etapa de inducción hormonal en el proceso de reproducción (David-Ruales et al., 2018); conocer la cantidad de larvas fecundadas en cultivo garantiza las condiciones óptimas para su salud y crecimiento (Rojas & Salazar, 2018).
Gran parte del sector acuícola adoptan mecanismos tradicionales de conteo de peces (Espinoza et al., 2019), requiriendo trabajo humano intensivo que consume tiempo; lo cual, agregan incertidumbre al conteo considerándose práctica lenta (Yang et al., 2021).
El conteo automático a diferencia de la técnica tradicional es una solución para optimizar el proceso, dado a su precisión de conteo y disminución del tiempo. Para Bellemo et al. (2019), su puesta en producción no es tarea fácil, ya que deben cumplir ciertos criterios de evaluación como características del pez, iluminación del ambiente y turbidez del agua.
En la región Amazonas, el Centro de Investigaciones Seasme del Instituto de Investigaciones de la Amazonía Peruana (IIAP), concebida para lograr el desarrollo sostenible en las comunidades nativas a través de la transferencia tecnológica, aplica la técnica de conteo por volumetría para conocer la eficiencia de la etapa de inducción hormonal en el proceso de reproducción inducida (Santos et al., 2020). Para realizar esta técnica se requiere como mínimo dos personas los cuales aplican este cálculo para conocer el universo de la producción (França et al., 2019). Los datos son registrados en un cuaderno de apuntes manteniendo un histórico que a posteriori a consecuencia de la manipulación continua lleguen a deteriorarse y alteren la información (Mejia et al., 2018). En tal sentido, identificamos que la aplicación de esta técnica arroja márgenes de error superior al 30% tardando un tiempo promedio de 2 horas (Yu et al., 2022).
La tecnología inadecuada para el manejo de líquidos es otro factor que repercute en los resultados del conteo y en consecuencia a los cálculos tradicionales de la producción (Lu & Luo, 2020), implicando daños mecánicos y pérdida de larvas por factor estrés (Hernández et al., 2019).
Para Cisneros-Montemayor & Cisneros-Mata (2018), los sistemas de visión artificial son una alternativa priorizable en el sector acuícola, resaltando sus ventajas de automatización, precisión y fiabilidad. Hernández-Ontiveros et al. (2018), en su investigación realizada en la estación “Brekarand” México implementó un contador automático integrado, tomando como objeto de estudio dos especies de tamaños variantes entre 0,5 y 2,3 cm en un ambiente controlado, obteniendo precisión promedio de 96,64% en el recuento de peces.
En un estudio realizado por Sánchez-Guashpa et al. (2021), aplicó el reconocimiento de peces utilizando redes neuronales convolucionales, constituido por 4 capas convolucionales al cual se aplicaron filtros de extracción de características de entrada a la red considerando 32, 64, 128 y 256 respectivamente. El modelo se entrenó con un set de datos de 36 140 imágenes de especies categorizadas en 52 géneros correspondientes a 94 especies, logrando un 97,55% de precisión en el sistema propuesto.
El objetivo de la presente investigación fue automatizar la técnica de conteo utilizando la red neuronal MaskRCNN para la identificación y segmentación de larvas de boquichico con dimensiones de 3 a 5 ml.
2. Materiales y métodos
Localización del estudio
La investigación se desarrolló en el Centro de Investigación Seasme del Instituto de Investigaciones de la Amazonía Peruana, Amazonas, Centro Poblado Nuevo Seasme, Distrito Nieva, Provincia Condorcanqui, Región Amazonas, al noroeste del Perú. Se encuentra a una altitud promedio de 190 msnm, con temperatura media de 26 °C y una precipitación media anual de 3121 mm (Mayca-Pérez et al., 2017).
Material biológico
La selección de reproductores se realizó aplicando criterios de selección no invasiva; es decir en hembras vientre abultado y flácido, papila rojiza y dilatada; en machos la expulsión de semen y emisión de ronquidos (Colorado et al., 2017). Se consideró 20 machos y 10 hembras. Poste-riormente, se trasladaron los reproductores en baldes de PVC a tanques de tratamiento hormonal previamente acondicionados (Contreras-Sánchez et al., 2020).
Utilizamos el protocolo de inducción hormonal que establece aplicar una estrategia de dos dosis de hormona en hembras estimulante 10% y desencadenante 90%, en el caso los machos estimulantes 50% y desencadenante 50%, con intervalo de 10 a 12 horas entre dosis, para el tratamiento se usó la hormona acetato de buserelina 0,0084 aplicando 1,3 ml/kg para hembras y 0,25 – 0,5 ml/kg en machos, inyectando intraperitoneal en la cavidad corporal del boquichico (Delgado et al., 2019).
El desove de peces lo realizamos de manera semi-natural con suaves masajes, recolectado los óvulos y esperma con el cual se realizó la fecundación aplicando movimientos suaves con plumas (Díaz et al., 2021). Los óvulos fecundados fueron trasladados a incubadoras de fibra de vidrio de 200 litros, colocando de 1000 a 1500 ml en cada una, tomando en cuenta el flujo de agua de 1,3 a 1,8 litros/minuto (Moncaleano et al., 2018).
Transcurrido aproximadamente 14 +-2 horas se obtuvo el objeto de estudio, el cual se cosechó aplicando la técnica de sinfoneo, utilizando mangueras plásticas de 1 pulgada de diámetro las cuales trasportaron las larvas hasta los cosechadores que fueron colocados en el interior de un recipiente (Montes-Petro et al., 2019).
Prototipo
Adquisición de la imagen
El prototipo utilizado para la adquisición de las imágenes consiste en una pecera de vidrio ajustada sobre un soporte metálico acondicionado para una cámara web; con dimensiones: ancho 20 cm, largo 34 cm y 5,4 cm de alto. Soporte metálico: alto 33,06 cm, ancho 25,6 cm, largo 39,6 cm. La cámara debe ir ubicada en el centro del soporte metálico enfocando en su totalidad a los bordes del interior de la pecera (Figura 1).
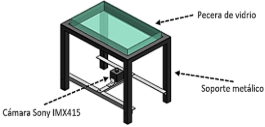
Figura 1. Estructura de prototipo.
Las imágenes fueron tomadas en un ambiente con luz controlada, con iluminación de tipo difusa de fluorescente en horarios de 9 a 11 de la mañana. Así mismo, a fin de contrastar el objeto de estudio utilizamos una cabina con fondo azul.
Se utilizó una cámara Sony IMX415 USB 2.0, con resolución de 3840(H) x 2160(V) a una distancia de 25 cm desde la base de cámara hasta la pecera. Consideramos 10 muestras controladas, de las cuales capturamos 60 imágenes para cada una, haciendo un total de 600 y 158 utilizadas para el entrenamiento.
Etiquetado de las imágenes
Este proceso consiste en la separación de cada uno de los objetos presentes en la escena de la imagen (Khokher et al., 2022), para esta etapa lo que se busca es separar las larvas del fondo, de tal modo que se obtengan únicamente las propiedades relacionadas con el objeto de estudio y no de objetos extraños.
Para este proceso se utilizó el software LabelImg, que permite marcar las larvas de la imagen y convertir sus coordenadas en archivos XML. Posteriormente, los archivos XML de la carpeta DATASET se convirtieron a un formato “coco”, para que la red neuronal pueda ser entrenada. LarvasUpdate.json contendrá la información de todas las imágenes y sus coordenadas almacenadas en un solo archivo (Anexo).
Entrenamiento de la red neuronal
Los algoritmos de entrenamiento modifican cada neurona para identificar a lo largo de las iteraciones el patrón que mejor se ajusta al objetivo (Hee-Jee et al., 2020). Para este proceso utilizamos el lenguaje de programación Python 3.9 y las librerías numpy 1.22.3, tensorflow 2.8.0 y pillow 9.1.0.
Para el entrenamiento de la red seleccionamos 600 imágenes de las cuales 158 fueron utilizadas. Se entrenó con un rango de 60 minutos mediante la red neuronal MaskRCNN. El equipo utilizado fue una laptop Windows 10 de 8GB de memoria RAM, tarjeta gráfica NVIDIA GeForce GTX 1650 y procesador Intel Core I5.
A continuación, se explica el proceso de entrenamiento de la red. La red “LARVA” utiliza 2 clases (background y larva) 64 pixeles como mínimo y 512 máximo para las imágenes, 400 épocas de entrenamiento con 5 validaciones. Las imágenes fueron divididas en 16 filas y 16 columnas y contados por secciones para luego acumularlo en un contador. Se cargó el DATASET con dos tipos de valores: 80% para el entrenamiento y 20% para la evaluación. Luego se cargó la red MaskRCNN para el entrenamiento en formato “coco”, indicando las capas que se entrenan: logística, caja, mascara y de clases. Con el objetivo de minimizar el error en el entrenamiento, se utilizó un paso (step) pequeño igual a 0,0000001 con 140 épocas. Culminado las configuraciones, se entrenó el algoritmo por un período de 60 minutos, esto a fin de llegar a un ‘loss’ bajo. Finalizado el entrenamiento, se obtuvo una carpeta llamada “logs” que contenía todos los modelos entrenados que sirvieron para reentrenar; estos archivos contenían la información necesaria para hacer predicciones en el futuro. La evaluación (Figura 2) muestra que a partir del “step” 51 empieza a subir, razón por la cual se eligió cualquier “step” menor a 51. La red se entrenó hasta el step 63 (Figura 3) y fue seleccionado el step 31.
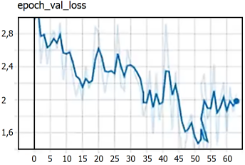
Figura 2. Evaluación step 31.
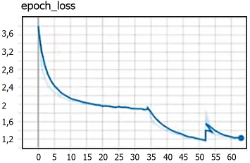
Figura 3. Entrenamiento step 63.
La Figura 4 muestra el proceso que se realizó para el entrenamiento de la red.
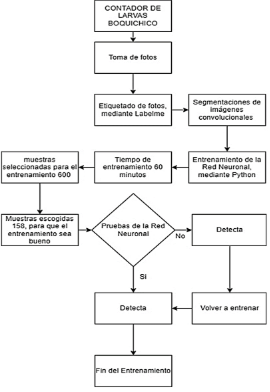
Figura 4. Procedimiento de entrenamiento de la red neuronal.
Figura 5. Diseño de conteo por tratamiento (T).
Tabla 1
Resultados del grupo control y grupo experimental
| T1 (ST) | | T2 (SVA) |
Repetición | Precisión (%) | Error (%) | Tiempo (s) | | Precisión (%) | Error (%) | Tiempo (s) |
1 | 59,4 | 40,6 | 2670 | | 91,0 | 9,0 | 59 |
2 | 56,8 | 43,3 | 2030 | | 91,3 | 8,8 | 61 |
3 | 62,3 | 37,8 | 2300 | | 90,1 | 10,0 | 63 |
4 | 67,0 | 33,0 | 2300 | | 92,4 | 7,7 | 64 |
5 | 70,5 | 29,5 | 2698 | | 94,0 | 6,1 | 63 |
6 | 62,9 | 37,1 | 1870 | | 95,2 | 4,8 | 59 |
7 | 65,7 | 34,4 | 1608 | | 94,0 | 6,1 | 58 |
8 | 58,8 | 41,3 | 1495 | | 92,1 | 7,9 | 59 |
9 | 67,2 | 32,8 | 1530 | | 94,5 | 5,6 | 62 |
10 | 73,8 | 26,3 | 1592 | | 91,9 | 8,1 | 62 |
T= Tratamiento; ST= Sistema tradicional; SVA: Sistema de visión artificial.
Diseño experimental y análisis de datos
Se utilizó un diseño completo al azar con dos sistemas de conteo de larvas: sistema tradicional - ST y sistema de visión artificial – SVA. Cada sistema tuvo 10 repeticiones, con una muestra de 2000 larvas de boquichico (Figura 5). Los datos fueron registrados en una cartilla física y digital (Excel); se armó un consolidado en una matriz general de datos y fueron procesados en el software estadístico InfoStat versión 2019. El procesamiento de los datos fue mediante el análisis T-STUDENT para dos muestras independientes con un nivel de significancia de 5%.
Conteo tradicional
Para la obtención del universo de larvas eclosionadas, utilizamos el método de conteo por volumetría, considerando 1 litros de agua como población y 0,25 ml de muestra, realizamos un total de 10 repeticiones, para posteriormente realizar la aproximación total utilizando la regla de tres simples directas (x será igual al número de larvas encontradas en 0,25 ml de agua (L), multiplicado por cantidad total de agua existente en 1 litro y dividido entre 0,25 ml de a muestra).
Conteo por visión computacional
Para este proceso, capturamos una imagen de la pecera mediante una cámara web para luego ser tratada mediante el sistema de visión artificial (x = L). L: Total de larvas encontradas en un litro de agua; x: Total de larvas.
Calculo para el error de conteo
Para el cálculo del error se consideró los siguientes datos para ambos tratamientos (x = 100 – (L*100/T). Donde L: número de larvas encontradas en 0,25 mililitros de agua; T: total de larvas de la muestra; x: porcentaje de error en el conteo.
Cálculo para el tiempo de conteo
Para calcular el tiempo en ambos tratamientos (ST – SVA), empleamos un cronometro digital a fin de conocer la duración por cada repetición.
3. Resultados y discusión
En la Tabla 1 se muestra los resultados obtenidos para el grupo control y grupo experimental.
Precisión de conteo de larvas
La prueba T- Student muestra para la variable precisión que p-valor es menor al 0,05 de significancia, cayendo en la región de rechazo; por lo que se rechaza la hipótesis nula y se acepta la hipótesis alterna; dónde los sistemas de conteo de larvas son diferentes.
En la Figura 6 se muestra las medias para la precisión del conteo con los dos sistemas; apreciando que la diferencia reportada por la prueba T, debido a que el sistema de visión artificial mostró un mayor nivel de precisión (92,65%), respecto al sistema tradicional (64,44%).
El sistema implantado es óptimo, dado que p-valor es menor a 0,05; la precisión en el conteo de peces es fundamental, influyendo en los valores morales de la organización y evitando la disconformidad de negocio (Meza & Candelaria, 2017). Por su parte Hee-Jee et al. (2020) constatan que un sistema es eficiente y tiene mayor relevancia si sus cálculos de conteo son exactos. De esto, el sistema de visión artificial mejora el proceso de conteo, permitiendo precisiones superiores al 90%.
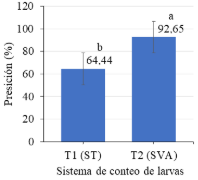
Figura 6. Comparación de medias de la precisión del conteo de larvas de boquichico.
Error de conteo
En la Figura 7 se muestran las medias para el error del conteo con los dos sistemas; apreciando que la diferencia reportada por la prueba T, debido a que el sistema de visión artificial mostró un menor nivel de error (7,41%); porcentaje de error que puede ser reducido realizando modificaciones en la pecera utilizada, puesto a que esta registra puntos muertos el cual aumenta el porcentaje de error de conteo, respecto al sistema tradicional (35,61%).
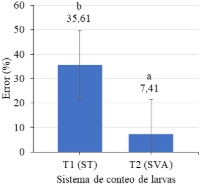
Figura 7. Comparación de medias del error del conteo de larvas de boquichico.
El sistema de visión artificial tiene márgenes de error menores al 10% cumpliendo con los parámetros de error aceptable del sector acuícola, dado a que p-valor es menor a 0,05; tal y como lo respalda el estudio de Vásquez-Salazar & Cardona-Mesa (2017); del mismo modo Puig-Pons et al. (2019) obtuvo errores menores a 10% es su estudio aplicado en España cumpliendo con los requisitos impuestos por la Comisión Internacional para la Conservación del Atún Atlántico.
Tiempo de conteo
En la Figura 8 se muestra las medias para el tiempo de conteo con los dos sistemas; apreciando que la diferencia reportada por la prueba T, es debido a que el sistema de visión artificial mostró un menor tiempo (61 s), respecto al sistema tradicional (2009,3 s).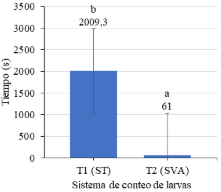
Figura 8. Comparación de medias del tiempo de conteo de larvas de boquichico.
El sistema de visión artificial reduce el tiempo de conteo (p-valor < 0,05), tal y como lo señala França et al. (2019); Vallejo et al. (2018) obtuvieron los mismos valores para p-valor posterior a la ejecución de su investigación. La Figura 8 muestra la diferencia significativa de los tratamientos aplicados, resaltando la eficiencia del sistema de visión artificial el cual registra un promedio de 61 segundos por tratamiento, Rosas-Echevarría et al. (2019) afirman que para obtener un sistema eficiente debe existir variación entre tratamientos aplicados.
4. Conclusiones
En función a los resultados de la prueba estadística T-Student, se concluye que la investigación ha logrado mejorar la técnica de conteo, bajo los 3 pilares del estudio: precisión (92,65%), disminución del error de 35,61% a 7,41% y disminución de tiempo de conteo de 2009,3 a 61 s. Este estudio evidencia que el contador automático de larvas basado en visión artificial influyó positivamente respecto al sistema tradicional, siendo una solución óptima para el conteo.
El sistema se desarrolló de manera que se pueda replicar en el sector acuícola; en función a su efectividad, debido a que los equipos son de bajo costo y el software es de código libre.
Para una posterior investigación se recomienda validar el prototipo realizando correcciones respecto a los bordes internos de la pecera y emplear mayor número de larvas.
Agradecimientos
A Dios, a nuestras familias, y al Instituto de Investigación de Amazonía Peruana, Amazonas, por el apoyo logístico brindado para la realización de la presente investigación.
ORCID
J. Guélac https://orcid.org/0000-0002-6897-3250
J. Sánchez https://orcid.org/0000-0001-8039-7682
M. Valles-Coral https://orcid.org/0000-0002-8806-2892
N. Nakagawa https://orcid.org/0000-0003-3403-026X
A. K. Chichipe-Puscan https://orcid.org/0000-0003-3298-2961
Referencias bibliográficas
Bellemo, V., Lim, Z., Lim, G., Nguyen, Q., Xie, Y., et al. (2019). Artificial intelligence using deep learning to screen for referable and vision-threatening diabetic retinopathy in Africa: a clinical validation study. The Lancet Digital Health, 1(1), 35-44.
Cisneros-Montemayor, A., & Cisneros-Mata, M. (2018). A medio siglo de manejo pesquero en el noroeste de México, el futuro de la pesca como sistema socioecológico. Relaciones Estudios de Historia y Sociedad, 39(153), 99-127.
Colorado, M., Abdala, D., Rojas, E., & Martínez, J. (2017). Repoblamiento de peces en el río Ranchería y transferencia tecnológica en el cultivo del pez nativo bocachico (Prochilodus reticulatus), a comunidades campesinas asen-tadas en la zona ribereña del río Ranchería en el depar-tamento de La Guajira , Colombia. Siembra CBA, 1, 79-91.
Contreras-Sánchez, W., Contreras-García, M., Mcdonal-Vera, U., Cruz-Rosado, L., & Martínez-García, R. (2020). Reproducción inducida del robalo chucumite (Centropomus parallelus) en Tabasco, México. AquaTIC, 32, 15-25.
David-Ruales, C., Machado-Fracalossi, D., & Vásquez-Torres, W. (2018). Desarrollo temprano en larvas de peces, clave para el inicio de la alimentación exógena. Revista Lasallista de Investigacion, 15(1), 180–194.
Delgado, M. Á., Cuesta, C. A., & Díaz, A. F. (2019). Evaluación de dos protocolos hormonales para la inducción del celo e inseminación artificial a término fijo (IATF) a vacas en el postparto temprano y en anestro, como herramienta para aumentar la productividad. LOGINN Investigación Científica y Tecnológica, 3(1), 94-104.
Díaz, Á., Cerrud, G., Cerrud, G., Junqueira, G., & Solis, L. (2021). Índices morfométricos y reproducción inducida de Cyphocharax magdalenae (Steindachner, 1878) sardina Maná. Visión Antataura, 5(1), 1-14.
Espinoza, L., Chilli, V., Pepe, R., Pino, J., & Contreras, Z. (2019). Captura, acondicionamiento y primer desove de sargo Anisotremus scapularis en la Región Tacna. Ciencia & Desarrollo, 25, 68-74.
França, P., Garcia, V., da Silva, A., Lewandowski, T., Detweiler, C., et al. (2019). Automatic live fingerlings counting using computer vision. Computers and Electronics in Agriculture, 167(September), 1-9.
Hee-Jee, S., Myeong Kwan, P., & Weon, J. (2020). Automatic Grader for Flatfishes Using Machine Vision. International Journal of Control, Automation and Systems, 18(12), 1-9.
Hernández-Ontiveros, J. M., Inzunza-González, E., García-Guerrero, E. E., López-Bonilla, O. R., Infante-Prieto, S. O., et al. (2018). Development and implementation of a fish counter by using an embedded system. Computers and Electronics in Agriculture, 145(December 2016), 53–62.
Hernández, L., Londoño, J., Hernández, K., & Torres, L. (2019). Los sistemas biofloc: una estrategia eficiente en la producción acuícola. CES Medicina Veterinaria y Zootecnia, 14(1), 70-99.
Khokher, M. R., Little, L. R., Tuck, G. N., Smith, D. V., Qiao, M., et al. (2022). Early lessons in deploying cameras and artificial intelligence technology for fisheries catch monitoring: where machine learning meets commercial fishing. Canadian Journal of Fisheries and Aquatic Sciences, 79(2), 257–266.
Lu, G., & Luo, M. (2020). Genomes of major fishes in world fisheries and aquaculture: Status, application and perspective. Aquaculture and Fisheries, 5(4), 163-173.
Mayca-Pérez, J., Medina-Ibañez, A., Velásquez-Hurtado, J. E., & Llanos-Zavalaga, L. F. (2017). Representaciones sociales relacionadas a la anemia en niños menores de tres años en comunidades Awajún y Wampis, Perú. Rev Peru Med Exp Salud Publica, 34(3), 414-436.
Mejia, B., Salas, A., & Kemper, G. (2018). An Automatic System Oriented to Counting and Measuring the Geometric Dimensions of Gray Tilapia Fingerlings Based on Digital Image Processing. Proceedings of the 2018 IEEE 25th International Conference on Electronics, Electrical Engineering and Computing, INTERCON 2018, 1–4.
Meza, B., & Candelaria, M. (2017). Innovación en el sector acuícola. Ra Ximhai, 13(3), 351–364.
Moncaleano, E., Sánchez, C., & Prieto, C. (2018). Estudio histológico y morfológico del desarrollo embrionario del pez capitán de la sabana (Eremophilus mutisii). Revista U.D.C.A Actualidad & Divulgación Científica, 21(2), 479-490.
Montes-Petro, C., Atencio-García, V., Estrada-Posada, A., & Yepes-Blandón, J. (2019). Reproducción en cautiverio de vizcaína Curimata mivartii con extracto pituitario de carpa. Orinoquia, 23(2), 63–70.
Sánchez-Guashpa, A., Pico-Valencia, P., Jiménez, P., & Holgado-Terriza, J. A. (2021). Sistema de Clasificación Automático de Peces Endémicos del Ecuador Usando Redes Neuronales Convolucionales. Revista Iberica de Sistemas e Tecnologias de Informacao, 45, 444-457.
Puig-Pons, V., Muñoz-Benavent, P., Espinosa, V., Andreu-García, G., Valiente-González, J. M., et al. (2019). Automatic Bluefin Tuna (Thunnus thynnus) biomass estimation during transfers using acoustic and computer vision techniques. Aquacultural Engineering, 85, 22–31.
Rojas, I., & Salazar, V. (2018). La acuicultura frente a los impactos de la actividad agrícola en la calidad de los servicios ambientales de la cuenca del río mayo. Una propuesta para su abordaje desde la economía ecológica. Revista de Alimentación Contemporánea y Desarrollo Regional, 28(51), 2-26.
Rosas-Echevarría, C., Solís-Bonifacio, H., & Cerna-Cueva, A. (2019). Sistema eficiente y de bajo costo para la selección de granos de café: una aplicación de la visión artificial. Scientia Agropecuaria, 10(3), 347-351.
Santos, D., Dallos, L., & Gaona-García, P. (2020). Algoritmos de rastreo de movimiento utilizando técnicas de inteligencia artificial y machine learning. Información Tecnológica, 31(3), 23-38.
Vallejo, H., Paucar, J., & Martinez, O. (2018). Visión artificial mediante el coeficiente de correlación para exámenes de retinoscopía. Maskay, 8(2), 75.
Vásquez-Salazar, R. D., & Cardona-Mesa, A. A. (2017). Diseño y construcción de un equipo portátil para conteo de alevines de tilapia roja. Revista Politécnica, 13, 101-111.
Yang, L., Liu, Y., Yu, H., Fang, X., Song, L., Li, D., & Chen, Y. (2021). Computer Vision Models in Intelligent Aquaculture with Emphasis on Fish Detection and Behavior Analysis: A Review. Archives of Computational Methods in Engineering, 28(4), 2785–2816.
Yu, X., Wang, Y., An, D., & Wei, Y. (2022). Counting method for cultured fishes based on multi-modules and attention mechanism. Aquacultural Engineering, 96, 102215.
Anexo
Código en Phyton de conversión de archivos XML (celeste), configuración (verde) y entrenamiento (azul) de la red neuronal
import labelme2coco
labelme_folder = "C:/Users/prel1/Desktop/jsones/"
save_json_path = "result/LarvasUpdate.json"
labelme2coco.convert(labelme_folder, save_json_path)
class CustomConfig(Config):
NAME = "larvas"
GPU_COUNT = 1
IMAGES_PER_GPU = 1
NUM_CLASSES = 1 + 1
IMAGE_MIN_DIM = 64
IMAGE_MAX_DIM = 512
STEPS_PER_EPOCH = 400
VALIDATION_STEPS = 5
BACKBONE = 'resnet50'
RPN_ANCHOR_SCALES = (8, 16, 32, 64, 128)
TRAIN_ROIS_PER_IMAGE = 32
MAX_GT_INSTANCES = 50
POST_NMS_ROIS_INFERENCE = 500
POST_NMS_ROIS_TRAINING = 1000
config = CustomConfig()
config.display()
import os
import sys
import json
import numpy as np
import time
from PIL import Image, ImageDraw
import skimage.draw
import random
import tensorflow as tf
ROOT_DIR = './'
from mrcnn import visualize
from mrcnn.config import Config
from mrcnn import model as modellib, utils
MODEL_DIR = os.path.join(ROOT_DIR, "logs/")
COCO_MODEL_PATH = os.path.join(ROOT_DIR, "logs/larvas20211126T1430/mask_rcnn_larvas_0052.h5")
if not os.path.exists(COCO_MODEL_PATH):
utils.download_trained_weights(COCO_MODEL_PATH)
class CustomConfig(Config):
NAME = "larvas"
GPU_COUNT = 1
IMAGES_PER_GPU = 1
NUM_CLASSES = 1 + 1
IMAGE_MIN_DIM = 64
IMAGE_MAX_DIM = 512
STEPS_PER_EPOCH = 400
VALIDATION_STEPS = 5
BACKBONE = 'resnet50'
RPN_ANCHOR_SCALES = (8, 16, 32, 64, 128)
TRAIN_ROIS_PER_IMAGE = 32
MAX_GT_INSTANCES = 50
POST_NMS_ROIS_INFERENCE = 500
POST_NMS_ROIS_TRAINING = 1000
config = CustomConfig()
config.display()
class CustomDataset(utils.Dataset):
def load_custom(self, annotation_json, images_dir, dataset_type="train"):
print("Annotation json path: ", annotation_json)
json_file = open(annotation_json)
coco_json = json.load(json_file)
json_file.close()
source_name = "coco_like"
for category in coco_json['categories']:
class_id = category['id']
class_name = category['name']
if class_id < 1:
print('Error: Class id for "{}" cannot be less than one. (0 is reserved for the background)'.format(
class_name))
return
self.add_class(source_name, class_id, class_name)
annotations = {}
for annotation in coco_json['annotations']:
image_id = annotation['image_id']
if image_id not in annotations:
annotations[image_id] = []
annotations[image_id].append(annotation)
seen_images = {}
len_images = len(coco_json['images'])
if dataset_type == "train":
img_range = [int(len_images / 5), len_images]
else:
img_range = [0, int(len_images / 5)]
for i in range(img_range[0], img_range[1]):
image = coco_json['images'][i]
image_id = image['id']
if image_id in seen_images:
print("Warning: Skipping duplicate image id: {}".format(image))
else:
seen_images[image_id] = image
try:
image_file_name = image['file_name']
image_width = image['width']
image_height = image['height']
except KeyError as key:
print("Warning: Skipping image (id: {}) with missing key: {}".format(image_id, key))
image_path = os.path.abspath(os.path.join(images_dir, image_file_name))
image_annotations = annotations[image_id]
self.add_image(
source=source_name,
image_id=image_id,
path=image_path,
width=image_width,
height=image_height,
annotations=image_annotations)
def load_mask(self, image_id):
image_info = self.image_info[image_id]
annotations = image_info['annotations']
instance_masks = []
class_ids = []
for annotation in annotations:
class_id = annotation['category_id']
mask = Image.new('1', (image_info['width'], image_info['height']))
mask_draw = ImageDraw.ImageDraw(mask, '1')
for segmentation in annotation['segmentation']:
mask_draw.polygon(segmentation, fill=1)
bool_array = np.array(mask) > 0
instance_masks.append(bool_array)
class_ids.append(class_id)
mask = np.dstack(instance_masks)
class_ids = np.array(class_ids, dtype=np.int32)
return mask, class_ids
def count_classes(self):
class_ids = set()
for image_id in self.image_ids:
image_info = self.image_info[image_id]
annotations = image_info['annotations']
for annotation in annotations:
class_id = annotation['category_id']
class_ids.add(class_id)
class_number = len(class_ids)
return class_number
dataset_train = CustomDataset()
dataset_train.load_custom("result/LarvasUpdate.json", "C:/Usersprel1/Desktop",'train')
dataset_train.prepare()
dataset_val = CustomDataset()
dataset_val.load_custom("result/LarvasUpdate.json","C:/Usersprel1/Desktop",'val')
dataset_val.prepare()
print(dataset_val.class_names)
model = modellib.MaskRCNN(mode="training", config=config, model_dir=MODEL_DIR)
init_with = "coco" # imagenet, coco, or last
if init_with == "imagenet":
model.load_weights(model.get_imagenet_weights(), by_name=True)
elif init_with == "coco":
model.load_weights(COCO_MODEL_PATH, by_name=True,
exclude=["mrcnn_class_logits", "mrcnn_bbox_fc",
"mrcnn_bbox", "mrcnn_mask"])
elif init_with == "last":
# Load the last model you trained and continue training
model.load_weights(model.find_last(), by_name=True)
dataset = dataset_train
image_ids = np.random.choice(dataset.image_ids, 4)
for image_id in image_ids:
print(image_id)
image = dataset.load_image(image_id)
mask, class_ids = dataset.load_mask(image_id)
print(dataset.image_reference(image_id))
visualize.display_top_masks(image, mask, class_ids, dataset.class_names)
from mrcnn.model import log
# Load random image and mask.
image_id = random.choice(dataset.image_ids)
image = dataset.load_image(image_id)
mask, class_ids = dataset.load_mask(image_id)
# Compute Bounding box
bbox = utils.extract_bboxes(mask)
print("image_id ", image_id, dataset.image_reference(image_id))
log("image", image)
log("mask", mask)
log("class_ids", class_ids)
log("bbox", bbox)
visualize.display_instances(image, bbox, mask, class_ids, dataset.class_names)
model.train(dataset_train, dataset_val,
learning_rate=0.00001,
epochs=80,
layers='heads')
class InferenceConfig(CustomConfig):
GPU_COUNT = 1
IMAGES_PER_GPU = 1
IMAGE_MIN_DIM = 64
IMAGE_MAX_DIM = 512
DETECTION_MIN_CONFIDENCE = 0.7
inference_config = InferenceConfig()
model = modellib.MaskRCNN(mode="inference", config=inference_config, model_dir=MODEL_DIR)
model_path = model.find_last()
assert model_path != "", "Provide path to trained weights"
print("Loading weights from ", model_path)
model.load_weights(model_path, by_name=True)
import skimage
import matplotlib.pyplot as plt
real_test_dir = 'test/'
image_paths = []
for filename in os.listdir(real_test_dir):
if os.path.splitext(filename)[1].lower() in ['.png', '.jpg', '.jpeg']:
image_paths.append(os.path.join(real_test_dir, filename))
for image_path in image_paths:
img = skimage.io.imread(image_path)
if img.ndim != 3:
image = skimage.color.gray2rgb(img)
if img.shape[-1] == 4:
image = img[..., :3]
print(image_path)
img_arr = np.array(image)
results = model.detect([img_arr], verbose=1)
r = results[0]
visualize.display_instances(img, r['rois'], r['masks'], r['class_ids'],dataset_val.class_names, r['scores'], figsize=(5,5))
model = modellib.MaskRCNN(mode="training", config=config,
model_dir=MODEL_DIR)
init_with = "coco" # imagenet, coco, or last
if init_with == "imagenet":
model.load_weights(model.get_imagenet_weights(), by_name=True)
elif init_with == "coco":
model.load_weights(COCO_MODEL_PATH, by_name=True,
exclude=["mrcnn_class_logits", "mrcnn_bbox_fc",
"mrcnn_bbox", "mrcnn_mask"])
elif init_with == "last":
# Load the last model you trained and continue training
model.load_weights(model.find_last(), by_name=True)
dataset = dataset_train
image_ids = np.random.choice(dataset.image_ids, 4)