COMPARACIÓN DE TRES PROTOCOLOS DE EXTRACCIÓN DE ADN EN Myliobatis peruvianus
COMPARISON OF THREE PROTOCOLS OF DNA EXTRACTION IN Myliobatis peruvianus
Carlos Alfonso Lino Amaya 1 *, Carlos Helí Quijano Jara 1, Cinthya Santa Cruz-López 2
1 Facultad de Ciencias Biológicas, Universidad Nacional de Trujillo, Av. Juan Pablo II s/n – Ciudad Universitaria, Trujillo, Perú.
2 Facultad de Ciencias de la Salud, Universidad Nacional de Jaén, Carretera Jaén - San Ignacio KM 24 - Sector Yanuyacu - Jaén
Carlos Alfonso Lino Amaya:
Carlos Helí Quijano Jara:
Cinthya Santa Cruz-López:
Artículo original
Recibido: 12 de octubre 2022
Aceptado: 29 de diciembre 2022
Resumen
Myliobatis peruvianus “raya” es una especie catalogada en peligro (EP) a nivel regional en base a la drástica disminución en los embarques de la especie en un 90%. Existe además falta de información de la especie en diferentes aspectos como la profundidad máxima que frecuenta; longevidad, tasa de incremento poblacional, careciendo también de información genética mediante el uso de marcadores moleculares de ADN. El objetivo de la presente investigación fue comparar tres protocolos de extracción de ADN para su posterior uso en estudios genéticos mediante el uso de marcadores moleculares de ADN. Se utilizaron tres protocolos de extracción los cuales fueron Fenol-Cloroformo, Sal común y Mezzomo, se midió la concentración y calidad de ADN con NanoDrop One C, los datos se sometieron a un análisis de varianza y a una prueba de comparaciones múltiples de Tukey, se obtuvo ADN de buena calidad en los protocolos de Sal común y Fenol-cloroformo; mientras que respecto a la concentración de ADN el protocolo que dio mejor resultado fue el de Sal común.
Palabras clave: Myliobatis peruvianus, calidad, concentración
Abstract
Myliobatis peruvianus “raya” is a regionally endangered (EP) species based on the drastic decrease in shipments of the species by 90%. There is also a lack of information on the species in different aspects such as the maximum depth it frequents; longevity, rate of population increase, also lacking genetic information through the use of DNA molecular markers. The objective of the present investigation was to compare three DNA extraction protocols for their subsequent use in genetic studies through the use of DNA molecular markers. Three extraction protocols were used, which were Phenol-Chloroform, Common Salt and Mezzomo, the concentration and quality of DNA were measured with NanoDrop One C, the data were subjected to an analysis of variance and a Tukey multiple comparison test, obtained good quality DNA in the common salt and phenol-chloroform protocols; while regarding the DNA concentration, the protocol that gave the best result was that of common salt.
Keywords: Myliobatis peruvianus, quality, concentration
* Autor para correspondencia: clino@unitru.edu.pe
Citar como:
Lino, C., Quijano, C., & Santa Cruz-López, C. 2022. Comparación de tres protocolos de extracción de ADN en Myliobatis peruvianus. REBIOL, 42(2): 194-198.
1. Introducción
La identificación de peces ha evolucionado en el tiempo, ha pasado de utilizar sus caracteres morfológicos externos proporciones, número de espinas y radios de las aletas, número de branquiespinas, número de escamas y color (Chirichigno, 1998); internos como es la morfología de los otolitos (García, 2012), mediante el uso de las claves taxonómicas, con mayor frecuencia marcadores moleculares para lo cual es fundamental realizar una extracción de ácidos desoxirribonucleicos de buena calidad y concentración (Astorga, 2008).
El tipo de tejido y la utilización de protocolos viables de extracción del ADN son puntos críticos, así se han realizado investigaciones donde se ha hecho extracción de ADN a partir de aletas (Nam et al., 2003; Wasko et al., 2003, Lopera-Barrero et al., 2006), sangre (Cummings y Thorgaard, 1994), escamas (Adcodk et al., 2000; Sire et al., 2000; Yue & Orban, 2001; Wasko et al., 2003), células bucales (Livia et al., 2006), óvulos (Aranishi, 2006) y músculo (Weber et al., 2003; Chakraborty et al., 2006), donde la utilización de Chelex 100 (Sigma, EUA), silica, micro-wave, nitrógeno líquido, fenol-cloroformo y sal común han sido bastante utilizados.
Existen diferentes protocolos de extracción de ADN, cuyo propósito es obtener una óptima cantidad (concentración) y calidad (pureza) de ADN. Los protocolos tradicionales de extracción del ADN consisten en cinco etapas principales: Homogeneización del tejido, lisis celular, separación de proteínas y lípidos, precipitación y redisolución del ADN (Dundass, 2008). El protocolo de extracción con fenol-cloroformo es el método más usado para obtener ADN genómico. Estos protocolos tienen buenos resultados para muestras de diversos orígenes. Sin embargo, es un método lento, laborioso y contaminante (Yue & Orban, 2001).
La extracción de ADN usando el protocolo con sal común (NaCl) es una alternativa simple, fácil, rápida y no contaminante que permite obtener ADN de buena calidad, en cantidades suficientes, desde muestras de tejido de peces; mientras que el protocolo salino es de manera fácil, rápida y no contaminante, que permite obtener ADN de buena calidad (Lopera-Barrero et al., 2008).
Myliobatis peruvianus es una especie que pertenece a la familia Myliobatidae, clase Chondrichthyes (Lamilla y Saenz, 2003) cuya distribución va desde Paita (Perú) hasta Curiñanco (Valdivia) en Chile (Lamilla, 2004). También se ha reportado su presencia en Ecuador (Coello & Herrera, 2010). Esta especie ha sido categorizada En peligro (EP) a nivel regional en base a la drástica disminución en los embarques de la especie en un 90% como nos indica Monsalve, (2016). Existe además falta de información de la especie en diferentes aspectos como la profundidad máxima que frecuenta; longevidad, tasa de incremento poblacional, careciendo también de información genética mediante el uso de marcadores moleculares de ADN (Fricke et al., 2020).
Por lo cual el objetivo de la presente investigación fue comparar tres protocolos de extracción de ADN para su posterior uso de estudios genéticos mediante el uso de marcadores moleculares de ADN.
2. Materiales y Métodos
Material biológico
Se colectaron muestras de tejido muscular de 25 individuos de Myliobatis peruvianus procedentes del Mercado Mayorista Pesquero Comphill S.A. - Víctor Larco, las cuales fueron colocadas en una caja térmica con hielo para su transporte y posterior almacenamiento a -80°C.
Protocolos de extracción de ADN
Para la extracción de ADN se utilizó tejido muscular (20-30 mg) de cada uno de los 25 individuos de M. peruvianus, utilizando el protocolo de extracción de Fenol-cloroformo (Bardakci y Skibinski, 1994), sal común (NaCl) (Aljanabi y Martinez, 1997) y protocolo de Mezzomo (Mezzomo et al; 2021).
Protocolo de extracción de Sal común
Las muestras de tejido muscular se colocaron en microtubos con 600 μL de tampón de lisis (2000 μL de Tris- HCL, 4000 μL de EDTA, 800 μL de NaCl) más 2000 μL de SDS y 14 μL de proteinasa K. Después se llevó al termostato durante 10 minutos a 55°C el cual aceleró el proceso de disolución de la muestra. Una vez que transcurrió el tiempo se le añadió 600 μL de NaCl a cada una de las 25 muestras y después se llevó a centrifugar por 10 minutos a 12000 rpm. El sobrenadante producto de la centrifugación se transfirió a microtubos nuevos, donde se precipitó con 700 μL de isopropanol frío, y se incubó a -20°C hasta el día siguiente. Las muestras de ADN se centrifugaron a 12000 rpm y se descartó el isopropanol cuidando que el pellet se desprenda, se lavó el pellet con 700 μL de alcohol al 70 %, luego se centrifugó a 12 000 rpm, se descartó el alcohol y luego se colocaron los tubos en un termostato a 45 °C por 15 minutos una vez seco el pellet resuspendió con 50 μL de tampón TE. El ADN obtenido se conservó a -20° C hasta su posterior utilización. (Aljanabi y Martinez, 1997).
Protocolo de extracción de Fenol – Cloroformo
Las muestras de tejido muscular se colocaron en micro-tubos con 600 μL de tampón de lisis (2000 μL de Tris- HCL, 4000 μL de EDTA, 800 μL de NaCl) más 2000 μL de SDS y 14 μL de proteinasa K. Después se llevó al termostato durante 10 minutos a 55°C. Después el ADN se purificó dos veces con Fenol- Cloroformo isomílico (15 μL) a las 25 muestras, después de cada lavado se incubó en una centrífuga por 10 minutos a 12000 rpm. El sobrenadante producto de la centrifugación se transfirió a microtubos nuevos, donde se precipitó con 700 μL de isopropanol frío, y se incubó a -20°C hasta el día siguiente. Las muestras de ADN se centrifugaron a 12000 rpm y se descartó el isopropanol cuidando que el pellet se desprenda, se lavó el pellet con 700 μL de alcohol al 70 %, luego se centrifugó a 12 000 rpm, se descartó el alcohol y luego se colocaron los tubos en un termostato a 45 °C por 15 minutos una vez seco el pellet resuspendió con 50 μL de tampón TE. El ADN obtenido se conservó a -20° C hasta su posterior utilización. (Bardakci y Skibinski, 1994).
Protocolo de Mezzomo
Las muestras de tejido muscular se colocaron en micro-tubos con 550 μL de tampón de lisis (37.5 μL de Tris- HCL, 3 μLde EDTA, 7.5 μL de NaCl) más 75 μL de SDS y 14 μL de proteinasa K. Después se llevó al termostato durante 10 minutos a 55°C. Una vez que transcurrió el tiempo se le añadió 50 μL de Cloroformo Alcohol Isomílico (CAI) a cada una de las 25 muestras y después se llevó a centrifugar por 10 minutos a 12000 rpm. Una vez terminado esto se transfirió el sobrenadante a microtubo nuevo y se añadió 50 μL de Cloroformo Alcohol Isoamílico (CAI) a cada una de las 25 muestras y repetir el procedimiento una vez más donde se incubó a – 20°C hasta el día siguiente. Las muestras de ADN se centrifugaron a 12000 rpm y se descartó el isopropanol cuidando que el pellet se desprenda, se lavó el pellet con 700 μL de alcohol al 70 %, luego se centrifugó a 12 000 rpm, se descartó el alcohol y luego se colocaron los tubos en un termostato a 45 °C por 15 minutos una vez seco el pellet resuspendió con 50 μL de tampón TE. El ADN obtenido se conservó a -20° C hasta su posterior utilización. (Mezzomo et al; 2021).
Comparación de protocolo de extracción de ADN
Para comparar los resultados de los 3 protocolos de extracción de ADN se midió la calidad y concentración del ADN (Scott et al. 2004), utilizando un NanoDrop One C, los valores fueron procesados en el programa estadístico Infostatat y Rstudio mediante la aplicación del Análisis de Varianza y prueba de comparaciones múltiples de Tukey con un 5% de significancia, para la concentración y calidad de ADN.
3. Resultados
Tabla 1. Valores promedio y desviación estándar de concentración y calidad del ADN en muestras de Myliobatis peruvianus “raya” del terminal del pesquero Comphill S.A.
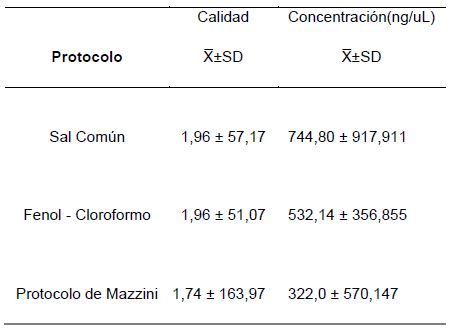
Figura 1. Promedio de calidad del ADN en muestras de Myliobatis peruvianus “raya” del Mercado Mayorista Pesquero Comphill S.A - Víctor Larco (F: Fenol - cloroformo, N: Mazzini y S: Sal común).
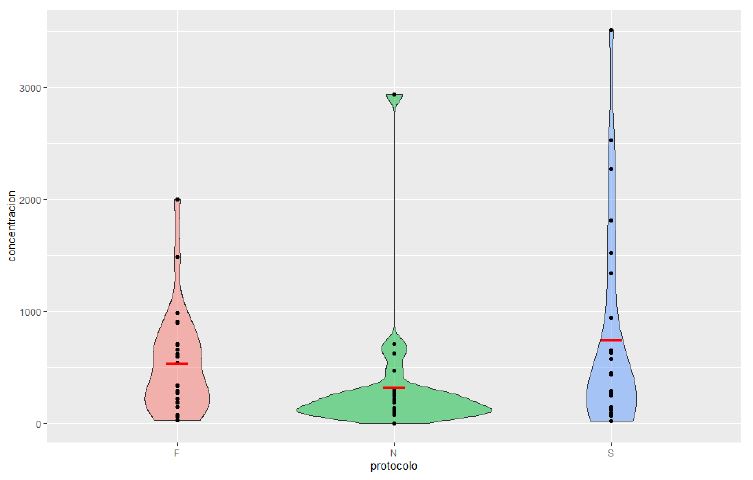
Figura 2. Promedio de concentración del ADN en muestras de Myliobatis peruvianus “raya” del Mercado Mayorista Pesquero Comphill S.A - Víctor Larco (F: Fenol - cloroformo, N: Mazzini y S: Sal común).
4. Discusión
Una etapa previa muy importante en los estudios genéticos utilizando los ácidos nucleicos es como separar y purificar estas moléculas, con el fin de poder emplearlo para otras técnicas de estudio (Checa, 2017). Actualmente se cuenta con diversos métodos de extracción los cuales la mayoría se basan en la extracción de ácidos nucleicos, entre los cuales tenemos aquellos que se basan en el uso de solventes orgánicos, Kits comerciales entre otros, presentando inconvenientes tanto en el costo, tiempo y laboriosidad (De Armas et al., 2011); sin embargo, existen protocolos sencillos, rápidos, reproducibles, económicos y sobre todo que se pueden utilizar en grandes cantidades de muestras (Ferrer et al., 2008) tales como en Sal Común y Protocolo de Mezzomo que permiten obtener resultados confiables tanto en calidad y concentración, los cuales pueden ser verificados mediante espectrofotometría, la cual nos permite determinar la pureza y concentración de muestra de ADN tomando en cuenta su absorbancia de un compuesto presente en una solución a una longitud de onda determinada, estando dentro de un rango de 1,8 a 2,0, para que puedan ser consideradas unas muestras en estado óptimo (Sambrook et al., 1989).
Como se observa en los resultados de la Tabla 1 y Figura 1, de extracción de ADN en las muestras de M. peruvianus “raya” recolectadas del terminal pesquero Comphill S.A. del Distrito de Buenos Aires, donde se compararon los 3 protocolos (Fenol Cloroformo, Sal común y Protocolo de Mezzomo), se obtuvieron los mejores valores en la calidad de ADN con los protocolos de Fenol Cloroformo y Sal común, con un promedio de 1,9; esto concuerda con lo indicado por Nelson et al. (2007) quienes mencionaban que para que una muestra de ADN tenga buena calidad tiene que estar dentro del rango de 1,8 a 2,0; respecto al protocolo de Mezzomo, se observó un promedio inferior al rango establecido con un 1,74, debido a la probable presencia de algunos contaminantes tales como proteínas.
En relación a los resultados de la Tabla 1 y Figura 2, la concentración del ADN podemos definir que todos los protocolos empleados brindan una buena concentración de ADN; sin embargo, el protocolo que brinda mejores resultados es de Sal Común con una media de 744,80 μg/ml. El motivo principal por el cual el protocolo de Sal Común presenta un alto valor de concentración es debido a que no requiere diversos lavados con solventes orgánicos tales como Fenol y Cloroformo o Alcohol Isoamílico, solventes que permiten una buena purificación del ADN, sin embargo, tiene la desventaja de provocar pérdida de ADN en cada proceso de purificación.
En la presente investigación, a diferencia de otras investigaciones, no se empleó ARNasa (Wasko et al., 2003; Lopera-Barrero et al., 2006) para obtener muestras libres o con baja concentración de ARN y por ende una calidad óptima de ADN; sin embargo, en base a los resultados obtenidos se podría concluir que no es imperativo su uso, además se buscaba estandarizar un protocolo de extracción sencillo y menos costoso considerando dentro de lo primordial en el Protocolo de Fenol-Cloroformo los reiterativos lavados que se le hacen a las muestras con estos solventes orgánicos (Wasko et al., 2003); mientras que en el Protocolo de Sal Común se prevaleció la temperatura en la cual se colocaron en el termostato (45°C) (Taberlet et al., 1996).
5. Conclusiones
Se logró obtener ADN de buena calidad en los protocolos de Sal común y Fenol-cloroformo; mientras que para el parámetro de concentración el protocolo que dio buenos valores es el de Sal común.
6. Agradecimiento
A la Dra. Zulita Prieto Lara, por brindar las facilidades del uso de las instalaciones del laboratorio de Genética de Poblaciones de la Facultad de Ciencias Biológicas de la Universidad Nacional de Trujillo.
7. Contribución de autores
C. A. Lino Amaya: Concepción, diseño, ensayos de laboratorio, elaboración y aprobación final de informe.
C. H. Quijano Jara: Concepción, diseño, ensayos de laboratorio, elaboración y aprobación final de informe
C. S. Cruz-López: Procesamiento de datos, elaboración y aprobación final de informe.
8. Conflicto de interés
Los autores declaran que no existe conflicto de interés
9. Referencias Bibliográficas
Adcodk, G.J., Bernal Ramázrez, J.H., Hauser, L., Smith, P. and Carvalho, G.R. (2000), Screening of DNA polymorphisms in samples of archived scales from New Zealand snapper. Journal of Fish Biology, 56, 1283-1287.
Aranishi, F. 2006. Single fi sh egg DNA extraction for PCR amplifi cation. Conservation Genetics 7,153-156.
Astorga, M. P. (2008). Estado actual del uso de marcadores moleculares en moluscos bivalvos de importancia para la acuicultura. En A. Lovatelli, A. Farías & I. Uriarte (Eds.). Estado actual del cultivo y manejo de moluscos bivalvos y su proyección futura Factores que afectan su sustentabilidad en América Latina. (pp. 277 – 287). Puerto Montt, Chile: Taller Técnico Regional de la FAO.
Chakraborty, A., Sakai, M. y Iwatsuki, Y. (2006), Museum fish specimens and molecular taxonomy: A comparative study on DNA extraction protocols and preservation techniques. Journal of Applied Ichthyology, 22, 160-166
Checa, R. A. (2017). Extracción de ADN. Bioquímica y biología molecular, Pg. 3-4.
Chirichigno N, Velez J. (1998). Clave para Identificar los Peces Marinos del Perú. 2ed. Lima, Perú: IMARPE.
Coello, D., & Herrera, M. (2010). Diversidad de peces demersales en la plataforma continental de Ecuador. Rev. Cienc. Del Mar y Limnologia. 4 (1), 54-64.
Cummings, S. A., & Thorgaard, G. H. (1994). Extraction of DNA from fish blood and sperm. BioTechniques, 17(3), 426-430.
De Armas R, Rodríguez MM, Bisset JA, Fraga J. (2005). Modificación de un método de extracción de ADN genómico de Aedes aegypti (Diptera: Culicidae). Rev Colomb Entomol. 31(2), 203-206.
Dundass, B. (2008). Comparación de los métodos de extracción de ácidos nucleicos automatizados con la extracción manual. Diagnosticos moleculares, 311- 316.
Ferrer E., Da Conceicão, F., Campioli. P, Lares M, López M, Rivera MG, Viettri M, Medina M, Salcedo M, Morocoima A, Herrera L. (2009). Validación de protocolos de PCR para el diagnóstico molecular de la enfermedad de Chagas. Salus, 12(S1), 163- 174.
Fricke, R., Eschmeyer, W. N., & Van Der Laan, R. (2020). Eschmeyer’s Catalog of Fishes: genera, species, references. Updated.
García N. L. C. (2012). Caracterización morfológica y molecular de 34 colectas nativas de jitomate (Solanum lycopersicum L.). Tesis de Maestría en Biotecnologia Agricola. Departamento de Fitotecnia. Universidad Autónoma Chapingo. 56p.
Lamilla, J. 2004. Rhinobatos planiceps. In: IUCN 2013. IUCN Red List of Threatened Species. Version 2013.2.
Lamilla, J., & Sáez, S. (2003). Clave taxonómica para el reconocimiento de especies de rayas chilenas (Chondrichthyes, Batoidei). Investigaciones Marinas, 31(2), 3-6.
Lopera-Barrero, N.M., Ribeiro, R.P., Sirol, R.N., Povh, J.A., Gomes, P.C., Vargas, L., & Streit, J. (2006). Genetic diversity in piracanjuba populations (Brycon orbignyanus) with the RAPD (Random Amplifi ed Polimorphic DNA) markers. Journal Animal Science 84,170.
Lopera-Barrero, N., Povh, J.A., Ribeiro, R.P., Gomes, P.C., Jacometo, C.B., & Da Silva T. (2008). Comparación de dos protocolos de extracción de ADN con muestras de aletas y larvas de peces extracción modificada en cloruro de sodio. Cien Inv Agr, 35(1), 77-86.
Monsalve, E. V. (2016). Categorización de especies marinas de mayor importancia comercial de la pesquería artesanal en la Región Lambayeque según los criterios de la Lista Roja Regional de la UICN. Universidad Nacional Pedro Ruiz Gallo. Lambayeque, Perú.
Nam, Y. K., Park, J. E., Kim, K. K., y Kim, D. S. (2003). A rapid and simple PCR-based method for analysis of transgenic fish using a restricted amount of fin tissue. Transgenic research, 12(4), 523–525.
Sambrook, J., Fritsch, E.F., y Maniatis, T., (1989). Clonación molecular: un manual de laboratorio, 2ª ed. Prensa de laboratorio de Cold Spring Harbor, Cold Spring Harbor, Nueva York.
Taberlet, P., Griffin, S., Goossens, B., Questiau, S., Manceau, V., Escaravage, N., Waits, L. P., y Bouvet, J. (1996). Reliable genotyping of samples with very low DNA quantities using PCR. Nucleic acids research, 24(16), 3189-3194.
Wasko, A. P., Martins, C., Oliveira, C., y Foresti, F. (2003). Non-destructive genetic sampling in fish. An improved method for DNA extraction from fish fins and scales. Hereditas, 138(3), 161-165.
Weber, L.P., Higgins, P.S., Carlson, R.I. y Janz, D.M. (2003), Development and validation of methods for measuring multiple biochemical indices of condition in juvenile fishes. Journal of Fish Biology, 63, 637-658.
Yue, G. H., & Orban, L. (2001). Rapid isolation of DNA from fresh and preserved fish scales for polymerase chain reaction. Marine biotechnology, 3(3), 199-204.