IDENTIFICACIÓN GENÉTICA DE BOVINO, PORCINO Y EQUINO EN PRODUCTOS DE ORIGEN ANIMAL
GENETIC IDENTIFICATION OF CATTLE, PIGS AND HORSES IN PRODUCTS OF ANIMAL ORIGIN
Yhan Carlos Rojas De La Cruz 1 *, Rufino Paucar Chanca 1, Jose Luis Contreras Paco 2
1 Laboratorio de Mejoramiento Genético, Facultad de Ciencias de Ingeniería, Universidad Nacional de Huancavelica, Huancavelica, Perú.
2 Laboratorio de Nutrición Animal y Evaluación de Alimentos, Facultad de Ciencias de Ingeniería, Universidad Nacional de Huancavelica, Huancavelica, Perú
Yhan Carlos Rojas De La Cruz
Artículo original
Recibido: 10 de agosto 2022
Aceptado: 7 de noviembre 2022
Resumen
La identificación de especies en alimentos de origen animal presenta elevada importancia tanto en cuestiones relacionados con la calidad como con la seguridad alimentaria. La adulteración en los alimentos es relevante por motivos económicos, religiosos, legislativos o de salud pública. El objetivo de este estudio se centró en identificar especies animales (bovino, porcino y equino) en productos cárnicos a fin de determinar la adulteración en la venta de mortadela (MO), hot dog (H.D), chicharon de prensa (C.P), chorizo (CH) y jamonada (JA). El ADN fue extraído de 5 regiones diferentes de cada embutido y se realizó la PCR utilizando primers especie-específico. El amplicón de la PCR de la región especie-específico fue de 271, 212 y 145 pb para bovino, porcino y equino, respectivamente. El H.D fue el único producto amplificado por los primers específicos de bovino. El C.P y CH fueron amplificados por los primers de porcino, dado a que ambos productos contenían carne de esta especie. No se observó amplificación en MO, H.D y JA, pese a que estos últimos incluían a la carne de porcino según la etiqueta declarada por la compañía. Estos resultados muestran la existencia de adulteración en la venta de estos productos. Para el caso de los primers de equino, ningún producto cárnico ha sido amplificado. Se concluye que hubo presencia de adulteración en ciertos productos, lo cual representa un riesgo potencial para la salud pública y pérdidas económicas para los consumidores.
Palabras clave: Adulteración, identificación genética, PCR, productos cárnicos
Abstract
The identification of species in animal-origin food is highly important in both quality and food safety issues. Adulteration in food is relevant for economic, religious, legislative or public health reasons. The objective of this study focused on identifying animal species (cattle, pigs and horses) in meat products in order to determine adulteration in the sale of mortadella (MO), hot dog (HD), pressed pork crackling (CP), chorizo (CH) and ham (JA). DNA was extracted from 5 different regions of each sausage and PCR was performed using species-specific primers. The species-specific region PCR amplicon was 271, 212 and 145 bp for cattle, pig, and horse meat, respectively. H.D was the only product amplified by the bovine-specific primers. The C.P and CH were amplified by the pig primers, since both products contained meat of this species. No amplification was observed in MO, H.D and JA, despite the fact that these included pork, according to the label declared by the company. These results show the existence of adulteration in the sale of these products. In the case of equine primers, no meat product has been amplified. It is concluded that there was a possible presence of adulteration in certain products, which represents a potential risk to public health and economic losses for consumers.
Keywords: Adulteration, genetic identification, meat products, PCR
* Autor para correspondencia: E. mail: yhan23ro@gmail.com
Citar como:
Rojas, Y., Paucar, R., Contreras, J. 2022. Identificación genética de bovino, porcino y equino en productos de origen animal. REBIOL, 42(2): 71-76.
1. Introducción
La autenticación de especies, la seguridad alimentaria y el control de los alimentos son una preocupación creciente en el mercado actual de todo el mundo (Barakat et al., 2014). Aunque existen varias leyes nacionales e internacionales que supervisan la calidad y seguridad de la carne y los productos cárnicos, la adulteración continúa siendo una práctica global (Li et al., 2020). La gran parte de la adulteración de los productos cárnicos tiene una motivación económica y en cualquier caso puede provocar graves riesgos para la salud pública, cómo la exposición a toxinas, patógenos o alérgenos en estos productos (Magiati et al., 2019; Zade, 2002).
El etiquetado incorrecto y la adulteración de productos cárnicos pueden ser intencionales o no (Cawthorn et al., 2013; Bottaro et al., 2014). El etiquetado incorrecto intencional consiste en la sustitución o adición de carne de bajo coste que no está declarada en la lista de ingredientes del producto (Ballin, 2010). Por el contrario, el etiquetado incorrecto no intencional suele deberse a una contaminación cruzada que habitualmente se produce cuando el equipo utilizado para procesar más de una especie de carne no se limpia apropiadamente (Bottaro et al., 2014). En cualquier caso, la identificación de especies en alimentos de origen animal es una cuestión fundamental en el control de alimentos (Colombo et al., 2002; Djurkin et al., 2017).
Existen varios métodos rápidos y fiables para la identificación de especies animales en productos procesados (Kumar et al., 2013). Tales métodos incluyen a la PCR especie-específico, PCR-RFLP, ADN barcoding, PCR-RAPD, entre otros. La PCR especie-específico se basa en la utilización de primers que amplifican únicamente el fragmento de interés de la especie diana. Varios estudios han demostrado la eficacia de esta técnica en la identificación de especies animales en productos cárnicos procesados con la finalidad de detectar adulteración (Mohamed et al., 2007; Salah et al., 2009; Keyvan et al., 2017; Tembe et al., 2018).
En nuestro país, la información disponible en relación a la identificación de especies en productos cárnicos es inexistente. Una supervisión eficiente es crucial para garantizar el adecuado desarrollo de la industria cárnica y las tecnologías de detección rápidas, efectivas, precisas y confiables son un soporte técnico imprescindible para este propósito. Por lo expuesto, el objetivo de este estudio fue identificar especies tales como bovino, porcino y equino en productos cárnicos y simultáneamente detectar la presencia de adulteración.
2. Materiales y Métodos
El presente estudio se desarrolló en la Región, Departamento y Provincia de Huancavelica en la localidad de Paturpampa, que se encuentra ubicado a una longitud oeste 12º 46' 67''S, Latitud sur 74º 57' 56''O, Altitud 3,760 m.s.n.m., en las instalaciones del Laboratorio de Mejoramiento Genético de la Universidad Nacional de Huancavelica. Se utilizó distintos embutidos tales como mortadela, hot dog, chicharon de prensa, chorizo y jamonada, comprados en diferentes tiendas comerciales de la ciudad de Huancavelica y almacenados a -20°C hasta la extracción del ADN. Los diferentes embutidos contenían diferentes especies según lo declarado en sus etiquetas: Mortadela (pollo, pavo y cerdo), hot dog (cerdo y res), chicharon de prensa (cerdo), chorizo (cerdo y pavo) y jamonada (pollo, pavo y cerdo). El ADN fue extraído de 5 regiones diferentes de cada embutido y se realizó la PCR utilizando primers especie-específico.
Extracción de ADN
La extracción de ADN genómico se realizó con el Quick-DNA™ 96 Plus Kit de acuerdo con las instrucciones del protocolo. La concentración y pureza del ADN se determinó utilizando el Espectrofotómetro NanoPhotometer ™ UV / VIS (Implen GmBH, Germany).
Primers especie-específico y amplificación por PCR
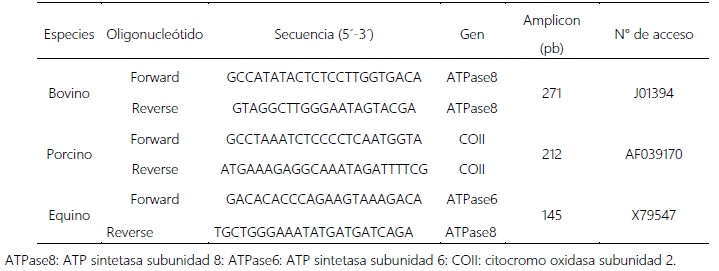
Los fragmentos de ADN especie-específicos de la especie bovino, porcino y equino se amplificaron con el uso de secuencias de los primers presentados en el Tabla 1 (Lahiff et al., 2001; Kesmen et al., 2009). Los tamaños del fragmento de la PCR de la región especie-específico fue de 271, 212 y 145 pb en el bovino, porcino y equino, respectivamente. La especificidad de cada par de primer especie-específico fue confirmado mediante la amplificación por PCR del ADN genómico de bovino, porcino y equino con cada set de primers. Para realizar la PCR se utilizó el Platinum PCR SuperMix de Invitrogen según las instrucciones del manual. La reacción de amplificación por PCR se realizó en un volumen de 60 μL; conteniendo 45 μL de Platinum PCR SuperMix, 5 μL de primers forward, 5 μL de primers reverse y 5 μL de ADN genómico. El Platinum PCR SuperMix a su vez contenía 22 U/mL de ADN Taq polimerasa recombinante complejada con platino Taq Anticuerpo, 22 mM Tris-HCl (pH 8,4), 55 mM KCl, 1,65 mM MgCl2, 220 μM dGTP, 220 μM dATP, 220 μM dTTP, 220 μM dCTP, y estabilizadores. Para mejorar la fiabilidad de la PCR, se utilizó ADN conocido como control positivo para cada especie. Después de un paso inicial de desnaturalización a 94 °C por 1 min, se realizaron 30 ciclos térmicos (Applied Biosystems™ SimpliAmp™ Thermal Cycler) de la siguiente manera: desnaturalización a 94 °C durante 50 seg, hibridación a 55 °C por 30 seg y extensión a 72 °C durante 1 min. Los productos de la amplificación de la PCR se determinaron mediante electroforesis en gel de agarosa al 1% y se visualizaron mediante fotodocumentador SmartView Pro 1100.
Tabla 1. Pares de primers utilizados en la identificación por PCR de bovino, porcino y equino.
3. Resultados y Discusión
El ADN genómico extraído de las muestras de embutidos (a excepción del chicharon de prensa y chorizo) presentaron un patrón común de degradación del ácido nucleico (Figura 1). Estos resultados coinciden con lo encontrado en estudios similares que consideran el análisis de productos procesados de origen de animal (Zalba, 2017: Martín et al., 2007). Generalmente la muestras que se someten a elevadas temperaturas tienden a desnaturalizar las moléculas de ADN. Por ello, es preferible utilizar primers dirigidos a segmentos de ADN inferiores a 200 pb en productos tratados térmicamente (Rodríguez et al., 2004).
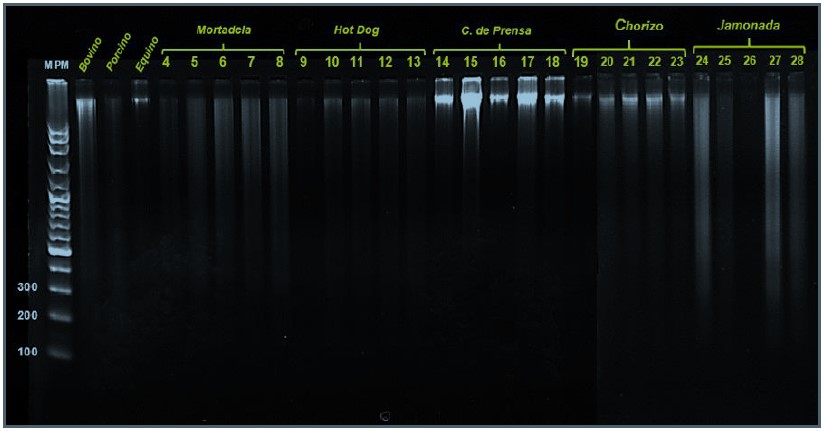
Figura 1. Electroforesis en gel del ADN genómico extraído de especies animales (bovino, porcino y equino) y embutidos (mortadela, hot dog, chicharon de prensa, chorizo y jamonada). M PM: marcador de peso molecular, 1 Kb plus DNA ladder.
La especificidad de cada par de primers especie-específico fue confirmado mediante la amplificación por PCR del ADN genómico de bovino, porcino y equino con cada set de primers, siendo utilizado este criterio en estudios de semejante naturaleza (Martín et al., 2007; Kesmen et al., 2007; Kesmen et al., 2009; Rojas et al., 2008). Los primers utilizados permitieron la amplificación del fragmento específico solamente en las muestras que contenían el ADN de la especie diana y no produjeron bandas cuando se utilizaron ADN procedente de otras especies animales. Los fragmentos específicos de ADN de 271, 212 y 145 pb se amplificaron satisfactoriamente con los pares de primers correspondientes al bovino, porcino y equino, respectivamente (Figura 2 y 3).
Para los primers de bovino se observó amplificación únicamente para el hot dog, pues este producto era el único que estaba constituido por carne de bovino y cerdo (Figura 2). Por el contrario, el resto de los productos no contenían carne de bovino según los ingredientes declarados en la etiqueta de los productos. Esto demuestra que para el caso de los primers de bovino no existió adulteración en la venta de los embutidos analizados.
En relación a los primers de porcino, se observó amplificación tanto para el chorizo y chicharon de prensa.
Sin embargo, no se observó amplificación para la mortadela, hot dog y jamonada, pese a que estos últimos estaban compuestos por carne de porcino según la etiqueta. Esté resultado ilustra la existencia de fraude en la comercialización de tales productos. En un estudio similar Tembe et al. (2018), utilizando la misma metodología encontraron que un 60% de salchichas de ternera estaban contaminadas con carne de pollo y cerdo, mientras que un 12,5% de empanadas de ternera también contenían carne de cerdo. Asimismo, otro estudio similar reveló que 43 de 80 muestras de salchicha de pollo presentaban casos de etiquetado incorrecto.
Específicamente, 24 muestras resultaron positivos para cerdo y bovino, 12 muestras dieron positivo para cerdo y 7 muestras fueron positivos para bovino (Bottaro et al., 2014).
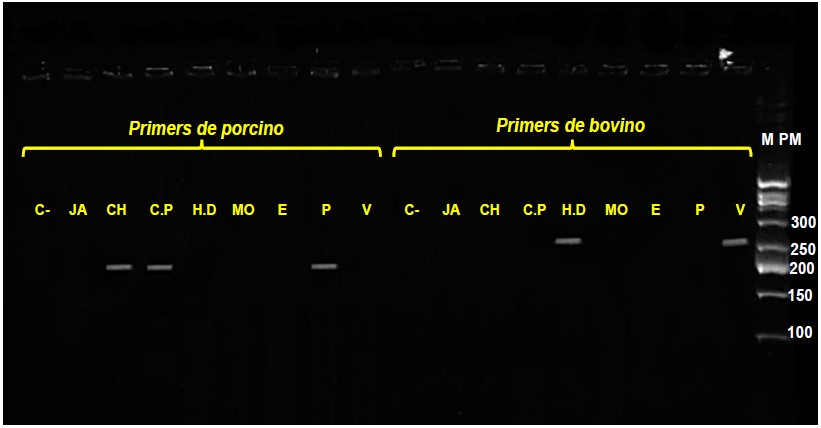
Figura 2. Fragmentos de PCR obtenidos usando primers específicos de bovino y porcino. M PM: marcador de peso molecular, 100-500 pb ladder, V; bovino, P; porcino, E; equino, MO; mortadela, H.D; hot dog, C.P; chicharon de prensa, CH; chorizo, JA; jamonada, C; control negativo.
Keyvan et al. (2017), encontraron cinco muestras con carne de ave y una con carne de ave y caballo juntas, en 37 muestras de sucuk, siete muestras con carne de ave en 32 muestras de salami y 2 muestras con carne de ave en 33 muestras de salchicha. En total, reportaron 15 muestras que contenían especies animales no declaradas en la etiqueta.
Concerniente a los primers utilizados para la especie equino, no se observó amplificación en ninguno de los productos analizados (Figura 3). Únicamente amplificó la especie equino, lo que demuestra la enorme especificidad de los primers utilizados. En general, para los tres pares de primers utilizados en el presente estudio no se observó amplificación en el control negativo, lo cual indica la ausencia de contaminación en el experimento.
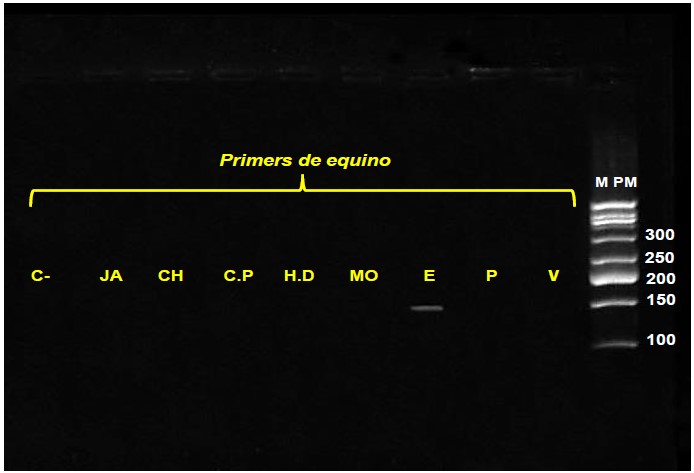
Figura 3. Fragmentos de PCR obtenidos usando primers específicos de equino. M PM: marcador de peso molecular, 100-500 pb ladder, V; bovino, P; porcino, E; equino, MO; mortadela, H.D; hot dog, C.P; chicharon de prensa, CH; chorizo, JA; jamonada, C; control negativo.
Debido a que la autenticación de especies animales en productos cárnicos es fundamental para salvaguardar al consumidor (Soares et al., 2013), el seguimiento constante para disminuir la adulteración es un requerimiento imprescindible para las autoridades quienes deben garantizar alimentos seguros, no adulterados y de buena calidad.
En comparación con las técnicas alternativas para la identificación de especies animales tales como la PCR-RFLP (Pascoal et al., 2004) o la secuenciación de ADN (Colombo et al., 2002), la PCR basado en la utilización de primers específicos brinda las ventajas de ser menos costosa y más útil para el análisis rutinario de gran cantidad de muestras
4. Conclusiones
Los tres fragmentos específicos presentes en el bovino, porcino y equino amplificaron de manera efectiva mediante la PCR cualitativa, permitiendo detectar la presencia/ausencia de estas especies en embutidos y a su vez detectar posibles adulteraciones en la venta de dichos productos.
5. Agradecimientos
Los autores expresan su agradecimiento al Fondo de Desarrollo Socioeconómico del Proyecto Camisea-FOCAM, por haber financiado el presente trabajo de investigación.
6. Contribución de los autores
Yhan Carlos Rojas De la Cruz: Adquisición de muestras, análisis e interpretación del experimento.
Rufino Paucar Chanca: Concepción y diseño del estudio
Jose Luis Contreras Paco: Borrador del artículo y revisión crítica del contenido intelectual.
7. Conflicto de interes
Los autores declaran que no existe conflicto de interés.
8. Referencias Bibliográficas
Barakat, H., El-Garhy, H.A., Moustafa, M.M. (2014). Detection of pork adulteration in processed meat by species-specific PCR-QIAxcel procedure based on D-loop and cytb genes. Appl Microbiol Biotechnol 98, 9805-9816.
Ballin, N.Z. (2010). Authentication of meat and meat products. Meat Science, 86, 577-587.
Bottaro, M.P., Marchett, A., Mottola, F., Shehuf, A., Pinto, D.I. (2014). Detection of mislabeling in packaged chicken sausages by PCR. Albanian J. Agric. Sci. 455-460.
Cawthorn, D.M., Steinman, H.A., Hoffman, L.C. (2013). High incidence of species substitution and mislabelling detected in meat products sold in South Africa. Food Control, 32, 440-449.
Colombo, F., Marchisio, E., Pizzini, A., Cantoni, C. (2002). Identification of goose species (Anser anser) in Italian mortara salami by DNA sequencing and a polymerase chain reaction with an original primer pair. Meat Sci 61, 291-294.
Djurkin, K.I., Samac, D., Margeta, V., Radišić, Ž., Vincek, D., Kušec, G. (2017). Efficiency of PCR-RFLP and species-specific PCR for the identification of meat origin in dry sausages. Czech J. Food Sci 35, 386-391.
Kesmen, Z., Gulluce, A., Sahin, F., Yetim, H. (2009). Identification of meat species by TaqMan-based real-time PCR assay. Meat Sci 82(4), 444-449.
Kesmen, Z., Sahin, F., Yetim, H. (2007). PCR assay for the identification of animal species in cooked sausages. Meat Science, 77, 649-653.
Keyvan, E., İPLİKÇİOĞLU, Ç.G., Bengi, Ç.K., Bilgen, N., ŞİRELİ, U.T. (2017). Identification of meat species in different types of meat products by PCR. Ankara Üniv Vet Fak Derg 64, 261-266.
Kumar, A., Kumar, R.R., Sharma, B.D., Gokulakrishnan, B., Mendiratta, S.J., Sharma, D. (2015). Identification of Species Origin of Meat and Meat Products on the DNA Basis: A Review, Critical Reviews in Food Science and Nutrition, 55 (10). 1340-1351.
Lahiff, S., Glennon, M., O´Brien, L., Lyng, J., Smith, T., Maher, M., Shilton, N. (2001). Species-specific PCR for the identification of ovine, porcine and chicken species in meat and bone meal (MBM). Molecular and Cellular Probes 15, 27-35.
Li, Y.C., Liu, S.Y., Meng, F.B., Liu, D.Y., Zhang, Y., Wang, W., Zhang, J.M. (2020). Comparative review and the recent progress in detection technologies of meat product adulteration. Compr Rev Food Sci Food Saf, 19, 2256-2296.
Magiati, M., Myridaki, V.M., Christopoulos, T.K., Kalogianni, D.P. (2019). Lateral flow test for meat authentication with visual detection. Food Chemistry, 274, 803–807.
Martín, I., García, T., Fajardo, V., López-Calleja, I., Rojas, M., Pavón, M.A., Martín, R. (2007). Technical note: Detection of chicken, turkey, duck, and goose tissues in feedstuffs using species-specific polymerase chain reaction. Journal of Animal Science, 85(2), 452-458.
Mohamed, M.M., Salah, M.A., Amr, A.E. (2007). Application of Species-Specific Polymerase Chain Reaction and Cytocrome b Gene for Different Meat Species Authentication. Biotechnology, 6, 426-430.
Pascoal, A., Prado, M., Castro, J., Cepeda, A., Barros-Velázquez, J. (2004). Survey of authenticity of meat species in food products subjected to different technological processes, by means of PCR-RFLP analysis. Eur. Food Res. Technol 218, 306–312.
Rodriguez, M.A., Garcia, T., Gonzalez, I., Asensio, L., Hernández, P.E., Martin, R. (2004). PCR identification of Beef, Sheep, Goat, and Pork in Raw and Heat-Treated Meat Mixtures. Journal of Food Protection 67(1), 172–177.
Rojas, M., González, I., Martín, R. (2008). Autentificación de carne y productos cárnicos procedentes de codorniz, faisán, perdíz y pintada mediante una técnica de PCR con cebadores especie-específicos. Revista Complutense de Ciencias Veterinarias 2(2), 199-206.
Salah, M., Mohamed, A., Khallid, M., Amany, S. (2009). Detection of Adulteration and Identification of Cat’s, Dog’s, Donkey’s and Horse’s Meat Using Species-Specific PCR and PCR-RFLP Techniques. Australian Journal of Basic and Applied Sciences 3(3), 1716-1719.
Soares, S., Amaral, J.S., Oliveira, M.B., Mafra, I. (2013). A SYBR Green real-time PCR assay to detect and quantify pork meat in processed poultry meat products. Meat Science 94, 115-120.
Tembe, D., Mukaratirwa, S., Zishiri, O.T. (2018). Undeclared Meat Species in Processed Meat Products from Retail Franchises in the Durban Metropole, KwaZulu-Natal Province, South Africa, Using Species-specific DNA Primers. Food Protection Trends 38(6), 440-449.
Zade, N.N. (2002). Methods and medicolegal aspects of meat speciation. In: Advances in Veterinary Public Health. Edi. Singh, S. P., Mahesh Kumar and Arun Kumar, IAVPHS Publication, 191-205.
Zalba, T. (2017). Detección de especies de atún (Género Thunnus) de alto valor comercial en conservas y en productos frescos. [Tesis para título. Universidad Pública de Navarra, España].