MEJORAMIENTO GENÉTICO EN PLANTAS AUTÓGAMAS
GENETIC IMPROVEMENT IN AUTOGAMOUS PLANTS
Juan Javier Pedro Huaman1*; Willian Genaro Blas Cerdán1; Dayanna Brigitte Reyes Pita1;
Lizbeth Fiorella Rodríguez Luján1; Sofía Fernanda Rosales Rodríguez1; Jannela Ruíz Ángeles1;
Fiorella Maribel Salas Pizarro1; Milushka Nicole Sánchez Llatas1; Luciana Sossiré Vásquez
Regalado1; Jackeline Varas Rodríguez1; Evelyn Elisabet Ventura Aguilar1; Wilfredo Eduardo
Villena Ulloa1; Yuri Irvin Yabar Rosales1
1 Facultad de Ciencias Biológicas, Universidad Nacional de Trujillo, Av. Juan Pablo II s/n – Ciudad Universitaria, Trujillo, Perú.
Artículo de revisión
Recibido: 22 de marzo 2021
Aceptado: 10 de junio 2021
Resumen
El avance de los métodos tradicionales y modernos para el mejoramiento de las plantas autógamas se ha incrementado en las últimas décadas, ambos métodos son las principales herramientas para conseguir incrementar la producción de los cultivos. Entre los métodos tradicionales más usados se tiene a la hibridación que se basa en el cruzamiento de individuos con diferentes genotipos, la selección; entre ellas a la selección masal, la selección por pedigree y el retrocruzamiento. En los métodos modernos tenemos: Selección asistida por marcadores moleculares, que combina muy bien con el mejoramiento tradicional; así también la Inducción de mutaciones cuando la variabilidad genética es baja; el cultivo de tejidos in vitro, la Ingeniería Genética y Mejoramiento de Genes. En el presente trabajo explicamos algunos ejemplos en plantas autógamas como en el caso de Phaseolus vulgaris “frijol” orientándose a mejora en condiciones de sequía principalmente, así como el mejoramiento genético de la Quinua, la cual presenta alogamia frecuente (0,5 a 17%), y por último tenemos el mejoramiento genético en el cultivo de arroz, utilizando marcadores moleculares para etiquetar los genes de interés. En conclusión, los métodos genéticos tradicionales nos dan alternativas sencillas y de bajo costo, aunque toman un mayor tiempo, por lo cual se está orientando a combinar los métodos tradicionales con métodos modernos.
Palabras claves: Plantas autógamas, selección, hibridación, retrocruzamiento, mejoramiento genético.
Abstract
The advance of traditional and modern methods for the improvement of self-pollinated plants has increased in recent decades, both methods are the main tools to achieve increased crop production. Among the most widely used traditional methods, hybridization is based on the crossing of individuals with different genotypes, selection; including mass selection, pedigree selection and backcrossing. In modern methods we have: Selection assisted by molecular markers, which combines very well with traditional breeding; thus also the Induction of mutations when the genetic variability is low; in vitro tissue culture, Genetic Engineering and Gene Improvement. In the present work we explain some examples in autogamous plants such as in the case of Phaseolus vulgaris "bean", mainly oriented towards improvement in drought conditions, as well as the genetic improvement of Quinoa, which frequently presents allogamy (0.5 to 17%), and finally we have genetic improvement in rice cultivation, using molecular markers to label the genes of interest. In conclusion, traditional genetic methods give us simple and low-cost alternatives, although they take a longer time, so it is being oriented to combine traditional methods with modern methods.
Keywords: Autogamous plants, selection, hybridization, backcrossing, genetic improvement.
*Autor responsable: jpedro@unitru.edu.pe
DOI: http://dx.doi.org/10.17268/rebiol.2021.41.01.14
Citar como:
Pedro, J., Blas, W., Reyes, D., Rodríguez, L., Rosales, S., Ruíz, J., Salas, F., Sánchez, M., Vásquez, L., Varas, J., Ventura, E., Villena, W., & Yabar, Y. 2021. Mejoramiento genético en plantas autógamas. REBIOL, 41(1):136-153.
1. Introducción
Las plantas autógamas se caracterizan por tener un alto porcentaje de autofecundación, en relación a la polinización cruzada natural. Este grupo de plantas normalmente presentan poblaciones que se conforman de solo una línea totalmente pura o una combinación de dos o más líneas también puras, por lo que puede formar poblaciones homogéneas o heterogéneas (Angulo & Ortiz, 2020).
En este último caso, cuando la población está formada por varios genotipos homocigotos distintos debido a mutaciones cuyo origen se debe a una forma híbrida, y por autofecundaciones sucesivas dio una progenie que poco a poco se fue separando en un determinado número de homocigotos (Camarena et al., 2014).
Basándose en los conocimientos teóricos, si una especie es exclusivamente autógama, en un futuro estará condenada a fracasar ya que no tendría forma de adquirir nuevas características y tendería a un declive. Entonces, al momento de hablar de plantas autógamas, se hará referencia a dichas especies como principalmente autógamas, aunque de una forma permanente van a presentar una diminuta alogamia ocasional, como parte de su mecanismo evolutivo (Vallejo & Estrada, 2016).
Para comprender más a fondo la importancia de las plantas autógamas, se mencionará las siguientes características (Peréz et al., 2019).
Un ejemplo de mejoramiento genético que se dio en el Perú fue en Solanum tuberosum “papa” como respuesta para solucionar la problemática en las deficiencias de hierro y zinc como las principales causas de la desnutrición en la población, de acuerdo a estudios realizados se evidenciaron que las enfermedades carenciales son mayores en las zonas rurales que en las zonas urbanizadas, por lo cual se procedió a realizar mejoramiento genético de este cultivo, con el fin de contribuir a una reducción en la mala nutrición causada por la deficiencia de micronutrientes (Hualla, 2017). Es por ello, que en el Centro Internacional de la Papa (CIP) se desarrollaron trabajos de mejoramiento genético en estas plantas para aumentar los niveles de micronutrientes esenciales como el hierro y zinc a través de la biofortificación de papa utilizando una población inicial diploide de los grupos taxonómicos stenotomum, goniocalyx y phureja, a través de métodos de selección recurrente que permite un aumento gradual de la frecuencia de genes favorables de la población (FAO et al., 2013).
Tabla 1. Características genéticas de las plantas autógamas

2. Materiales y Métodos
CARACTERÍSTICAS GENERALES DE LAS PLANTAS AUTÓGAMAS:
Las plantas autógamas se llaman así porque su reproducción se da por autofecundación, se encuentran conformadas por una mezcla única de homocigotos, estas especies presentan un porcentaje muy bajo de fecundación cruzada de forma natural. En este tipo de plantas no se encuentra presente el cruzamiento genético, es decir no realizan el intercambio de sus genes con otras plantas por lo que tienen un genotipo permanente, es por ello, que cuando se requiere realizar mejoramiento, existe un mayor interés por evaluar las frecuencias genotípicas de la población. Las plantas autógamas se consideran como líneas puras porque descienden de sí mismas por autofecundación, obteniendo líneas homocigotas para todos sus caracteres (Vallejo & Estrada, 2016).
Las plantas autógamas tienen la misma constitución genética y son uniforme en todos sus caracteres, sin embargo, en estas especies se puede llegar a encontrar algunos casos de heterogeneidad y esto se debe a mutaciones o a la combinación de loci que pudo darse en el progreso evolutivo de estas poblaciones. Finalmente, una de las principales características de las plantas autógamas se debe a que pueden tolerar la endogamia sin perder su vigor, es así cuando ocurre selección en generaciones segregantes se logra aislar líneas puras homocigotas (Poehlman, 1971).
PRINCIPALES PLANTAS AUTÓGAMAS DE INTERÉS NACIONAL E INTERNACIONAL
A nivel mundial, se puede considerar una serie de especies autógamas, permitiendo su mejoramiento a partir de diferentes métodos que se explicarán en el presente estudio.
Entonces, se mencionarán algunas de estas especies que generan grandes toneladas de producción para el consumo humano. Por ejemplo, en zonas altas de América Latina, encontramos al “frijol ñuña”, planta altoandina, subespecie de Phaseolus vulgaris L. “frijol común”, que cumple una importante función en la nutrición de los pobladores debido a que contiene 20% de proteínas y 62% de carbohidratos (Camarena et al., 2014). También tenemos a Solanum lycopersicum “tomate”, una de las plantas nativas considerada dentro de las más importantes hortalizas a nivel mundial; esto debido a que, entre otras razones, posee una fuente de minerales, vitaminas y antioxidantes como el licopeno, así mismo, tiene un gran potencial para su mejoramiento genético debido a la exposición de muchas enfermedades y factores ambientales adversos a los cuales pueden estar sometidas (Cuya, 2018).
Otra de las plantas a considerar, es Solanum tuberosum “papa” que se divide en dos subespecies: tuberosum y andigenum, las más importantes de acuerdo con el área sembrada y producción; sin embargo, existe un grupo phureja que es especialmente importante, debido a que posee una fuente de genes para su mejoramiento con respecto a la resistencia de patógenos, como virus, bacterias y algunos insectos (Seminario et al., 2018). Así mismo, cabe mencionar que el Perú en el año 2014, fue el segundo país con mayor producción de papa en América, detrás de Estados Unidos (Reategui et al., 2019).
El arroz, considerado como alimento base de países árabes y de ciertos países de Latinoamérica, como Perú, se han desarrollado nuevas variedades de dicho cultivo llamadas “IR-43”, “Tinajones”, “Mallares”, “Fortaleza”, “Capirona”, “La Esperanza” y "La Conquista" (Camarena et al., 2014).
Otras especies de plantas autógamas se lograron obtener por selección ejecutadas por culturas peruanas antiguas, son especies vegetales con alto valor nutritivo, como Lupinus mutabilis Sweet “chocho” o “tarwi” que contiene un alto valor de proteínas, presenta variedades: Cusco, Huamachuco la planta es tolerante a antracnosis, H6 de buen rendimiento, Sacacatani de alto rendimiento y tardío (Iris, 2018). Chenopodium quinoa “quinua”; es un grano que posee un alto valor nutritivo, debido a que aporta ácidos grasos insaturados, proteínas y minerales, presentando una extensa variabilidad genética permitiendo así adaptarse a diversos ambientes ecológicos; por lo cual, el Ministerio de Desarrollo Agrario y Riego (MIDAGRI) menciona que existen alrededor de 100 variedades de quinua, de las cuales en Perú se cuenta con 11 de ellas: Quillahuaman, Salcedo INIA, Illpa INIA, INIA 415 Pasankalla, INIA 420 Negra Collana, INIA 427 Amarilla Sacaca, INIA 431 Altiplano, INIA 433 Santa Ana, INIA 437 Roja del Norte, INIA 438 Acollina y la nueva variedad INIA 441 - Señor del Huerto desarrollada por medio del Instituto Nacional de Innovación Agraria (INIA) en la región de Ayacucho (Ministerio de Desarrollo Agrario y Riego, 2021).
Cultivos que no son oriundos del Perú pero poseen una alta importancia en la dieta principalmente de las poblaciones altoandinas son Triticum aestivum “trigo”, se considera un alimento contribuyente del 20% del total de las calorías que son ingeridas en las zonas rurales; se conoce a la variedad llamada “OLLANTA”, un trigo harinero con un parámetro de plasticidad para tolerar los distintos tipos de enfermedades causadas por la “Roya Amarilla” (Lizarraga, 2015). Hordeum vulgare L. “cebada” uno de los cultivos con una amplia adaptación; presenta dos ecovariedades: distichum, conocida como cebada cervecera, y vulgare llamada cebada forrajera (Velasco et al., 2020). Este cultivo tiene una función importante en el rendimiento de grano, la calidad física del grano, la cual permiten mejorar la estabilidad del rendimiento, debido a ello es que se desarrollan variedades (Pérez et al., 2019).
MÉTODOS DE MEJORAMIENTO TRADICIONAL EN PLANTAS AUTÓGAMAS
Hibridación
Este modelo se sustenta en el cruzamiento de individuos con diferentes genotipos para lograr obtener las características de los genes deseables en la nueva generación. Este método se usa como principal estrategia para mejorar las especies ya existentes en especies superiores. Para obtener buenos resultados, se seleccionan los progenitores, teniendo en cuenta las características que se desea mezclar (Angulo & Ortiz, 2020). La hibridación puede combinar las características biológicas de especies distantes, que rompe los límites de las especies y amplifica variaciones genéticas dando como resultado la creación de nuevas variedades (Liu et al., 2020). Se puede clasificar a la hibridación en dos tipos en función de los tipos de cultivos involucrados: la hibridación interespecífica o también llamada hibridación a distancia y la hibridación intraespecífica o llamada hibridación cercana.
La hibridación a distancia se refiere a cruces entre especies diferentes, en contraste con la hibridación cercana, que ocurre entre variedades de la misma especie o subespecie. El concepto de hibridación distante se refiere a la hibridación entre biotipos con diferentes distribuciones geográficas (Chen et al., 2017).
La hibridación cercana, o la hibridación intraespecífica, también puede conducir a la mutación genética en la descendencia híbrida; sin embargo, los rangos de variación y reproducción genética resultantes son limitados. Los híbridos distantes pueden contener los genomas de diferentes especies, sentando las bases para la recombinación entre los genes. La formación de cepas híbridas distantes fértiles puede crear nuevos tipos de variación, e incluso nuevas especies. En términos de fenotipo, la hibridación distante integra las fortalezas de ambos padres para conferir ventajas híbridas en relación con la apariencia, el crecimiento, tasas de supervivencia, resistencia a enfermedades y otros rasgos. En términos de genotipo, la hibridación distante provoca cambios a nivel del genoma para producir diploides, triploides y tetraploides. La hibridación distante también conduce a la aparición de la variación del ADN recombinante en la descendencia. En la figura 1, podemos observar un diagrama que representa el procedimiento que se lleva a cabo para la obtención de plantas híbridas (Liu, 2010; Liu et al., 2014; Liu et al., 2020).
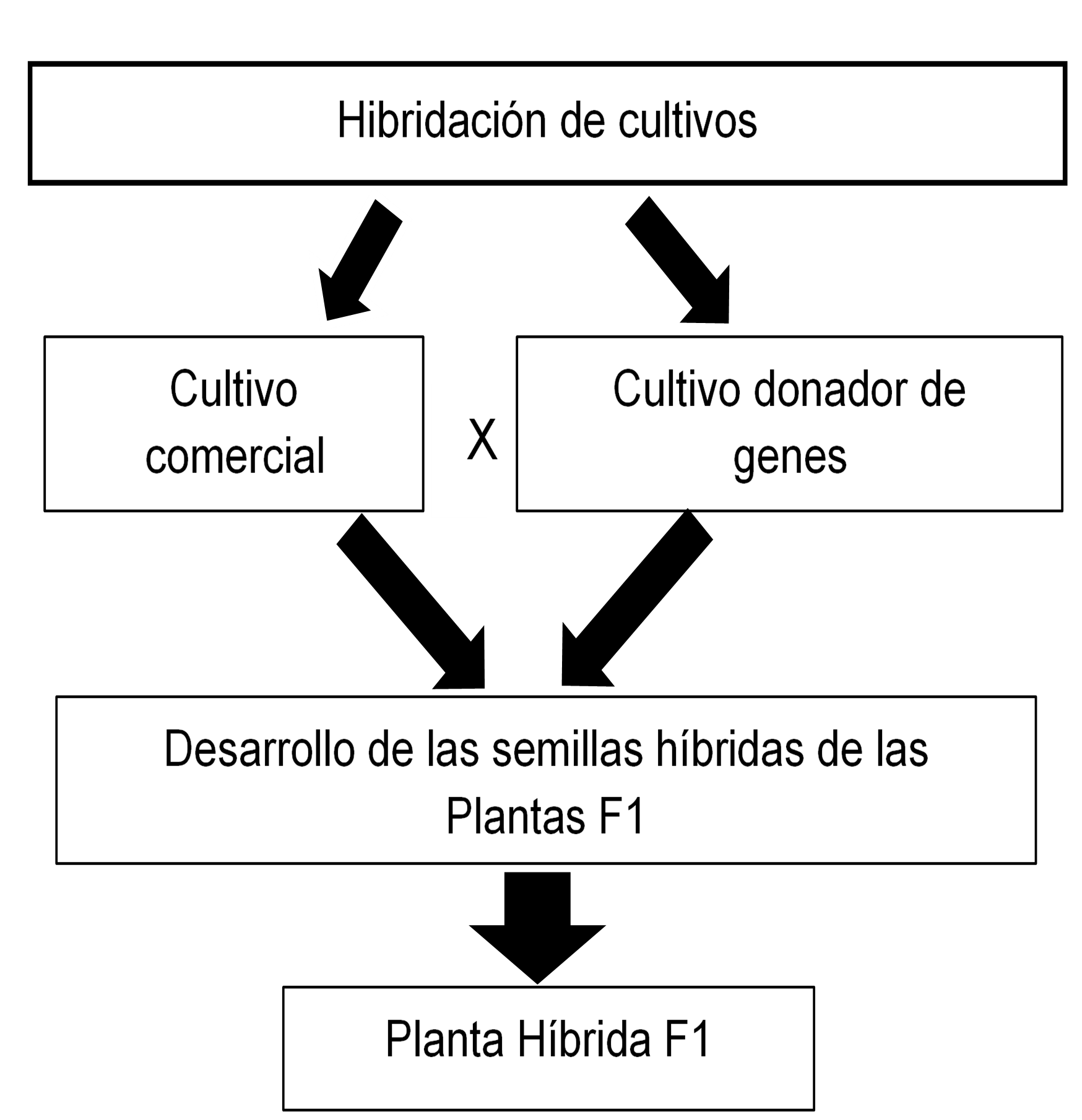
Figura 1. Esquema de hibridación de cultivos
Nota: Adaptado de Liu et al. (2014).
De acuerdo con Poehlman (1971), la hibridación incluye los siguientes métodos para la obtención de nuevas variedades, se puede elegir el más adecuado según la necesidad y características del cultivo:
Remoción de anteras
Este proceso es denominado emasculación y consiste en utilizar pinzas y otros instrumentos de precisión para remover las anteras eliminando completamente el polen antes de que ocurriera la polinización. La precisión y el grado de dificultad están relacionados con la envoltura floral.
Destrucción de polen por medio de calor, frío o alcohol
Se utiliza diferentes medios para hacer inviable el polen mediante el uso de calor, frio o alcohol, la elección del método dependerá de la resistencia y condiciones propias de cada cultivo, principalmente en especies gramíneas como el sorgo, arroz y otras soportan temperaturas de 45 a 48ºC las cuales son suficientes para hacer inviables los granos de polen, así como el uso de agua fría hasta casi el punto de congelación tiene un efecto similar. Lo importante es asegurar que el polen no haya fecundado las flores para estar seguros de que no ocurrió una autofecundación que imposibilitaría la hibridación.
Polinización sin emasculación
Existen algunos cultivos que no necesitan emasculación ni hacer inviable el polen puesto que presentan autoincompatibilidad por lo cual no pueden ser fecundadas por sus propios granos de polen, estas pertenecen al grupo de plantas androestériles que incluyen algunas que no llegan a desarrollar estructuras masculinas, por ejemplo, todas estas plantas se pueden hibridar de manera directa puesto que no tienen posibilidad de que se hayan autofecundado de manera natural. Por último, cabe resaltar que las plantas no son 100% androestériles por lo cual es necesario considerar dicha característica antes de elegir si se aplicara un método de emasculación o no (Poehlman, 1971).
Selección
Este método cumple con la acción de seleccionar plantas dentro de una población que presente características favorables, llegando así a una reducción en la variabilidad genética. Dentro de este método encontramos al método por Selección masal, este método implica a un gran número de individuos con características fenotípicas que aportan en la siguiente generación, este método es muy usado para poblaciones heterogéneas formadas por líneas puras (Angulo & Ortiz, 2020).
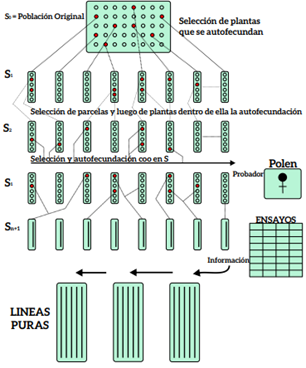
Figura 2. Selección individual
Nota: Esta es obtenida por el proceso de autofecundación hasta una homocigosis total, para la obtención de líneas puras. Tomado de Mejoramiento de plantas Autógamas (p. 57) por Cubero (2003).
El método de selección masal puede ser usada para diversos casos como: para variedades locales, dentro de este caso tenemos selección masal única, repetida y con separación de formas, cada uno de estos casos fue utilizado para diferentes mejoras en diversos cultivares, de este método es importante para purificación de variedades, siendo usado para determinar la presencia de fenotipos diferentes a los originales (Quispe, 2017).
Otro método muy usado es la Selección genealógica (método por pedigree). Este método se basa en el aislamiento del mejor genotipo, debido a que se seleccionan las plantas que presenten mejores características, logrando así presentarlas como nuevos cultivos. En la figura 3, podemos observar un diagrama de la selección mediante el método genealógico (Vallejo & Salazar, 2016).
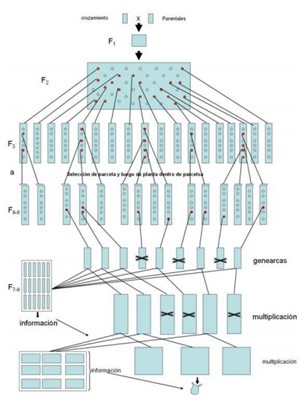
Figura 3. El método genealógico (Pedigree)
Nota: Este método se basa en el aislamiento del mejor genotipo, debido a que se seleccionan las plantas que presenten mejores características. Tomado de Mejoramiento de plantas Autógamas por Cubero (2003).
Retrocruzamiento
El retrocruzamiento tiene como finalidad conseguir variedades resistentes a cualquier patógeno o enfermedad, así como la modificación de características cualitativas, este método tradicional nos permite lograr la modificación de cualquier carácter, sólo sí presenta una alta tasa de heredabilidad (Arteaga, 2011).
En un retrocruzamiento, tenemos 2 factores importantes, el progenitor recurrente y el progenitor donante. El primero conserva casi la totalidad del genotipo y es con quién se cruza sucesivamente al híbrido. En cambio, el segundo a partir del cual se intenta transferir uno o más genes al progenitor recurrente. En el retrocruzamiento generalmente se transmite un solo gen o unos pocos genes total o parcialmente dominantes. Aunque también es posible transferir genes recesivos, pero realizar ese procedimiento resulta mucho más complicado y laborioso, ya que para ello primero es necesario realizar la autofecundación después del primer retrocruzamiento, con el propósito de obtener homocigotos que se puedan retrocruzar nuevamente entre sí mismos (Lea, 2017). A continuación, presentamos un esquema de retrocruzamiento donde se busca transferir un gen de resistencia a una variedad comercial de adecuado rendimiento (Figura 4):
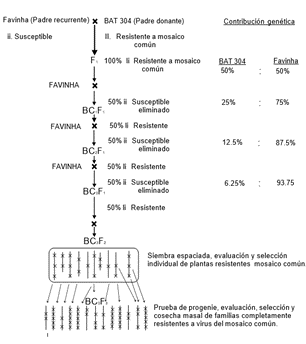
Figura 4. Retrocruzamiento
Nota: Este método se basa en la transferencia del gen dominante II de resistencia para virus del mosaico común (VMC). Tomado de Conceptos básicos para el mejoramiento del frijol por Singh (1985).
Otros métodos
La hibridación, la selección y el retrocruzamiento no son los únicos métodos de mejoramiento tradicional en plantas autógamas, sino que por el contario existe una gran variedad de ellos, que pueden ir combinándose inclusive con algunos conceptos más actuales que permiten mejorar los logros obtenidos en el mejoramiento genómico vegetal (Hualla, 2017).
La asociación del método de pedigrí con el procedimiento REML/Blup puede ser útil para ayudar a los fitomejoradores en un programa de reproducción autógama. Se seleccionaron seis líneas dentro de las progenies superiores para continuar el programa de desarrollo de líneas grupales de Formosa. Por otro lado, tenemos el método poblacional o masal, el cual hace referencia a la obtención de líneas homocigotas. Finalmente, tenemos el método de la descendencia de semilla única o S.S.D, este procedimiento consiste en usar una semilla de cada planta de la F2 (segunda generación) para obtener la F3 (tercera generación) y de tal forma consecutivamente (Cortes et al., 2019).
MÉTODOS DE MEJORAMIENTO MODERNO EN PLANTAS AUTÓGAMAS
Selección asistida por marcadores moleculares
La selección asistida por marcadores moleculares (SAM) representa una eficaz herramienta biotecnológica que nos da la facultad de seleccionar plantas con ciertas propiedades o características de interés en cualquier estadio de sus etapas fenológicas. Esta técnica se basa en la identificación de secuencias de ADN muy relacionadas entre sí con características de productividad (marcador molecular). Debido a ello, podemos entender a la SAM como la selección de una propiedad o característica determinada en una planta por medio de la presencia de un marcador molecular (Arruabarrena et al., 2015).
Dicha técnica se sustenta en dos correlaciones: Locus de rasgo cuantitativo y Locus de rasgo cuantitativo marcador. Un locus de rasgo cuantitativo (QTL), se precisa como una posición genética (locus) relacionada o asociada con cierto grado de variación fenotípica en un rasgo específico. Estos marcadores representan polimorfismos ensayables con cierto grado de asociación con un QTL en un acervo genético determinado. Ambos conjuntos de correlaciones pueden ser de amplio alcance o poseer una aplicación limitada, y el éxito o la fiabilidad de SAM está directamente determinada por la fuerza de dichas correlaciones. El hecho de que tan próximos o relacionados se encuentren el marcador molecular del gen y el locus que contiene el rasgo de interés agronómico representa el efecto de ligamiento genético que existe entre los mismos comprendida en una población determinada (Platten et al., 2019).
La selección asistida por marcadores moleculares se ha venido empleando con el objetivo de aislar y seleccionar genotipos con las características deseadas directamente a nivel de ADN. Esta técnica trae varios beneficios cuando se utiliza con otros métodos de mejoramiento convencional, como lo son el bajo costo, el rápido desarrollo en el avance de la generación y una alta eficiencia de selección. Una de las grandes ventajas de SAM en comparación con los métodos de mejoramiento tradicionales es la reducción en el tiempo de obtención del genotipo deseado, puesto que nos permite seleccionar individuos en las primeras etapas de desarrollo, así como la eliminación anticipada de genotipos indeseables. Dicha técnica se emplea con varios propósitos, como retrocruzamiento asistido, mapeo de QTL y para identificar individuos con un mayor nivel de homocigosidad en poblaciones segregantes (Oliveira et al., 2018). La SAM ha sido utilizada ampliamente para caracteres de herencia simple y en una menor proporción para caracteres poligénicos, aunque ya existen algunos casos exitosos en la mejora de caracteres cuantitativos empleando estas técnicas (Alvarez, 2011).
En cuatro variedades diferentes de Avena sativa “avena”, se utilizaron 42 marcadores en condiciones de invernadero, estas variedades se cultivaron separándolas en 8 recipientes de plástico y se sometieron en una temperatura de 15-17°C y 16 horas de luz diarias. Asimismo, los marcadores moleculares ayudaron a determinar las diferencias entre el patógeno “roya de tallo” y las variedades de avena; demostrando que en la variedad Karma se visualiza los marcadores que poseen resistencia de Obsidiana y Cevamex (Torres, et al., 2007).
En el cultivo de Solanum tuberosum “papa”, se utilizó el marcador M45 para determinar la resistencia al virus PVY. La metodología que se utilizó para dicha reacción se basa en ciertas condiciones, tales como 94°C por 60 segundos seguidos de 30 ciclos de desnaturalización a 94°C por 20 segundos, con una temperatura de hibridación 61°C por 20 segundos y extensión a 72°C por 20 segundos seguidos de una extensión final a 72°C durante 20 min en un termociclador Genius Techne. Se obtuvo que el M45 es eficiente para evitar la presencia de Ryadg, debido a que es una herramienta necesaria para la selección asistida en marcadores. Por lo que facilita su elección a ser incluido en el desarrollo de variedades resistentes al PVY (virus Y de la papa) (Torres, 2004). Por otro lado, en un estudio en el cultivo de “papa”, se usó cinco marcadores moleculares que amplificaron la banda en más del 80 % de las poblaciones con genes de resistencia a ciertos patógenos como lo son el virus PVY y PVX; el nematodo G. pallida y G. rostochiensis, el tizón P. infestans y la verruga S. endobioticum (Gabriel, et al., 2016).
En el proyecto experimental del Cultivo de Capsicum annuum “pimiento” para conocer la resistencia al fitopatógeno Phytophthora capsici, se utilizaron los marcadores moleculares como son SCAR OpD04.717, PR-1 y EAS, que presentan resistencia a pudrición de raíz. Asimismo, las condiciones óptimas de laboratorio, para los marcadores fueron 2-4 minutos aproximadamente de desnaturalización, 30 ciclos de 30 s. a 94°C, un alineamiento de 30 s entre 42 °C a 58 °C y se utilizó un termociclador T100TM marca BioRad. Además, en el primer marcador molecular SCAR OpD04. 717, no se identificó resistencia al patógeno en 22 accesiones de pimiento, en el segundo marcador molecular PR-1 se identificó resistencia al patógeno en 20 acciones de pimiento y en el tercer marcador molecular EAS se identificó resistencia en 21 entradas/accesos de pimiento. Por lo tanto, ninguno de estos marcadores logró identificar nuevos materiales de resistencia al patógeno en el cultivo de pimiento, es por ello que no son integrados en programas de mejoramiento genético (García, 2019). Sin embargo, en otro estudio se obtuvo que el marcador SCAR OpD04.717 no permitió la discriminación entre accesiones resistentes y susceptibles a P. capsici, ya que tampoco se identificaron 11 accesiones de pimiento con resistencia al hongo; es por esto que existe utilidad potencial como nuevas fuentes de resistencia a pudriciones de la raíz causada por P. capsici para programas de mejoramiento genético tradicional, pero no para selección asistida por marcadores moleculares mediante OpD04.717 (Mendéz, et al., 2015).
Inducción de mutaciones
En las plantas autógamas, la heterocigosis es un recurso extremadamente importante al comienzo de la mejora, ya que generalmente se asocia con la presencia de variabilidad genética. Mutación e hibridación inducidas puede aumentar la proporción de loci heterocigotos de forma distinta. En especies diploides, después de algunas generaciones de autofecundación, la heterosis reducirse a la mitad debido a la endogamia. Se pueden utilizar varios mecanismos para crear y expandir los loci en heterocigosis, como mutaciones inducidas e hibridaciones artificiales (Rita et al. 2017).
En una investigación realizada en el valle Jequetepeque, Región La Libertad, Perú, se buscó mejorar el rendimiento de Oryza sativa “arroz” induciendo mutaciones mediante la aplicación de rayos gamma. Se utilizó 250 gr. de semilla, se les aplicó dosis de 150, 250 y 350 Gy de rayos gamma; se tomó las mejores ocho líneas mutantes y se pasó a la siembra con suelo batido, fertilización con N/Ha en Urea y el método de riego fue por inundación. Como resultado se obtuvo que todas las líneas mutantes presentaron un acortamiento de 15 días en el tiempo de maduración, por otro lado, solo dos de ellas (líneas MC 25 – 20 – 1 y MC 35 – 45 – 4) reflejaron los mejores rendimientos pese a no superar a su calidad molinera. Sin embargo, este avance permite continuar investigando la utilización de radiaciones como posible fuente de inducción de mutaciones favorables en arroz (Rodríguez, 2017).
La técnica de inducción de mutaciones también se aplicó en un trabajo de investigación realizado en Huancavelica, Perú, donde se buscó mejorar la estructura genética de Triticum aestivum “trigo” utilizando el agente mutagénico químico azida de sodio. Utilizaron 400 semillas de trigo humedeciéndolas en dosis de 0,0 mM, 0,5 mM, 1,0 mM, 1,5 mM, 2,0 mM, 2,5 mM, 3,0 mM y 3,5 mM de azida de sodio durante una hora, cada dosis se consideró un tratamiento. Se sembraron en vasos utilizando el método de Aruba con vermiculita. Los resultados obtenidos indican que se obtuvo el mayor resultado con en una dosis de 0.5 mM, como por ejemplo aumentando el diámetro del tallo, longitud de raíces, altura de la planta y otras cualidades (Benito, 2019).
Cultivo de Tejidos
Cultivo de tejidos in vitro ha demostrado que es un método muy favorable ya que las técnicas que se aplican son más fáciles y se necesita menos presupuesto en la producción, sobre todo ayuda a disminuir el tiempo que se necesitaría para obtener nuevas plantas comparado con el requerido en campo. Otras de las ventajas son que disminuye considerablemente la probabilidad de contraer patógenos en las plantas en comparación con otros métodos, y sus tasas de multiplicación son altas. Algo que no favorece al aplicar el método de tejidos cultivados in vitro es que el material genético se puede perder producto de las contaminaciones y también es necesario que el personal calificado esté vigilando todo el tiempo (Bonilla et al., 2015).
Como ejemplo, se menciona la regeneración en cultivo in vitro de plantas de tomate (Solanum lycopersicum L.), mediante el cual se evalúa la respuesta organogénica de los explantes. En este proceso de regeneración en cultivo in vitro se usaron tres variedades de tomate (Money-Maker, Micro-Tom y Tomate de colgar), así también se hizo uso de cinco tipos de explantes, los cuales fueron obtenidos de plántulas axénicas (Maestu, 2018).
Durante este proceso se realizó la siembra de explantes obtenidos de las plantas axénicas, se incubó durante 30 días de en placa para luego evaluar la respuesta organogénica en un medio de cultivo con los constituyentes y reguladores de crecimiento adecuados. Finalmente, se logró definir las fases que ocurren durante el proceso de organogénesis, dicho proceso se completó exitosamente, siendo este evaluado por el desarrollo desorganizado de los callos, las yemas adventicias y la aparición de brotes, estos resultados nos permiten confirmar que es posible regenerar plantas mediante esta metodología. Las ventajas que este procedimiento incluyen: conseguir plantas que estén libre de patógenos lo cual serviría para saneamiento clonal y posteriormente producir semillas certificadas. Los resultados obtenidos para las variedades utilizadas son normales y con un porcentaje muy bajo de contaminación (menor al 5%), logrando la organogénesis en valores mayores al 90% de explantes, resultados bastante promisorios para seguir realizando investigaciones con el fin de estandarizar el protocolo para obtener una producción masiva. La figura 5 nos muestra el desarrollo de un explante de la variedad Money-maker donde podemos observar la presencia de callos y su desarrollo (Maestu, 2018).

Figura 5. Seguimiento del explante de Money Maker de un cotiledón
Nota: Fases del seguimiento del explante de un cotiledón del genotipo Money Maker A) Inicio del cultivo, B) Tras una semana de cultivo, C) Tras dos semanas de cultivo, D) Tras tres semanas de cultivo, E) Tras cuatro semanas de cultivo. Adaptado de Regeneración de plantas en cultivo in vitro de tomate (Solanum lycopersicum L.) por Maestu (2018).
Otra técnica muy utilizada es la de micropropagación, en este caso se estudió la germinación y micropropagación de tomate de cáscara (Physalis ixocarpa), para este procedimiento se realizaron tratamientos de imbibición haciendo uso de reguladores y ácidos orgánicos en las semillas a usar, los reguladores usados fueron: ácido giiberélico (AG), los ácidos orgánicos: benzoico (AB), salicílico (AS), sulfosalicílico (ASS), con unas concentraciones de 10-2M, 10-4M y 10-6M y para la micropropagación se usaron medios MS y PCL2 adicionada con 100 mg L-1 de mio-inositol, así también se usaron explantes como: hipocótilos, cotiledones y segmentos nodales. Con respecto al uso del ácido Giberélico, este generó una rápida síntesis de amidas y aminoácidos logrando incrementar la germinación, así también se encontró relacionado al aumento de fenoles en las semillas lo cual se relaciona con el proceso morfogenético del embrión. Por otro lado, el tratamiento de ácidos orgánicos con ASS presentaron un mayor incremento en germinación, siendo estos derivados de los fenilpropanoides funcionando como una defensa para las plantas. Gracias a estos resultados se concluyó que el ácido Giberélico y los ácidos orgánicos dieron una respuesta positiva con un incremento en la germinación de las plántulas en menor cantidad de tiempo. En la Figura 6, podemos observar los resultados del estudio, se puede observar el desarrollo de plántulas in vitro inclusive hasta alcanzar el estado fenológico de floración (García, 2015).

Figura 6. Floración in vitro de tomatillo tetraploide (Physalis ixocarpa) y explantes nodales.
Nota: En la siguiente imagen logramos observar los explantes nodales en los cultivos in vitro y la floración de tomatillo tetraploide (Physalis ixocarpa ) dando a conocer la eficacia de los pretratamientos en las semillas, debido a que esta incrementa la tasa de germinación. Adaptado de Germinación y micropropagación de tomate de cáscara (Physalis ixocarpa) tetraploide por García (2015).
Ingeniería genética y mejoramiento de genes
Actualmente, el uso generalizado de genes es una prioridad en la protección de los recursos genéticos para la agricultura sostenible. Asimismo, se han utilizado técnicas de cultivo con la aplicación de biotecnología basada en la ingeniería genética y la adquisición de plantas transgénicas. Con el auge de la nueva biotecnología, se han realizado esfuerzos para distinguir entre la proliferación de cultivos convencionales y la proliferación de plantas no convencionales. Por lo tanto, no es exagerado decir que el fitomejoramiento pronto será reemplazado por nueva biotecnología. La biotecnología, junto con la genética, la fisiología y la biometría, son otras herramientas que pueden ser de gran ayuda en la producción de nuevas variedades. Los fitomejoradores, los fisiólogos y los fitopatólogos deben fortalecer los grupos de trabajo interdisciplinarios y complementarse para abordar cuestiones de interés común (Vallejo & Estrada, 2016).
El fitomejoramiento convencional es muy útil cuando se trata de mejora de cultivos, pero principalmente implica cultivar y probar muchos cultivos durante varias generaciones, una tarea larga y tediosa. La ingeniería genética, que significa un cambio directo en la composición genética, tiene varias ventajas sobre la producción convencional (Dong & Ronald, 2019).
La mayoría de las plantas modificadas genéticamente se basan en especies con enfoques transgénicos convencionales o, más recientemente, en tecnología de edición del genoma. En los diseños transgénicos, la codificación de genes requiere la presencia de caracteres del genoma de interés agronómico de diferentes sitios por mutación (Dong & Ronald, 2019). Estas actividades a menudo conducen a la integración de un ADN foráneo. Por el contrario, la modificación del genoma permite mutaciones en plantas de ADN endógeno, como la eliminación, liberación y modificación del ADN, para una variedad de propósitos a largo plazo (Barrangou & Doudna, 2016).
Cambiar la vulnerabilidad de las estrategias de la virulencia de patógenos tiene un gran potencial para la protección de los cultivos. Sin embargo, puede crear resistencia (McGrann et al., 2014), es importante para la evaluación de los efectos secundarios en la susceptibilidad de los genes. Debe verse si la resistencia se encuentra en las condiciones descritas en estudios de laboratorio robustos o en buenos productos en condiciones de campo o si los productos son buenos en el campo.
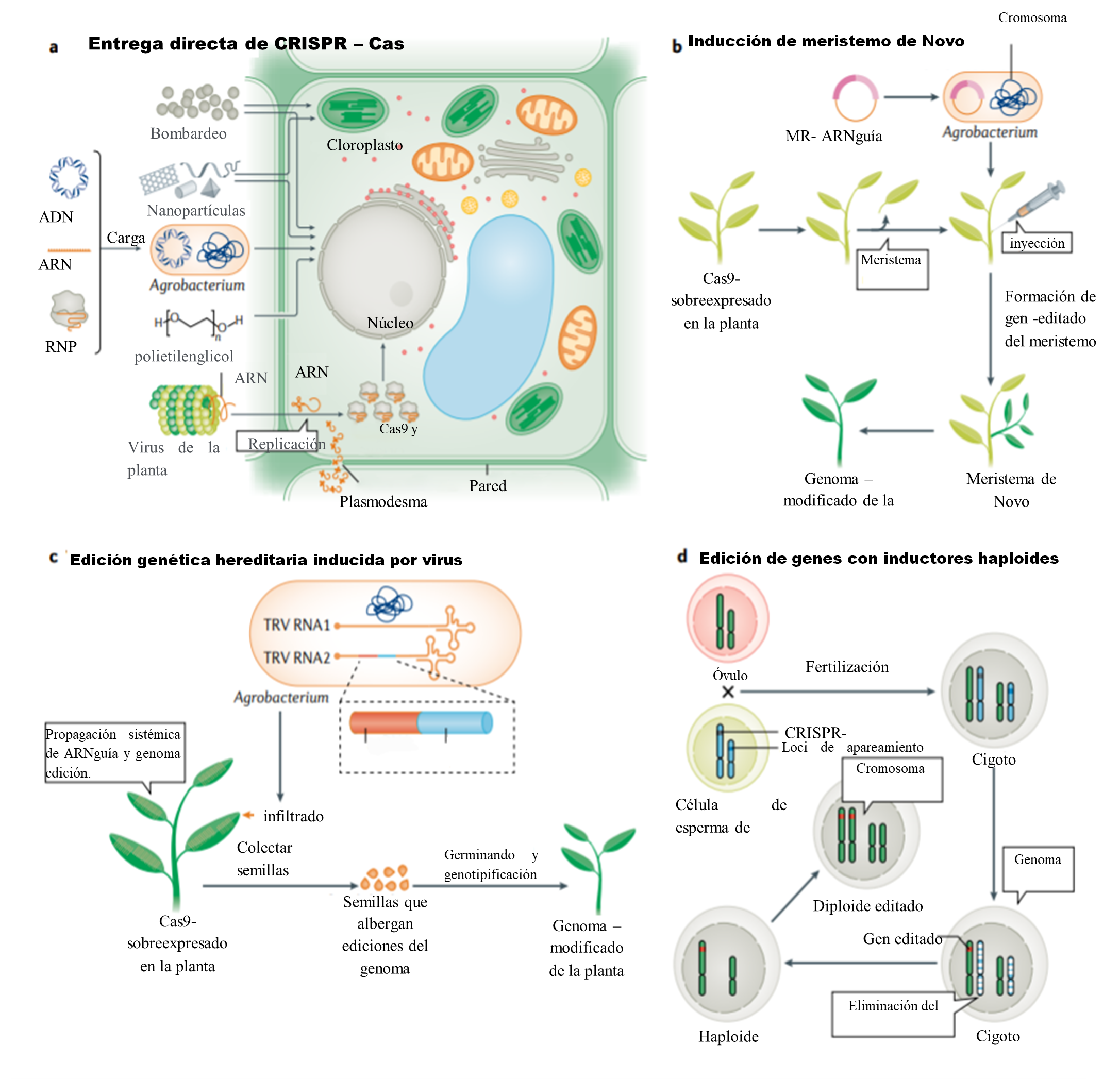
Figura 7. Estrategias para la entrega de CRISPR-Cas a las plantas.
Nota: Adaptado de Zhu et al. (2020) Applications of CRISPR - Cas in agriculture and plant biotechnology. Nature Reviews Molecular Cell Biology.
Edición Génica
La modificación de genes es posible gracias a la creación de dominios ligados al ADN funcionales y eficientes que permiten a los científicos transferir el ADN modificado. Normalmente, muchos métodos de modificación del genoma requieren el fracaso de las mediciones bidimensionales (DSB) en la secuenciación del ADN. Antes de Cas9, los dominios de unión a ADN basados en proteínas (p. Ej., Dedo de zinc o TALE’s) combinados con el dominio de nucleasa para formar nucleasas específicas de secuencia (SSN) podían producir DSB estructurados. Cas9 tiene funciones similares y su interacción con el ADN está controlada por un entorno ARN-ADN, lo que facilita el proceso de numeración SSN. Además, el Cas 9 se puede convertir en nicasas o simplemente ADN superpuesto formando uno o dos nuevos modelos estructurales (Atkins at el., 2020).
Los reactivos de edición del genoma generalmente se transfieren a las células vegetales de dos formas: Bacterias capaces de transferir ADN directamente a las plantas (como Agrobacterium tumefaciens), o por balística bombardeando partículas de oro. La susceptibilidad celular a las agrobacterias es un rasgo complejo que resulta en eficiencias de transmisión muy variables entre el ecotipo y la especie. El segundo enfoque es el bombardeo Biolística, que habilita cualquier cosa que pueda unirse a partículas de oro (generalmente ADN, ARN, proteínas o cualquier combinación). Sin embargo, la eficiencia es baja. Aunque ambos métodos son adecuados para pruebas a corto plazo (genéticas o de otro tipo), la transmisión preferida de partículas virales provoca la necesidad de otras medidas para producir virus que sean completamente superiores al original. Además de aumentar la eficiencia agrícola, han surgido recientemente varias biotecnologías relacionadas con CRISPR-CAS. Por ejemplo, en ausencia de un sistema de transmisión robusto, se utiliza para clasificar genes en plantas, así como una serie de dispositivos innovadores que se están desarrollando para producir plantas modificadas genéticamente que permiten el uso de un semi genoma complejo o la reproducción del ADN. Por ello, CRISPR-Cas se utiliza para estudiar la regulación de la expresión génica en varios campos. Además, CRISPR-Cas, debido a su facilidad de uso y fuertes propiedades ortogonales, está adaptado para la edición de genomas multifuncionales y productivos y diversos sustratos en biología de plantas artificiales. En la figura 7, podemos apreciar las principales estrategias que se tiene para entregar los sistemas CRISPR-Cas en plantas para la edición genética precisa (Zhu et al., 2020).
CASOS DE MEJORAMIENTO EN PLANTAS AUTÓGAMAS
Mejoramiento genético en la quinoa
La especie Chenopodium quinoa Willd “quinua”, es una planta con elevada nutrición y gran capacidad de adaptación, ya que puede crecer en diferentes tipos de agroecosistemas, esta especie presenta tolerancia a factores abióticos y bióticos. La quinoa genera atención en varios países debido a sus características nutritivas que se encuentra en sus semillas, ya que contiene gluten en bajas cantidades y existe un excelente equilibrio en sus elementos esenciales (Nobuyuki et al., 2017). Este cultivo procedente de los Andes, es altamente tolerante a condiciones extremos de estrés, sequía, temperaturas elevadas o bajas, a salinidad elevada y a bajos nutrientes en diferentes tipos de suelos, especialmente los arenosos (Gómez et al., 2017). Esta planta es ampliamente adaptativa, pero algunas variedades no lo son; esto nos lleva a examinar diferentes variedades que presenten adaptación a diferentes zonas agroecológicas que se encuentran en el Perú. También se sabe que la producción de esta especie abarca 61,490 toneladas por año, y a la vez presentan variedad de genotipos tolerantes a diversos factores como eventos naturales extremos (sequías y heladas). Esta especie también presenta una mejora en alimentos (calidad) y rendimiento. Sin embargo, los caracteres observables (fenotipos) que presenta son fácil de distinguir, es por lo que actualmente se siembran diversas especies comerciales. Por lo tanto, se tiene que mejorar a nivel genotípico la calidad que presentan los granos, su rendimiento y las tolerancias a distintos factores (Bedoya et al., 2018). Asimismo, la biotecnología moderna, más específicamente los marcadores moleculares de ADN, permiten una caracterización adecuada de los genotipos del florecimiento en la variedad de especies que serán mejoradas. También se encontrarán alimentos de calidad disponibles y presentarán tolerancia a diferentes factores. Sin embargo, para poder identificar los genotipos de la quinoa, se utilizan los marcadores moleculares (SSR), basados en repeticiones de secuencia simple, ya que son esenciales para la investigación de la variación génica y ayuda a comprender mejor su dinámica. Se encuentran 26 variedades de esta especie donde los iniciadores informativos como QCA053, QCA015 y QCA034, son fáciles en su lectura e interpretación, ya que son polimórficos y discriminantes (Romero et al., 2019).
La planta de “quinoa” presenta un porcentaje de alogamia natural, donde esta varía de 0.5 % a 17%, ya que sus líneas puras se perfeccionaron y estas a su vez fueron elegidas por sus características de adaptación y rendimiento (Mellado, 2014). Por lo tanto, en esta especie se ha realizado mejoramiento en los siguientes caracteres fenotípicos como son la altura, color y semilla de la planta, también el grano (volumen) y la tolerancia a enfermedades. Además, la respuesta y ganancia de las mismas serán directamente proporcionales; ya que esta selección, permite que la selección natural actúe en caracteres heredables, estos caracteres son favorecidos en ambientes donde los genotipos mejorados puedan destacar; se sabe que actualmente existen diversas variedades de especies obtenidas por la llamada “selección masal” en diversos países como por ejemplo en Colombia que se tiene a la Dulce de Quitopamba y en Bolivia que tienen a Real. Asimismo, Perú presenta las variedades Blanca de Junín, Blanca de Juli, Amarilla de Marangani, Rosada de Junín y Blanca de Hualhuas (Gómez, 2015).
Análisis RT-qPCR de genes expresados desigualmente en quinua
Se realizó una investigación de la expresión mediante la extensión qPCR, se observó que una vez representado inmediatamente por medio de la secuencia codificante completa (CDS) frente a la secuencia genómica de quinua, observamos que un 90% presentaba una armadura como la información genética (e < 1e -10; puntuación > = 500; cobertura > = 50%) (Yasui et al., 2016).
Este análisis permitió identificar a los genes asociados a condiciones de sequía así como elaborar un posible mecanismo metabólico por el cual se pueda explicar dicha resistencia, en la cual se elaboró una reconstrucción de una ruta metabólica, que se presenta por estrés a causa de la sequía, esta vendría a ser la ruta ácido abscísico (ABA), este mecanismo involucra cinco genes, los cuales, tresde ellos están ubicados en plastoides: NCED3, ABA1 y ABA4 y 2 se encuentran en el citogel: ABA2 y ABA3. Se agregaron 2 genes del camino de la biosíntesis ABA, son quienes intervienen con el transporte: ABCG25, ABCG40 y ABA, también hay algunos implicados mediante una reacción ante el estrés, los cuales se eligieron a partir de las variaciones encontradas a través de RNA-Seq en la quinua: CqCAP160, CqHSP 20, CqLEA, CqAP2/ERF, CqPP2 C, CqP5 CS y CqHSP83 (Raney et al., 2014).
La finalidad de este análisis de expresión mediante qPCR de 15 unigenes elegidos, es discutir las variaciones del ARN-Seq que nos permite identificar cambios en los transcritos y expresión de genes de manera experimental con los modelos teóricos de análisis in silico, el cuál demostró que es un modelo semejante al de la expresión en el modelo computacional (Yasui et al., 2016).
Mejoramiento genético en Frijol
Phaseolus vulgaris, se le considera de suma importancia porque el 50% de este grano, es consumido a nivel mundial. Su producción se basa en distintos ambientes, haciendo que el frijol sea considerado una especie universal. Pero, se conoce que un 60% de los sembríos de Phaseolus vulgaris, han sido cultivados con una amenaza de sequía entrecortada y final (Mayor, 2015).
De la mención anterior, dicha sequía es quien ocasiona que haya una baja productividad en el campo, como medida se ha tenido que ejecutar diversas técnicas para afrontar las consecuencias de la sequía, dos de las secuelas son el riego adicional y realizar prácticas agronómicas. A pesar de adaptar esa medida, no ha servido de mucho debido a los costos de implementar estas opciones. De esa manera se hace necesario el establecimiento de planes de mejoramiento genético que hagan que la producción del cultivo sea factible y sostenible (Mayor, 2015).
Cuando se utiliza el método de mejoramiento genealógico nos ofrece tener una visión total de una probable técnica combinatoria específica y total de cada uno de los genotipos, incluso luego de haber pasado varias generaciones, de esta manera se logrará determinar buenos progenitores en los futuros procesos de mejoramiento, trabajándose principalmente en mejoramiento de rendimiento en condiciones de sequía (Chaves et al., 2018).
También se viene trabajando en mejoramiento para solucionar problemas de enfermedades del cultivo como el ataque causado por el Virus del Mosaico Dorado Amarillo del Frijol (BGYMV) viéndose resaltado el uso de variedades resistentes, estas se logran a través de la introducción de genes que son dominantes de hipersensibilidad I y genes recesivos de resistencia previamente combinados, introduciéndolo en diversos tipos de frijol que son susceptibles al BGYMV (López et al., 2018).
Mejoramiento genético mediante Agrobacterium tumefaciens en Phaseolus vulgaris “frijol”
El frijol común Phaseolus vulgaris, es uno de los alimentos indispensables para contribuir en la nutrición humana (Broughton et al., 2003; Beaver y Osorno, 2009). Por lo cual el propósito de este trabajo fue desarrollar un protocolo de transformación genética mediado por Agrobacterium tumefaciens en Phaseolus vulgaris “frijol” debido a que la certeza para el mecanismo de integración, la inclinación para realizar inserciones limpias y únicas (sin ninguna inserción de ADN no deseado), además de una disminución en el costo operacional van a constituir una ventaja considerable para los mecanismos de transferencia directa de genes con el método de la biobalística (Broughton et al., 2003). Como objetivo principal se proyectó una adaptación del frijol común (Phaseolus vulgaris L.) en un protocolo de transformación genética mediado por Agrobacterium tumefaciens , a partir de una metodología eficiente en soya.
En esta técnica se utilizó la cepa de Agrobacterium tumefaciens EHA105 que portaba el plásmido pCAMBIA 1305.2. Este plásmido le otorgará a dicha bacteria una resistencia contra el antibiótico kanamicina y su T-DNA contendrá un gen reportero β-Glucuronidasa (GUSPlusTM) que será detenido por un intrón y con un promotor 35S. Como tanteador de selección para las plantas va a portar un gen que tendrá una resistencia contra el antibiótico higromicina (HPTII) con doble promotor o causante 35S (Figura 8) (Barrera, 2018).
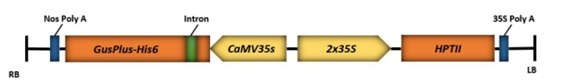
Figura 8. Fragmento del plasmido p CAMBIA 1305.2
Nota: De izquierda a derecha: RB, borde derecho del T-DNA. CaMV35s, impulsor 35s del virus del mosaico controlando el gen GusPlus con finalizador Nos. HPTII, gen resistente a higromicina con doble promotor 35S (2x35S) y terminador 35S. LB, borde izquierdo del T-DNA. Tomado de transformación genética mediada por Agrobacterium tumefaciens en frijol común (Phaseolus vulgaris L.) por Barrera (2018).
Mejoramiento genético del arroz
El grano de arroz no tiene precursores de b-caroteno (provitamina A) en el endospermo. Por ejemplo, millones de niños pobres de las zonas productoras de arroz de Asia padecen de deficiencia de vitamina A, se logró conferir al arroz la capacidad de producir el precursor de la vitamina A (b-caroteno) mediante la transformación genética de tres genes (fitoeno sintasa, fitoeno desaturasa y licopeno ciclasa) en la variedad de arroz Taipei 309 y luego se transfirió al cultivo de arroz IR64. Sin embargo, el productor de oro rosa del IRRI aún se encuentra en la fase de prueba (Hefferon, 2015).
Otros genes de deficiencia nutricional son el hierro y el zinc, que no están ampliamente disponibles en los granos de arroz. Se han aplicado enfoques de producción transgénicos y convencionales. Recientemente, el gen de la ferritina de soja (Sferit-1) para altos niveles de hierro (Fe) y el gen NAS para zinc (Zn) se transfirieron al cultivo de arroz IR64 por mutación. Por lo tanto, el contenido de Fe y Zn en las semillas de arroz transgénico en un campo inundado aumentó a 15 μg-1 y 45 μg-1, respectivamente, en comparación con los granos de arroz ordinarios (Trijatmiko et al., 2016).
Otros rasgos relacionados con la mejora del potencial de rendimiento del arroz son el número de granos por panícula, el tamaño y ramificación de las panículas, el tamaño del grano, los días al encabezado, peso de mil granos y resistencia al encamado. Estos rasgos no son controlados por un solo factor genético, sino que están controlados por un gran número de genes de pequeño efecto que surgen de rasgos de componentes coordinados y genes subyacentes predisposiciones (Jena & Nissil, 2017).
Estos rasgos que mejoran el rendimiento están controlados por loci de rasgos cuantitativos (QTL). A través del mapeo fino de QTL, se han identificado varios genes que mejoran el rendimiento en el arroz (Liu et al., 2015).
El gen de resistencia al alojamiento, SCM2 / APO1, que codifica la proteína que contiene la caja F, controla el número de granos por panícula y el diámetro del culmo. Una transcripción abundante del gen SCM2 que causa tallos fuertes y aumenta su expresión en panículas en desarrollo (Zhang et al., 2016). Estas líneas expresaron los genes que mejoran el rendimiento en diferentes antecedentes genéticos que se desarrollaron a través de la selección asistida por marcadores (SAM) y antecedentes de análisis utilizando chips 6 K Infinium SNP (Kim et al., 2016).
Se realizó un estudio basado en la técnica CRISPR / Cas9 del gen OsGA20ox2 en un cultivo de arroz índica, originalmente una variante en la secuencia de aminoácidos en plantas mutantes y esto ocasionaba la disminución de giberelinas y que aumente el rendimiento de cada planta. Como resultado de este estudio se obtuvo que la edición del gen OsGA20ox2 mostraba alteración significativa en la expresión de algunas proteínas, entre ellas la gliceraldehído-3-fosfato deshidrogenasa, ATP sintasa putativa, fructosa-bisfosfato aldolasa 1, S-adenosil metionina sintetasa 1 y giberelina 20 oxidasa 2, afectando al crecimiento de la planta. Los resultados de la investigación se observan en la figura 9 (Han et al., 2019).

Figura 9. Fenotipo de plántula de plantas JGR silvestres y mutantes en condiciones controladas y tratamiento con GA3, A plantas en condiciones controladas, B plantas tratadas con 10 μM GA3.
Nota: Adaptado de Han et al. (2019).
3. Conclusiones
Las plantas autógamas, llamadas así porque su reproducción es por autofecundación, es decir que ambos gametos provienen de la misma planta, las plantas autógamas son consideradas como una línea pura de homocigotos para todos sus caracteres. A nivel mundial se les considera de gran importancia a estas plantas por su gran demanda para el consumo alimentario de las personas y por su gran contenido nutricional, en América Latina se encuentra una de las principales especies de plantas autógamas como Phaseolus vulgaris “frijol ñuña” que contiene altos porcentajes en carbohidratos y proteínas, otra especie de gran importancia es Lycopersicon esculentum que genera también una gran demanda comercial, entre otras especies de igual importancia se tiene a Solanum tuberosum, Oryza sativa, Lupinus mutabilis y Triticum aestivum.
Se determinó que existen muchos métodos de mejoramiento en plantas autógamas ya sea por selección, hibridaciones, inducción de mutaciones, cultivo de tejidos, la ingeniería genética y muchos más.
La obtención de variedades mejoradas es un desafío, es por este motivo que se describen diferentes métodos. Para lograr comprender los resultados de la selección es necesario llegar a conocer la estructura genética de las poblaciones.
La biotecnología complementa y no reemplaza muchas áreas de la investigación agrícola tradicional. Para una mejora de los recursos genéticos en la alimentación y en la agricultura, ofrece variedad de herramientas. Estas nos ayudan en programas diversos de recuperación y protección facilitando el diagnóstico, tratamiento y prevención de enfermedades de plantas. La biotecnología también puede aplicarse en conexión con investigadores de nuevos materiales y herramientas que aumentan la eficiencia del trabajo. Por tanto, se puede considerar que los programas de inversión basados en biotecnología amplían la precisión de los métodos convencionales. Asimismo, la ingeniería genética es posiblemente el concepto más importante de tecnología de coexistencia, ya que permite a los científicos integrar material genético entre organismos distintos.
4. Contribución de los autores
J. P., W.B.: Concepción, diseño, revisión crítica del artículo y aprobación de la versión final. J.P., D.R., L.R., S.R., J.R., F.S., M.S., L.V., J.V., E.V., W.V. y Y.Y.: Recopilación de información, organización de la información, resumen de la información, redacción del resumen, elaboración de las conclusiones, revisión del documento durante todo el proceso de edición y consignación de citas y referencias bibliográficas. Todos los autores han leído el manuscrito final y aprobado la versión.
5. Conflicto de intereses
Los autores declaran no tener conflictos de intereses.
6. Referencias bibliográficas
Álvarez, M. (2011). La selección asistida por marcadores (MAS, “Marker-assisted selection”) en el mejoramiento genético del tomate (Solanum lycopersicum L.). Cultivos tropicales, 32(2), 154-171.
Angulo, I. y Ortiz, M. (2020). Mejoramiento Genético en Plantas Alógamas y Autógamas. [Monografía de Licenciatura, Universidad Nacional de Colombia].
Arruabarrena, A., Gonzales, M., Rubio, L. y Giménez, G. (2015). Selección asistida por marcadores en el mejoramiento genético de tomate. Biotecnología para el sector productivo. Revista INIA Uruguay, 40(1), 43 - 46.
Arteaga, A. (2011). Simulación de dos estrategias de mejoramiento genético de plantas aplicando la plataforma qu- gene. [Tesis de maestría, Universidad Nacional de Colombia].
Atkins, P. A., & Voytas, D. F. (2020). Overcoming bottlenecks in plant gene editing. Current Opinion in Plant Biology, 54(1), 79–84.
Barrangou, R. y Doudna, J. (2016). Applications of CRISPR technologies in research and beyond. Nat Biotechnol, 34(9), 933-941.
Beaver, J. S., & Osorno, J. M. (2009). Achievements and limitations of contemporary common bean breeding using conventional and molecular approaches. Euphytica, 168(2), 145–175.
Barrera Moreno, J. J. (2018). TRANSFORMACIÓN GENÉTICA MEDIADA POR Agrobacterium tumefaciens EN FRIJOL COMÚN (Phaseolus vulgaris L.). Universidad de los Llanos, Colombia.
Bedoya, N., Puni, G., Mujica, A., Talamini, E. y Domingos, A. (2018). Quinoa Expansion in Peru and Its Implications for Land Use Management. Sustainability 10(2), 532.
Benito, M. (2019). Modificación de la estructura genética del trigo (Triticum aestivum L) por inducción mutagénico con azida de sodio. [Tesis de grado, Universidad Nacional De Huancavelica].
Bonilla, M. (2015). Conservación in vitro: una perspectiva para el manejo de los recursos fitogenéticos. Revista De Investigación Agraria Y Ambiental, 6(1), 67 - 82.
Broughton, W. J., Hernández, G., Blair, M., Beebe, S., Gepts, P., & Vanderleyden, J. (2003). Beans (Phaseolus spp.) – model food legumes. Plant and Soil, 252(1), 55–128.
Camarena, F., Chura, J. y Blas, R. (2014). Mejoramiento Genético y Biotecnológico de Plantas. Universidad Nacional Agraria de La Molina (UNALM). Lima -Perú.
Chaves, N., Polanía, J., Muñoz, C., Rao, I. y Beebe, S. (2018). Caracterización fenotípica por resistencia a sequía terminal de germoplasma de frijol común. Agronomía Mesoamericana, 29(1), 1-17.
Chen, J., Luo, M., Li, S., Tao, M., Ye, X., Duan, W., Zhang, C., Qin, Q., Xiao, J., & Liu, S. (2017). A comparative study of distant hybridization in plants and animals. Science China Life Sciences, 61(3), 285–309.
Cortes, D., Santa, R., Vettorazzi, J., Ramos, H., Pio, A. y Pereira, M. (2019). Development of superior lines of papaya from the Formosa group using the pedigree method and REML/Blup procedure. Bragantia, 78(3), 350-360.
Cubero, J. (2003). Métodos de mejora de autógamas mediante selección con cruzamiento. Mundi-Prensa.
Cuya, C. (2018). Selección de accesiones promisorias de tomate silvestre (Lycopersicon sp.). [Tesis de grado, Universidad Nacional Agraria La Molina].
Dong, O. y Ronald, P. (2019). Genetic Engineering for Disease Resistance in Plants: Recent Progress and Future Perspectives. Plant physiology, 180(1), 26-38.
FAO, FIDA y PMA. (2013). El estado de la inseguridad alimentaria en el mundo 2013. Las múltiples dimensiones de la seguridad alimentaria. Roma, FAO.
Gabriel, J., Veramendi, S., Pinto, L., Pariente, L., y Angulo, A. (2016). Asociaciones de marcadores moleculares con la resistencia a enfermedades, caracteres morfológicos y agronómicos en familias diploides de papa (Solanum tuberosum L.). Revista Colombiana de Biotecnología, 18(1),17-32.
García, H. (2015). Germinación y micropropagación de tomate de cáscara (Physalis ixocarpa) tetraploide. Revista Mexicana de Ciencias Agrícolas, (12), 2301-2311.
García, J. (2019). Uso de marcadores moleculares de ADN asociados con la resistencia a (Phytophthora capsici) en el cultivo del chile. Division de Ciencias de la Salud, Biológicas y Ambientales.
Gómez, A., López, C., Cantos, M., García, L., Molina, R., Cotrino, J., Espinós, J. y González, A. (2017). Surface chemistry and germination improvement of Quinoa seeds subjected to plasma activation. Scientific Reports, 7(5924), 1-12.
Gómez, L. (2015). Quinoa Breeding. En K. S. Murphy y J. Matanguihan (Eds.) Quinoa: Sustainnable production, variety improvement, and nutritive value in agroecological systems. (pp. 87-109). Wiley-Blackwell.
Han, Y., Teng, K., Nawaz, G., Feng, X., Usman, B., Wang, X., Luo, L., Zhao, N., Liu, Y., & Li, R. (2019). Generation of semi-dwarf rice (Oryza sativa L.) lines by CRISPR/Cas9-directed mutagenesis of OsGA20ox2 and proteomic analysis of unveiled changes caused by mutations. 3 Biotech, 9(11), 387.
Hefferon, K. (2015) Nutritionally enhanced food crops: progress and prospectives. International Journal of Molecular Sciences 16(2), 3895-391.
Hualla, V. (2017). Ganancias genéticas en el contenido de hierro y zinc en papas diploides en tres ciclos de selección recurrente. [Tesis de posgrado, Universidad Nacional Agraria La Molina].
Iris, A. (2018). Lupinus mutabilis (tarwi). Leguminosa andina con gran potencial industrial. Fondo Editorial de la Universidad Nacional Mayor de San Marcos.
Jena, K. y Nissil, E. (2017). Genetic Improvement of Rice (Oryza sativa L.) en H. Campos y D.S. Caligari (Eds.). Genetic Improvement of Tropical Crops (pp. 111 – 127). Springer, Cham.
Kim, S., Ramos, J., Ashikari, M., Virk, P., Torres, E., Nissila, E., Hechanova, S., Mauleon, R. y Jena, K. (2016) Development and validation of allele-specific SNP/indel markers for eight yield-enhancing genes using whole-genome sequencing strategy to increase yield potential of rice, Oryza sativa L. Rice, 9(12), 1 – 17.
Lea, A. (2017). Marker assisted backcross selection for virus resistance in pea (Pisum sativum L.). [Master's Thesis, North Dakota State University].
Liu, S. (2010). Distant hybridization leads to different ploidy fishes. Science China Life Sciences, 53(4), 416–425.
Liu, D., Zhang, H., Zhang, L., Yuan, Z., Ming, H. & Zheng, Y. (2014). Distant Hybridization: A Tool for Interspecific Manipulation of Chromosomes en A., Pratap & J., Kumar (Eds.), Alien Gene Transfer in Crop Plants, Volume 1: Innovations, Methods and Risk Assessment (pp. 25-42). Springer Science+Business Media.
Liu, L., Tong, H., Xiao, Y., Che, R., Xu, F., Hu, B., Liang, C., Chu, J., Li, J. y Chu, C. (2015) Activation of Big Grain 1 significantly improves grain size by regulating auxin transport in rice. Proceedings of the National Academy of Sciences, 112 (35), 11102-11107.
Liu, Q., Liu, J., Yuan, L., Lu, L., Tao, M., Zhang, C., Qin, Q., Chen, B., Ma, M., Tang, C. & Liu, S. (2020). The establishment of the fertile fish lineages derived from distant hybridization by overcoming the reproductive barriers. Reproduction 159 (6), 237-249.
Lizarraga, I. (2015). Rendimiento y caracterización agronómicos de 50 líneas elite de trigo de primavera (30 TH - ESWYT) procedentes del CIMMYT-México en la E.E.A “El Mantaro”,2010-11. [Tesis de pregrado, Universidad Nacional del Centro del Perú].
López, O., Ugalde, J. y Acosta (2018) Rendimiento y tolerancia a sequía de genotipos de frijol negro en el Estado de Veracruz. Fitotecniamexicana, 31, 35-39.
Maestu, E. (2018). REGENERACIÓN DE PLANTAS EN CULTIVO IN VITRO DE TOMATE (Solanum lycopersicum L.). Trabajo de final de grado. Universidad Politécnica de Valencia, España.
Mayor, V. (2015). Identificación de QTLs de frijol común (Phaseolus vulgaris) asociados a tolerancia a sequía. [Tesis de doctorado, Universidad Nacional de Colombia].
McGrann, G., Stavrinides, A., Russell, J., Corbitt, M., Booth, A., Chartrain, L., Thomas, W. y Brown, J. (2014). A trade off between mlo resistance to powdery mildew and increased susceptibility of barley to a newly important disease, Ramularia leaf spot. Journal of experimental botany, 65(4), 1025–1037.
Mellado, M. (2014). Nociones básicas sobre genética y mejoramiento vegetal y animal. Revisión de antecedentes. Instituto de Investigaciones Agropecuarias, Centro Regional de Investigación Quilamapu.
Méndez, R., Rodríguez, R., Ramírez, M., Álvarez, M., Vázquez, E., Arcos, G., Tenorio, V., Hernández, S. y Mayek, N. (2015). Identificación de fuentes de resistencia a pudriciones de la raíz en germoplasma de chile serrano (Capsicum annuum L.). Revista mexicana de ciencias agrícolas, 6(7), 1507-1518.
Ministerio de Desarrollo Agrario y Riego. (2021). El Perú se consolida como el primer productor y exportador mundial de quinua. Revista digital de la Cámara de Comercio de Lima.
Nobuyuki, M., Masami, T., Miki, F., Shota, F., Yasufumi, K., Mariko, U., Kojiro, T., Tsutomu, T., Eiji, N., Hiroharu, M., Yasuo, Y. y Yasunari, F. (2017). The genome of Chenopodium quinoa. Nature, 542, 307-510.
Oliveira, P., Barbosa, A., Diniz, R., Oliveira, E., y Ferreira, C. (2018). Molecular marker assisted selection for increasing inbreeding in S1 populations of cassava. Anais da Academia Brasileira de Ciencias, 90(4), 3853–3869.
Pérez, R., Pérez, D. y Hernández, E. (2019). Aporte de la selección recurrente al mejoramiento genético del arroz en Cuba. Infociencia 23(1), 1 - 10.
Platten, J., Cobb, J., y Zantua, R. (2019). Criteria for evaluating molecular markers: Comprehensive quality metrics to improve marker-assisted selection. PloS one, 14(1), 1 - 20
Poehlman, J. M. (1971). Mejoramiento genético de las cosechas. LIMUSA.
Quispe, S. (2017). Quinto ciclo de selección masal estratificada en maíz morado (Zea mays L.), Canaán 2735 msnm, Ayacucho. [Tesis de pregrado, Universidad Nacional de San Cristóbal de Huamanga].
Raney, J., Reynolds, D., Elzinga, D., Page, J., Udall, J. y Jellen, E. (2014). Análisis de transcriptoma del estrés inducido por la sequía en la quinua (Chenopodium quinoa). Soy. 5, 338–357.
Reategui, K., Aguirre, N., Oliva, R. y Aguirre, E. (2019). Fenología y rendimiento de cuatro variedades de papa en el Altiplano. Scientia Agropecuaria, 10(2), 265-274.
Rita, C., Trevisani, N., Viera, T., Guidolín, A. y Meirelles, J. (2017). Nivel de heterocigosidad y su relación con la variabilidad genética mecanismos en frijoles. Revista de Ciencia Agronómica, 48(3), 1-2.
Rodríguez, D. (2017). Potencial de rendimiento de líneas mutantes de arroz (Oryza sativa l.) desarrolladas mediante aplicación de rayos gamma en condiciones del valle de Jequetepeque. [Tesis de pregrado, Universidad Nacional Agraria La Molina].
Romero, R., Mujica, A., Pineda, E., Ccamapaza, Y. y Zavalla, N. (2019). Genetic identity based on simple sequence repeat (SSR) markers for Quinoa (Chenopodium quinoa Willd.). Ciencia e investigación agraria, 46(2), 166-178.
Seminario, J., Villanueva, R. y Valdez, M. (2018). Rendimiento de cultivares de papa (Solanum tuberosum L.) amarillos precoces del grupo Phureja. Agron. Mesoam. 29(3):639-653.
Singh, S.P.(1985). Conceptos basicos para el mejoramiento del frijol por hibridacion. In: López Genes, Marceliano; Fernández O., Fernando O.; Schoonhoven, Aart van (eds.). Frijol: Investigación y producción. Programa de las Naciones Unidas (PNUD); Centro Internacional de Agricultura Tropical (CIAT), Cali, CO. p. 109-126.
Trijatmiko, K., Dueñas, C., Tsakirpaloglou, N., Torrizo, L., Arines, F., Adeva, C., Balindong, J., Oliva, N., Sapasap, M., Borrero, J., Rey, J., Francisco, P., Nelson, A., Nakanishi, H., Lombi, E., Tako, E., Glahn, R., Stangoulis, J., Chadha-Mohanty, P., Johnson, A., Tohme, J., Barry, G. y Slamet-Loedin, I. (2016). Biofortified indica rice attains iron and zinc nutrition dietary targets in the field. Scientific reports, 6(19792).
Torres, D. (2004). Selección asistida por marcadores para la detección de resistencia extrema a PVY en Solanum tuberosum. [Tesis de Licenciatura, Universidad de la Republica de Uruguay].
Torres, I., González M., Villaseñor, H., Huerta, J., Pineda, E., Espitia,E., Guevara, R. y Guevara, L. (2007). Marcadores genéticos de resistencia a roya de tallo (Puccinia graminis Persoon f. sp. avenae) en avena (Avena sativa L.). Agricultura técnica en México, 33(3), 221-230.
Vallejo, F. y Estrada, E. (2016). Mejoramiento Genético de Plantas: Segunda Edición. Universidad Nacional de Colombia.
Velasco, Y., Sana, W y Morillo, A. (2020). Caracterización agromorfológica de cebada (Hordeum vulgare L.) en el Municipio de Chivatá Boyacá, Colombia. Biotecnología en el Sector Agropecuario y Agroindustrial, 18(2), 103-116.
Yasui, Y., Hirakawa, H., Oikawa, T., Toyoshima, M., Matsuzaki, C. y Ueno, M. (2016). Borrador de secuencia genómica de una línea endogámica de quinua (Chenopodium quinoa), un cultivo alotetraploide con gran adaptabilidad ambiental y propiedades nutricionales excepcionales. ADN Res. 23, 535–546.
Zhang, W., Wu, L., Wu, X., Ding, Y., Li, G., Li, J., Weng, F., Liu, Z., Tang, S., Ding, C. y Wang, S. (2016). Lodging Resistance of Japonica Rice (Oryza sativa L.): Morphological and Anatomical Traits due to top-Dressing Nitrogen Application Rates. Rice, 9(1), 31.
Zhu, H.; Li, C.; Gao, C. (2020). Applications of CRISPR-Cas in agriculture and plant biotechnology. Nature Reviews Molecular Cell Biology.