RESUMEN
Los genes blaKPC y blaOXA-48 codifican enzimas carbapenemasas en muchas enterobacterias, confiriéndoles resistencia a los antibióticos carbapenémicos, por ello la presente investigación tuvo por objetivo detectar genes blaKPC y blaOXA-48 en cultivos de Escherichia coli y Klebsiella pneumoniae, aislados a partir de urocultivos positivos en el Instituto Regional de Enfermedades Neoplásicas - IREN Norte, Perú. A 100 cultivos bacterianos, se determinó fenotípicamente la presencia de enzimas carbapenemasas, y los cultivos que resultaron positivos a estas pruebas, fueron sometidos a la detección de genes blaKPC y blaOXA-48 en ADN genómico y plasmídico, mediante PCR convencional. Se encontró que el 59 % de los cultivos bacterianos mostraron fenotípicamente producir betalactamasas de espectro extendido (BLEE), además n=6 resultaron ser positivos a carbapenemasas, pero, en la detección molecular, ninguno de los cultivos bacterianos evidenció poseer los genes buscados. Por lo que se concluyó que en los cultivos de E. coli y K. pneumoniae, aislados a partir de urocultivos positivos en el Instituto Regional de Enfermedades Neoplásicas - IREN Norte, Perú, no se detectó los genes blaKPC y blaOXA-48, sin embargo, fenotípicamente el 6 % evidenciaron poseer carbapenemasas.
Palabras claves: Escherichia coli, Klebsiella pneumoniae, blaKPC, blaOXA-48 y carbapenemasas.
ABSTRACT
The blaKPC and blaOXA-48 genes encode carbapenemases enzymes into many enterobacteria, giving them resistance to carbapenem antibiotics, for this reason, the present investigation aimed to detect blaKPC and blaOXA-48 genes in Escherichia coli and Klebsiella pneumoniae cultures, isolated from positive urine cultures at the Regional Institute of Neoplastic Diseases - IREN Norte, Peru. At in 100 bacterial cultures, the presence of carbapenemase enzymes was phenotypically determined, and the cultures that were positive in these tests were subjected to the detection of blaKPC and blaOXA-48 genes in genomic and plasmid DNA, by conventional PCR. It was found that 59 % of bacterial cultures showed phenotypically producing extended-spectrum betalactamases (BLEE), in addition, n=6 turned out to be positive to carbapenemases, but, in molecular detection, none of the bacterial cultures proved to possess the genes sought. So, it was concluded that in the crops of E. coli and K. pneumoniae, isolated from positive urine cultures at the Regional Institute of Neoplastic Diseases - IREN Norte, Peru, the blaKPC and blaOXA-48 genes were not detected, however, phenotypically 6 % reported possessing carbapenemases.
Keywords: Escherichia coli , Klebsiella pneumoniae, blaKPC gene, blaOXA-48 gene and carbapenems.
http://dx.doi.org/10.17268/rebiol.2020.40.02.04
1. INTRODUCCIÓN
Dentro del grupo de antibióticos, de amplio espectro más utilizados, se encuentran los carbapenémicos, los cuales son hidrolizados por enzimas carbapenemasas de las bacterias, ocasionado el aumento de resistencia a estos antibióticos a nivel mundial (Ferranti et al., 2018). Estas enzimas están incluidas en las clases A, B y D, según la clasificación de Ambler, donde, “las enzimas clases A y D incluyen a ß-lactamasas que tienen un residuo de serina en su sitio activo y pertenecen a serin-betalactamasas”, por otro lado, “las enzimas de clase B tienen uno o dos iones zinc como cofactor enzimático, denominándose metalo-betalactamasas” (Katchanov et al., 2018).
Se ha demostrado que, las carbapenemasas, son frecuentes en bacilos Gram negativos, principalmente en Klebsiella pneumoniae y Escherichia coli , además, “las carbapenemasas frecuentemente identificadas son enzimas del tipo KPC, NDM-1, IMP, VIM, OXA-48 (de oxacilinasa) y OXA-181” (Nordmann et al., 2012). Especial atención recibe la enzima KPC, detectada en Carolina del Norte en 1996, y codificada por el gen blaKPC localizado en el transposón Tn4401, o en elementos genéticos similares a Tn4401(Escandón-Vargas et al., 2017). Por otro lado, OXA-48 es una betalactamasa de clase D, que hidroliza las penicilinas y los carbapenémicos; el gen que lo codifica es blaOXA-48 y se ha detectado solo en plásmidos de bacterias de la familia Enterobacteriaceae (Poirel et al., 2012).
Los fármacos utilizados en las quimioterapias en pacientes con cáncer, poseen propiedades mutagénicas y genotóxicas que afectan el genoma bacteriano contribuyendo a la resistencia a los antibióticos (Chistyakov et al., 2018). En Los centros de salud de pacientes oncológicos se ha reportado a E. coli y K. pneumoniae, como los patógenos bacterianos más comunes y de mayor producción de betalactamasas de espectro extendido (BLEE) (Chander y Shrestha, 2013). En España, un estudio reportó que de 203 aislamientos confirmados de K. pneumoniae, productora de carbapenemasas, 153 presentaron el gen blaOXA-48 y 6 el gen blaKPC (Oteo et al., 2013); así mismo; otra investigación en el mismo país recolectó 3 901 aislamientos de K. pneumoniae, de donde 171 fueron carbapenemasas positivos, de los cuales, 85 de ellos presentaron el gen blaOXA-48 (Yoon et al., 2018).
En países latinoamericanos también se han reportado estos genes; en Brasil, se aislaron 297 especies bacterianas, de los cuales, 100 (33,7 %) fueron positivos para KPC, de estos K. pneumoniae tuvo la tasa de positividad más alta de (60,0 %) (Saldanha et al., 2016). Un estudio en Chile demostró una incidencia moderada para E. coli productora de BLEE en la Unidad de Cuidados Intensivos, en tanto K. pneumoniae fue la que tuvo mayor incidencia. El estudio concluyó que las Enterobacterias resistentes a carbapenémicos, es un problema que afecta a 79 % de los servicios hospitalarios estudiados (Acuña et al., 2017).
En el Perú, el primer reporte de KPC en K. pneumoniae fue en el 2013, pero en el 2016 se informó la caracterización de la enzima KPC-2 producida en K. pneumoniae ST340 (Horna et al., 2017). Por otro lado, entre los años 2013 y 2017, se estudió la presencia de genes de resistencia a los carbapenémicos en enterobacterias aisladas en 12 hospitales, donde se identificaron 83 cepas con carbapenemasas, de las cuales 26 (31,3 %) portaron el gen blaKPC (Sacsaquispe-Contreras y Bailón-Calderon, 2018).
El incremento de la resistencia bacteriana frente a los antibióticos es un problema mundial, sobre todo contra aquellos antibióticos que son considerados como últimas alternativas y recursos para el tratamiento, como lo son los carbapenémicos. En el Perú es de interés en salud pública, la detección de estas bacterias y debido a que son pocos los estudios en la zona norte de nuestro país sobre el tema expuesto, se realizó un estudio descriptivo con el objetivo de detectar genes blaKPC y blaOXA-48 en cultivos de E. coli y K. pneumoniae aislados a partir de urocultivos positivos en el Instituto Regional de Enfermedades Neoplásicas – IREN Norte, Perú.
2. MATERIAL Y MÉTODOS
Material biológico
Se evaluaron 72 cultivos de Escherichia coli y 28 de Klebsiella pneumoniae, aislados de urocultivos positivos y proporcionados por el servicio de laboratorio clínico del Instituto Regional de Enfermedades Neoplásicas - IREN Norte, Perú. Además, se usó como controles para los ensayos, las cepas bacterianas K. pneumoniae ATCC® 700603, E. coli ATCC® 25922, K. pneumoniae ATCC® BAA-1705 y K. pneumoniae ATCC® BAA-1706.
Métodos y técnicas
Test de sensibilidad a antibióticos para detección de BLEE
Se realizó de acuerdo con los lineamientos del Clinical and Laboratory Standards Institute (CLSI, 2015). El método fue la difusión en discos (Figura 3), en la cual se usó placas de Petri con Agar Mueller Hinton, y discos de ceftazidima 30 ug, ceftazidima-ácido clavulánico 30/10 ug, cefotaxima 30 ug y cefotaxima-ácido clavulánico 30/10 ug. Se aplicó el mismo método para las cepas control, K. pneumoniae ATCC 700603, como control positivo; y E. coli ATCC 25922, como control negativo.
Detección fenotípica de enzimas carbapenemasas
Se realizó de acuerdo con los lineamientos del CLSI propuestos para el método de Hodge modificado (Argente et al., 2017; CLSI, 2015). Se utilizó como antibiótico al meropenem 10 ug, y como cepas controles, K. pneumoniae ATCC BAA-1705, como control positivo; y K. pneumoniae ATCC BAA-1706, como control negativo (Figura 4).
Detección fenotípica de enzimas carbapenemasas clase A
Se utilizó el test con Ácido Fenil Borónico (Pasteran et al., 2010). Se trabajó con los cultivos bacterianos que resultaron positivos en el método de Hodge modificado, y se siguió el procedimiento del test de difusión de doble disco usando como antibióticos, discos de imipenem 10 ug y meropenem 10 ug (Figura 5),
Detección fenotípica de enzimas carbapenemasas clase D
Se utilizó el test con discos de Piperacilina/Tazobactam (Huang et al., 2014). Se trabajó con los cultivos bacterianos que resultaron positivos en el método de Hodge modificado, y se siguió el procedimiento propuesto por el CLSI (2015), usando como antibióticos, discos de meropenem 10 ug y piperacilina/tazobactam 100/10 ug.
Detección molecular de genes blaKPC y blaOXA-48
Los cultivos bacterianos positivos a las pruebas fenotípicas en donde se detectó enzimas carbapenemasas, fueron sometidas a este ensayo.
A dichos cultivos, se extrajo y purificó ADN genómico usando el kit Gene Jet Genomic Dna Purif; y para la extracción y purificación de ADN plasmídico se usó el kit Gene JET Plasmid Miniprep.
Se evalúo la integridad del ADN genómico y plasmídico mediante electroforesis, así como también la concentración y pureza mediante espectrofotometría con una relación de absorbancia 260/280.
Para realizar la detección de los genes en estudio mediante la Reacción en Cadena de la Polimerasa (PCR), se utilizó el kit Platinium PCR SuperMix, electroforesis en gel de agarosa al 1 %, y para el registro fotográfico un equipo BioDocAnalyze. Las cepas bacterianas usadas como controles fueron K. pneumoniae ATCC® BAA-1705, como control positivo; y E. coli ATCC® 25922, como control negativo (Figura 6).
3. RESULTADOS
A 72 cultivos de Escherichia coli y 28 de Klebsiella pneumoniae, aislados de urocultivos positivos y proporcionados por el servicio de laboratorio clínico del Instituto Regional de Enfermedades Neoplásicas - IREN Norte, Perú, se les realizó ensayos para detectar fenotípicamente enzimas tipo betalactamasas de espectro extendido (BLEE) y carbapenemasas, resultados que se muestran en las Tablas 1-3.
Tabla1. Número de cultivos bacterianos de E. coli y K. pneumoniae, en los cuales se detectó fenotípicamente enzimas tipo betalactamasas de espectro extendido (BLEE).

Tabla 2.Número de cultivos de E. coli y K. pneumoniae que evidenciaron fenotípicamente poseer enzimas carbapenemasas, según el Test de Hodge modificado (MHT).

Tabla 3.Número de cultivos de E. coli y K. pneumoniae que evidencian producir enzimas tipo carbapenemasas de clase A y D.

A los 4 cultivos de E. coli que resultaron positivos a la prueba con Ácido Fenil Borónico, que indicó fenotípicamente poseer carbapenemasas clase A, se realizó ensayos para detectar genes blaKPC en ADN genómico y plasmídico respectivamente, mediante PCR convencional; los resultados de dichos ensayos se muestran en las Figuras 1 y 2.

Figura 1.Detección de gen blaKPC en ADN genómico por PCR convencional. Carriles: 1) Marcador de Peso Molecular (100pb); 2) Control positivo; 3 Control negativo; 4-7) cultivos bacterianos evaluados.

Figura 2.Detección de gen blaKPC en ADN plasmídico por PCR convencional. Carriles: 1) Marcador de Peso Molecular (100pb); 2) Control positivo; 3-6) cultivos bacterianos evaluados.
4. DISCUSIÓN
Las infecciones en el tracto urinario son las más frecuentes en las personas, y,Escherichia coli junto con Klebsiella pneumoniae son las bacterias que más predominan como causantes de estas infecciones. Estos patógenos han desarrollado alta resistencia contra los betalactámicos debido a la producción de las enzimas denominadas betalactamasas de espectro extendido (BLEE) (Chander y Shrestha, 2013). En este estudio, como se muestra en la Tabla 1, el 59 % (n=59) de los cultivos bacterianos estudiados evidenció fenotípicamente poseer BLEE, siendo más frecuente en cultivos de E. coli (61 %) que K. pneumoniae (54 %). Estos datos se sustentan en el estudio de Chander y Shrestha (2013) quienes encontraron que E. coli está relacionado con los más altos porcentajes de BLEE a diferencia de K. pneumoniae.
Se ha reportado resistencia a los carbapenémicos en bacterias productoras de BLEE, en las cuales coexisten genes que codifican para enzimas que confieren resistencia a otros antibióticos, eso explica como las enzimas carbapenemasas confieren resistencia a la mayoría de los betalactámicos (Bakthavatchalam et al., 2016), es por ello, que en las prueba fenotípica para detectar a las enzimas carbapenemasas, el 7 % de los cultivos de E. coli evaluados resultaron positivos al test de Hodge modificado, mientras que en el caso de K. pneumoniae solo el 4 % resultaron positivos (Tabla 2). Sin embargo, esta prueba puede presentar resultados como falsos positivos de hasta 25 %. Informes reportaron que ello podría deberse a la hiper producción del Amp C y a miembros de la familia de BLEES como CTX-M (Pasteran et al., 2010). Por tanto, debido a la falsa positividad que puede presentar el test del Hodge modificado, se realizó la prueba con discos de ácido Fenil Borónico como confirmación, debido a que éste tiene la propiedad de inhibir a las carbapenemasas de clase A. En tal sentido, como se observa en la Tabla 3, de los 6 cultivos que resultaron positivos al test de Hodge modificado, 4 reaccionaron positivamente ante el Ácido Fenil Borónico, siendo todas E. coli.
Por otro lado, para confirmar la presencia de carbapenemasas de clase D se realizó el test con discos de piperacilina/tazobactam, debido a que, OXA-48 presenta alta resistencia a tales discos. A pesar de ello, no se confirmó ningún cultivo bacteriano positivo para esta prueba (Tabla 3). Estos resultados coinciden con estudios realizados en Lima – Perú durante los años 2013 al 2017, donde ya se ha reportado enzimas de clase A (enzima KPC) sin embargo, hasta la fecha no se ha reportado ninguna enzima de clase D (enzima OXA – 48) (Sacsaquispe-Contreras y Bailón-Calderón, 2018).
En la prueba de PCR que se realizó a los 4 cultivos bacterianos que resultaron positivos a la prueba con Ácido Fenil Borónico no se evidenció ningún resultado positivo para la detección de genes blaKPC y blaOXA-48 ni para la reacción con el ADN genómico (Figura 1) ni para con el ADN plasmídico (Figura 2). En el caso de la ausencia del gen blaKPC, esto puede ser explicado debido a que en las carbapenemasas de clase A no solo está la enzima KPC, sino otras enzimas como las de tipo IMI, SME, NMC y GES como lo afirman Cifuentes et al. (2012), que en este caso no fueron motivo de estudio, pero que muy probablemente pudieron haberlos portado. Sobre la ausencia del gen blaOXA 48 que codifica para la enzima OXA – 48, estos resultados se apoyan en estudios como lo mencionan Vera-Leiva et al. (2017), donde se ha detectado la presencia de esta enzima en países europeos, África y Chile, mas no existe reporte de la detección de esta enzima en Perú.
En el Perú los últimos casos reportados para la presencia del gen blaKPC corresponden al año 2017 en un estudio realizado en Lima (Horna et al., 2017), sin embargo, en Trujillo Perú aún, no se han reportado enterobacterias portadoras de estos genes.
5. CONCLUSIÓN
En los 100 cultivos de Escherichia coli y Klebsiella pneumoniae, asilados a partir de urocultivos positivos en el Instituto Regional de Enfermedades Neoplásicas - IREN Norte, Perú; no se detectó presencia de los genes blaKPC y blaOXA-48, sin embargo, el 6 % de ellos evidenciaron fenotípicamente poseer enzimas carbapenemasas.
6. AGRADECIMIENTOS
Los autores expresan su agradecimiento a los responsables del Laboratorio de Genética, Reproducción Asistida y Biología-Molecular, de la Universidad Privada Antenor Orrego, y del Laboratorio de Fisiología y Genética Bacteriana, de la Universidad Nacional de Trujillo, por brindar sus instalaciones para la realización de las pruebas moleculares y fenotípicas, respectivamente, de la presente investigación.
7. CONTRIBUCIÓN DE LOS AUTORES
Todos los autores participaron en la redacción del manuscrito inicial, revisión bibliográfica, y en la revisión y aprobación del manuscrito final. Y.G., G.H y D.Z. realizaron los ensayos de detección fenotípica. Y.G., G.H., R.C. y P.M. realizaron los ensayos de detección genotípica.
8. CONFLICTO DE INTERESES
Los autores declaran no tener conflicto de interés alguno.
9. REFERENCIAS BIBLIOGRÁFICAS
Acuña, M., Cifuentes, M., Silva, F., Rojas, A., Cerda, J., y Labarca, J. (2017). Incidencia de bacterias multirresistentes en unidades de cuidados intensivos de hospitales chilenos. Rev Chilena Infectol, 34(6), 570-575. https://doi.org/10.4067/S0716-10182017000600570.
Argente, M., Miró, E., Martí, C., Vilamala, A., Alonso-Tarres, C., Ballester, F., Calderón, A., Gallés, C., Gasós, A., Mirelis, B., Morta, M., Olsina, M., Sauca, G., Sierra, M., Rivera, A., Navarro, F. (2017). Molecular characterization of OXA-48 carbapenemase-producing Klebsiella pneumoniae strains after a carbapenem resistance increase in Catalonia. Enferm Infecc Microbiol Clin. 37(2), 82-89. https://doi.org/10.1016/j.eimc.2018.02.003.
Bakthavatchalam, Y., Anandan, S., y Veeraraghavan, B. (2016). Laboratory Detection and Clinical Implication of Oxacillinase-48 like Carbapenemase: The Hidden Threat. J Glob Infect Dis, 8(1), 41-50. http://dx.doi.org/10.4103/0974-777X.176149.
Cifuentes, M., García, P., San Martin, P., Silva, F., Zúñiga, J., Reyes, S., Rojas, R., Ponce, R., Quinilla, R., Delpiano, L., y Wolff, M. (2012). Primer caso de detección de blaKPC en Chile: desde Italia a un hospital público de Santiago. Rev Chilena Infectol. 29(2), 224-228. http://dx.doi.org/10.4067/S0716-10182012000200018.
Chander, A., y Shrestha, C. (2013). Prevalence of extended spectrum betalactamase producing Escherichia coli and Klebsiella pneumoniae urinary isolates in a tertiary care hospital in Kathmandu, Nepal. BMC Research Notes, 6, 487-493. http://dx.doi.org/10.1186/1756-0500-6-487.
Chistyakov, V., Prazdnova, E., Mazanko, M., Churilov, M., y Chmyhalo, V. (2018). Increase in Bacterial Resistance to Antibiotics after Cancer Therapy with Platinum-Based Drugs. Mol Biol, 52(2), 232-236. https://doi.org/10.1134/S0026893317050077.
Clinical and Laboratory Standards Institute [CLSI]. (2015). Estándares de desempeño para pruebas de susceptibilidad de disco antimicrobiano; estándar aprobado, doceava edición CLSI documento M02-A12. Instituto de Estándares Clínicos y de Laboratorio, Wayne, PA. EE.UU.
Escandón-Vargas, K., Reyes, S., Gutiérrez, S., y Villegas, M. (2017). The epidemiology of carbapenemases in Latin America and the Caribbean. Expert Rev Anti Infect Ther, 15, 277-297. http://dx.doi.org/10.1080/14787210.2017.1268918.
Ferranti, M., Schiaroli, E., Palmieri, M., Palmieri, M., Repetto, A., Vecchiarelli, A., Francisci, D., Mencacci, A., y Monari, C. (2018). Carbapenemase-producing Enterobacteriaceae isolates resistant to last-line antibiotics in an Italian general hospital. New Microbiol. 41(4),274-281. PMID: 30252925.
Horna, G., Velasquez, J., Fernandez, N., Tamariz, J., y Ruiz, J. (2017). Characterization of the first KPC-2- producing Klebsiella pneumoniae ST340 from Perú. J Glob Antimicrob Resist, 9, 36-40. https://doi.org/10.1016/j.jgar.2016.12.011.
Huang, T., Poirel, L., Bogaerts, P., Berhin, C., Nordmann, P., y Glupczynski, Y. (2014). Temocillin and piperacillin/tazobactam resistance by disc diffusion as antimicrobial surrogate markers for the detection of carbapenemase producing Enterobacteriaceae in geographical areas with a high prevalence of OXA-48 producers. J Antimicrob Chemother, 69(2), 445-450. http://dx.doi.org/10.1093/jac/dkt367.
Katchanov, J., Asar, L., Klupp, E.M., Both, A., Rothe, C., König, C., Rohde, H., Kluge, S., y Maurer, F.P. (2018). Carbapenem-resistant Gram-negative pathogens in a German university medical center: Prevalence, clinical implications and the role of novel βlactam/β-lactamase inhibitor combinations. PLoS One, 13(4): e0195757. http://dx.doi.org/10.1371/journal.pone.0195757.
Nordmann, P., Dortet, L., y Poirel, L. (2012). Carbapenem resistance in Enterobacteriaceae: here is the storm! Trends Mol Med, 18,263. http://dx.doi.org/10.1016/j.molmed.2012.03.003.
Oteo, J., Saez, D., Bautista, V., Fernandez-Romero, S., Hernandez-Molina, J.M., Perez-Vasquez, M., Aracil, B., y Campos, J. (2013). Carbapenemase-Producing Enterobacteriaceae in Spain in 2012. Antimicrob Agents Chemother. 57(12), 6344-6347. http://dx.doi.org/10.1128/AAC.01513-13.
Pasteran, F., Mendez, T., Rapoport, M., Guerriero, L., y Corso, A. (2010). Controlling False – Positive Results Obtained with the Hodge and Masuda Assays for Detection of Class A Carbapenemase in Species of Enterobacteriaceae by Incorporating Boronic Acid. J Clin Microbiol, 48(4), 1323-1332. http://dx.doi.org/10.1128/JCM.01771-09.
Poirel, L., Bonnin, R., y Nordmann, P. (2012). Características genéticas del plásmido generalizado que codifica la carbapenemasa OXA-48. Agentes antimicrobianos y quimioterapia, 56(1), 559–562. http://dx.doi.org/10.4067/S0716-10182017000500476.
Saldanha, P.C., Souza, A., Garcia, S., Gomez, S., Monteiro-Neto, V., Melo, M.M., García, A.C., Gómez, R., Goncalves, S., y Quaresma, M.R. (2016). Phenotypic and molecular detection of the blaKPC gene in clinical isolates from inpatients at hospitals in São Luis, MA, Brazil. BMC Infect Dis, 16,737. https://doi.org/10.1186/s12879-016-2072-3.
Sacsaquispe-Contreras, R., y Bailón-Calderon, H. (2018). Identificación de genes de resistencia a carbapenemicos en enterobacterias de hospitales de Perú, 2013-2017. Rev Peru Med Exp Salud Pública, 35(2), 25-64. http://dx.doi.org/10.17843/rpmesp.2018.352.3829.
Vera-Leiva, A., Barría-Loaiza, C., Carrasco-Anabalón, S., Lima, C., Aguayo-Reyes, A., Domínguez, M., Bello-Toledo, H., y González-Rocha, G. (2017). KPC: Klebsiella pneumoniae carbapenemasa, principal carbapenemasa en enterobacterias. Rev Chilena Infectol, 34(5), 476-484. http://dx.doi.org/10.4067/S0716-10182017000500476.
Wojkowska-Mach, J., Godman, B., Glassman, A., Kurdi, A., Pilc, A., Rozanska, A., Skoczyński S., Wałaszek, M., y Bochenek, T. (2018.) Antibiotic consumption and antimicrobial resistance in Poland; findings and implications. Antimicrob Resist Infect Control, 7, 136-146. https://doi.org/10.1186/s13756-018-0428-8.
Yoon, E.J., Kim, J.O., Kim, D., Lee, H., Yang, J.W., Lee, K.J., y Jeong, S.H. (2018). Klebsiella pneumoniae Carbapenemase Producers in South Korea between 2013 and 2015. Front Microbiol, 25, 9-56. https://doi.org/10.3389/fmicb.2018.00056.
Citar como:
González, Y.; Huayán, G.; Zavaleta-Verde, D.; Mercado, P.; Castillo, R. 2020. Detección de genes de resistencia a carbapenémicos en Escherichia coli y Klebsiella pneumoniae aislados de un centro de salud de Trujillo - Perú. REBIOL 40(2):160 -169. DOI: http://dx.doi.org/10.17268/rebiol.2020.40.02.04.
Anexos

Figura 3.Diferencias entre los diámetros de los halos de inhibición que confirmaron un resultado positivo a la prueba de sensibilidad antimicrobiana para detectar BLEE en uno de los cultivos bacterianos evaluados.

Figura 4..Hendidura que indicó un resultado positivo a la prueba de Hodge modificado, confirmando la presencia fenotípica de carbapenemasas en uno de los cultivos bacterianos evaluados.

Figura 5.Ensanchamiento de halos hacia el Ácido Fenil Borónico (APB), indicó resultado positivo a la presencia de enzimas carbapenemasas de clase A.
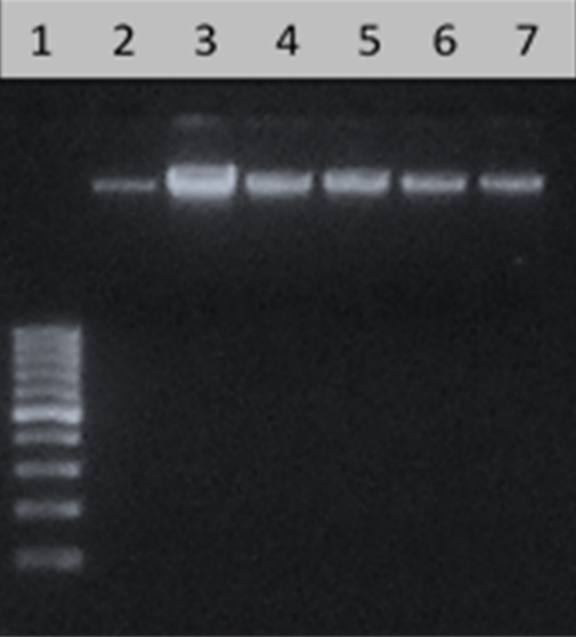
Figura 6.Perfil electroforético para verificar integridad de ADN genómico. Carriles: 1) Marcador de Peso Molecular (100pb); 2) Control positivo; 3) Control negativo; 4-7) cultivos bacterianos evaluados.