Artículo de revisión
Historia evolutiva de los genes Sox durante del proceso de duplicación génica
Evolutionary history of the Sox genes throughout gene duplication
Anthony Leon 1
1 SARS International Centre for Marine Molecular Biology
Thormøhlens Gate 55, 5008 Bergen. Noruega
*Autor para correspondencia: Anthony.Leon@uib.no
Recibido: 18 de Noviembre, 2019. Aceptado: 2 de Diciembre, 2019.
Resumen
La familia de genes Sox codifica importantes factores de transcripción involucrados en las primeras etapas de desarrollo embrionario. Contienen una región conservada llamada caja HMG que es, al menos, 50% similar a la caja HMG del gen Sry. Se han propuesto diversos modelos filogenéticos que permitan una mejor comprensión de su evolución. Dichos modelos sugieren una posible aparición en el último ancestro común, así como una posterior divergencia en los metazoos tempranos debido a varios eventos de duplicación de genoma. Las copias duplicadas adquirieron nuevas funciones o pasaron por un proceso de sub-funcionalización que condujo a la aparición de nuevas subfamilias génicas en diferentes grupos animales. Aquí, revisamos el conocimiento actual sobre la familia de genes Sox, su evolución y sus funciones.
Palabras clave: Genes Sox, proteínas SOX, caja HMG, duplicación génica, filogenia.
Abstract
Sox gene family encodes important transcription factors involved in early stages of embryonic development. They contain a conserved region called HMG-box which is at least 50% similar to the HMG-box of Sry gene. Several phylogenetic models have been proposed for a better understanding of their evolution suggesting a possible emergence in the last common ancestor, and a posterior divergence in early metazoans due to several genome duplication events. Duplicated copies acquired new functions or went through sub-functionalization leading to the emergence of new gene sub-families in different animal clusters. Here, we review the current knowledge on Sox gene family, their evolution and their functions.
Keywords: Sox genes, SOX proteins, HMG-box, gene duplication, phylogeny.
DOI: http://dx.doi.org/10.17268/rebiol.2019.39.02.06
INTRODUCCIÓN
Los genes Sox codifican una familia de factores de transcripción (FT) que son esenciales en varios procesos de desarrollo. Contienen un dominio de unión al ADN llamado HMG (High mobility group) en el caso de las proteínas (o “caja-HMG”, en el caso de los genes), el cual es altamente conservado en la mayoría de ellos. Parecen estar restringidos solo a animales y varios estudios los han clasificado en subgrupos según la homología de sus dominios HMG y otros motivos estructurales (Bowles et al, 2000). Se cree que los genes Sox emergieron en los primeros metazoos, los cuales ya poseían una caja-HMG parcial que luego paso por procesos de duplicaciones, mutaciones dispersas y adquisición funcional. De hecho, algunas de estas cajas HMG duplicadas dejaron copias redundantes libres que pudieron adquirir nuevas funciones, o ser silenciadas y/o reducidas por mutaciones acumulativas. Estos eventos moleculares condujeron a la formación de 6 grupos Sox en los primeros animales bilaterales; mientras que los posteriores reordenamientos en sus dominios proteicos permitieron la aparición de grupos adicionales. La característica más obvia de la conservación del dominio HMG dentro de los diversos grupos de la familia Sox es su posición dentro de la proteína entera, así como la longitud total de la misma (Bowles et al, 2000; Wegner, 1999).
En este artículo, revisaremos aspectos generales de los genes Sox, así como historia evolutiva en metazoos, pasando por los eventos de duplicación de genes hasta los eventos moleculares que condujeron a la divergencia de varios grupos con gran complejidad. Luego, nos centraremos en la forma en que la evolución de estos genes condujo a los procesos de neo-funcionalización y sub-funcionalización. Finalmente, mencionaremos las principales diferencias y funciones de los grupos de genes Sox después del proceso evolutivo.
Proteínas SOX como factores de transcripción importantes
Los factores de transcripción son esenciales para el desarrollo y mantenimiento de los organismos. Este es el caso de las proteínas SOX, que están involucradas en las primeras etapas del desarrollo embrionario y, dado que presentan un dominio de unión al ADN, también se consideran factores de transcripción arquitectónicos involucrados en la coordinación de la estructura local de la cromatina (Canning & Lovell-Badge, 2002; Chau et al, 2000).
SOX significa “Sry-related HMG box” (caja-HMG relacionada al gen Sry), y estos genes comparten convencionalmente al menos un 50% de similitud con la secuencia de la caja-HMG del gen Sry. Dicho gen codifica una proteína de 79 aminoácidos, dispuesta en tres hélices. De acuerdo con su dominio HMG, las proteínas SOX están compuestas en 2 familias: "TCF / SOX / MATA" (dominios HMG específicos de secuencia única) y "HMG / UBF" (dominios HMG múltiples). Es importante mencionar que todas las proteínas SOX presentan dominio HMG, pero no todas las proteínas con dominio HMG son proteínas SOX. De hecho, todas en conjunto forman parte de un grupo completo de proteínas llamado Grupo de Alta Movilidad (Bowles et al, 2000; Chau et al, 2000; Qi-long et al, 2012). Según la similitud en la secuencia de la caja-HMG, la función, la estructura del gen y su ubicación en los cromosomas, las familias de genes Sox se puede subdividir en los grupos A-J. El grupo Sox A (el cual corresponde al gen Sry) es específico de los mamíferos eutherianos, mientras el resto de los grupos se encuentran en la mayoría de los metazoos superiores, excepto los grupos G y J (Figura 1). Los miembros de algunos subgrupos SOX comparten características adicionales al dominio HMG. Por ejemplo, los miembros del Grupo E que incluye SOX9 y SOX10 comparten una identidad de aminoácidos del 41% en su dominio C-terminal de 66 aminoácidos que, en SOX9, se sabe que es necesario para la transactivación (Bowles et al, 2000; Chau et al, 2000; Argentaro et al, 2000). En la actualidad, se han clonado más de 40 pares ortólogos de genes Sox en el reino animal, incluidos más de 30 en vertebrados y más de una docena en invertebrados. A partir de ellos, se han identificado 22 genes Sox en mamíferos, 20 de ellos presentes en humanos y ratones. Muchos de estos presentan funciones puntuales en los procesos de neurogénesis, condrocitogénesis, diferenciación sexual y otros. Por estas razones, son de gran importancia en el desarrollo de los tejidos donde se expresan ya que, al producirse mutaciones en dichos genes, es probable que también ocurra un desarrollo anormal en sus tejidos diana (Qi-long et al, 2012; Schepers et al, 2002; Koopman et al, 2004; Cremazy et al, 1998; Popovic et al, 2009).
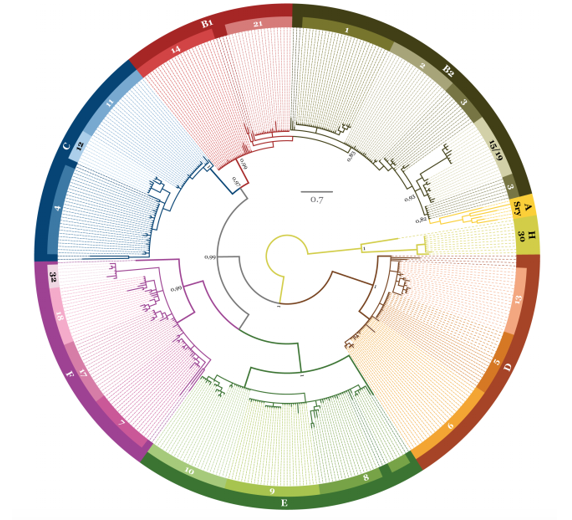
Figura1. Filogenia propuesta de la familia Sox: Inferencia bayesiana basada en las secuencias de los dominios HMG de las proteínas SOX. Cada grupo contiene al menos un representante en invertebrados (Extraído de Voldoire, 2013).
Origen de los genes Sox
Al igual que otros factores de transcripción, es posible que los genes Sox tengan su origen en los metazoos tempranos, ya que dicho evento estaría correlacionado con la diferenciación morfológica y la adquisición de complejidad celular en todos los animales. Además, varios estudios muestran que la ausencia de ciertas familias de factores de transcripción específicas de metazoos se correlaciona con el bajo potencial de diferenciación celular en organismos más primitivos, como los coanoflagelados (Crow & Wagner, 2006; King et al, 2008). Filogenéticamente, aún existe una gran controversia sobre el origen de los metazoos a partir del grupo porífera, ya que las esponjas podrían constituir un grupo separado dentro la clasificación clásica de los animales. Sin embargo, se cree que tanto los coanoflagelados, las esponjas y otros metazoos tempranos compartieron un ancestro común unicelular a fines del período Precámbrico, hace más de 600 millones de años. Sin embargo, esta hipótesis conlleva a algunas preguntas: ¿Cuál fue el antepasado de los animales? ¿Era parecido a una esponja, o a un coanoflagelado? ¿Dichos organismos presentaban genes Sox? ¿Y cómo evolucionaron estos genes?
Choanoflagellatea, el grupo hermano de los animales
Los coanoflagelados son los parientes más cercanos conocidos de los metazoos y constituyen su grupo hermano dentro del grupo monofilético Filozoa. Por esta razón, los investigadores creen que el ancestro común de ambos fue un organismo similar a un coanoflagelado y es probable que tuviera características primitivas de multicelularidad. Esto significa que dicho ancestro pudo organizarse en colonias y adoptar funciones específicas con respecto a otros miembros de la misma colonia. Estos organismos unicelulares poseen algunas proteínas de señalización y adhesión que son características de los metazoos, como las proteínas Sox-like (similares a Sox). Sin embargo, no poseen una gran cantidad de factores de transcripción que si están presentes en los metazoos, y exhiben una posible pérdida de genes homeobox durante su historia evolutiva. Esta pérdida sugiere que, en el curso de la evolución de los coanoflagelados, la complejidad de los factores de transcripción y la diversidad de tipos celulares podrían haber sufrido una reducción secundaria (Crow & Wagner, 2006; King et al, 2008; Larroux et al, 2008).
N. King y colaboradores, luego de trabajar con el coanoflagelado Monosiga brevicollis, determinaron que este posee un conjunto de familias de factores de transcripción que, anteriormente, se creía ser específico de metazoos. Estos FT pueden haber tenido roles críticos durante la evolución de los antepasados metazoos al regular la expresión diferencial de ciertos genes y permitir la emergencia de múltiples tipos celulares dentro de un solo organismo. El posterior incremento de tamaño en las familias de FT, dentro de los eumetazoos, se correlaciona con la complejidad morfológica y de tipo celular dentro de este filo. Esto sugiere que, el aumento en el número de genes que codifican FT puede haber sido un factor importante en la expansión del potencial de desarrollo y la aparición de nuevos tipos celulares (King et al, 2008; Larroux et al, 2006; Larroux et al, 2008).
Es posible que gran parte del repertorio de genes necesarios para el desarrollo de los animales bilaterales haya evolucionado antes de la divergencia entre esponjas, cnidarios y bilaterales. Sin embargo, los mecanismos de invención de nuevos genes en la base del filo Metazoa, y la integración de dichos genes para crear la red de señalización celular y regulación transcripcional fundamental para el desarrollo de todos los animales, sigue siendo un misterio. Se ha sugerido que los reordenamientos extensivos dentro de los dominios de las proteínas podrían haber conducido a nuevas funciones en la línea evolutiva de los metazoos y coanoflagelados. Por otro lado, también se conoce que una gran diversidad de genes que contienen cajas homeobox evolucionaron antes de la divergencia entre esponjas y eumetazoos (como ANTP, Prd-like, Pax, POU, LIM-HD, Six y TALE). Con respecto a los grupos Sox, Bowles afirma que para cada uno de los grupos SOX actualmente reconocibles, ya existía una única forma ancestral antes del origen del linaje de los vertebrados (Bowles et al, 2000; Cock et al, 2010; Degnan et al, 2009).
Evolución de los factores de transcripción en metazoos tempranos
Clásicamente, se consideran a las esponjas como el grupo de metazoos más antiguo, ya que la posición filogenética de los ctenóforos aún no ha sido resuelta. Los coanocitos presentes en las esponjas comparten características similares a los coanoflagelados, y debido a esto se cree que estas puedan provenir a partir de dichos organismos unicelulares. En comparación con los eumetazoos, los poríferos exhiben algunas diferencias remarcables. Por ejemplo, las esponjas parecen no tener mesodermo y, según estudios en la demosponja Amphimedon queenslandica, no poseen FT involucrados en el desarrollo del mesodermo. Por otro lado, si poseen varios factores de transcripción implicados en la determinación o diferenciación de músculos y nervio (PaxB, Lhx, SoxB, Msx, Mef2, Irx y bHLH), a pesar de carecer de un sistema neuromuscular. La presencia de SoxB en las esponjas indica que este gen fue uno de los primeros de la familia Sox en aparecer durante la evolución de los metazoos. De hecho, Larroux y colaboradores indican la presencia de 4 genes Sox en A. queenslandica (AmqSoxB1, AmqSoxB2, AmqSoxC y AmqSoxF) (Cock et al, 2010; Degnan et al, 2009; Larroux et al, 2008; Srivastava et al, 2010).
Sin embargo, Amphimedon carece de otros genes, como los genes Hox, y de familias completas de factores de transcripción que son importantes en el modelado del sistema nervioso y el plan corporal en animales bilaterales. Se desconoce la forma en que los metazoos tempranos adquirieron gran complejidad después divergir de las esponjas, pero muchos autores han establecido una correlación entre el proceso de duplicación genómica y la evolución de la complejidad y diversidad (Crow & Wagner, 2006; Degnan et al, 2009). Kaessmann da un ejemplo con respecto a la duplicación genómica en esponjas. Se sugiere que la primera duplicación de genes Pax ocurrió antes de que los poríferos divergieran. Esto implica que las esponjas, las cuales carecen de células neurales y de órganos modelados por genes Pax en animales superiores, ya presentaban al menos dos copias de estos genes (Kaessmann, 2010; Breitling & Gerber, 2000).
Duplicación genómica y duplicación génica.
La duplicación genómica (o conocida como whole genome duplication, o WGD) es el evento por el cual el genoma entero de un organismo se duplica; mientras que la duplicación génica corresponde a la duplicación de un gen, o grupo de genes específicos, dentro del genoma de un organismo (más conocida como duplicación en tándem). Ambos procesos constituyen un mecanismo importante para la evolución de la complejidad fenotípica, la diversidad, la innovación y el origen de nuevas funciones genéticas. Además, se considera que los eventos de duplicación genómica reducen el riesgo de extinción a través de varios medios: por redundancia funcional, robustez mutacional y mayores tasas de evolución y adaptación (Meyer & Van de Peer, 2005; Van de Peer et al 2009).
En los últimos 50 años, se ha discutido sobre que posibles mecanismos hayan permitido a nuestro ancestro común primordial aumentar su material genético y desarrollar los componentes moleculares responsables de la diversidad de las especies actuales. Sobre esto, Susumo Ohno en 1970 propuso que el genoma de los animales vertebrados fue sometido a dos rondas de duplicación total (hipótesis 2R). Es decir, un genoma ancestral deuterostomado dio lugar a dos después de la primera duplicación, y luego a cuatro genomas después de la segunda duplicación. La prueba que estos eventos sucedieron es que, en la mayoría de los casos, los genes duplicados de la misma familia están ordenados en grupos vinculados y mantienen el mismo orden de genes a pesar de encontrarse en diferentes cromosomas (Crow & Wagner, 2006; Meyer & Van de Peer, 2005).
En este contexto, Srivastava explica que «aproximadamente las tres cuartas partes de 1286 familias de genes específicas de animales surgieron por diversos procesos de duplicación en la base del filo Metazoa. Además, duplicaciones génicas adicionales, y la posterior divergencia de los eumetazoos, aumentaron aún más el número de familias de FT (el cual es de 2 a 34 veces mayor en eumetazoos que en Amphimedon)» (Srivastava et al, 2010). En vertebrados, es ampliamente aceptado que la duplicación del grupo de genes Hox es el resultado de duplicaciones de genoma completo antes de la aparición de vertebrados, gnatóstomos y peces teleósteos. El ejemplo más popular sobre la correlación putativa entre la duplicación genómica y una mayor diversidad de especies es el caso de los peces de la clase Actinopterygii; ya que las múltiples rondas de duplicación genómica por las que han atravesado han contribuido a su éxito evolutivo (Larroux et al, 2006; Larroux et al, 2008). De hecho, estos genes duplicados son el resultado de una duplicación segmentaria a gran escala antes de la radiación de los peces teleósteos, lo que respalda una "teoría de duplicación de genoma completo, específica en los peces” (duplicación 3R) (Koopman et al, 2004). En resumen, es ampliamente aceptado que todos los vertebrados han compartido dos antiguos eventos de duplicación genómica completa (1R, 2R), mientras que los peces teleósteos derivan de un linaje que experimentó una duplicación genómica adicional (3R). Esto sugiere que, a pesar de que los descendientes de un linaje que sufrió duplicación genómica no sobreviven con frecuencia; si lo hacen, su linaje evolutivo puede ser muy exitoso. Por esas razones, no se considera la duplicación genómica como un callejón sin salida, sino como un proceso que proporciona nuevas oportunidades para el éxito evolutivo (Koopman et al, 2004; Qi-long et al, 2012).
En el caso de los genes Sox, se han identificado 14 genes Sox en el cnidario Nematostella vectensis, y es probable que al menos 8 de estos genes sean resultado de duplicaciones específicas en este linaje. De la misma forma, también se han identificado 6 familias de genes Sox en los bilaterales. Dentro de los vertebrados, dichos genes se encuentran duplicados tanto en algunos anfibios, como Odorrana schmackeri, y en peces teleósteos, como Danio rerio y Takifugu rubripes. La preservación de múltiples copias de genes podría ser el resultado de la partición de subfunciones ancestrales; sin embargo, algunas de estas copias también han sufrido algunas modificaciones, como es el caso de los miembros del subgrupo SoxB en los deuterostomados, en donde perdieron sus intrones. Este grupo Sox se dividen en los subgrupos B1 y B2; y en términos de función, los genes SoxB1 actúan como activadores de la transcripción mientras que los SoxB2 generalmente desempeñan un papel como represores. Interesantemente, el subgrupo SoxB se expandió independientemente en diferentes linajes animales, como es el caso de los vertebrados donde presentan un aumento de complejidad a nivel molecular (Chau et al, 2000; Qi-long et al, 2012, Larroux et al, 2008; Popovic et al, 2009; McKimmie et al, 2005). En H. sanchiangensis, Qi-long propuso que existían dos destinos evolutivos para los genes Sox duplicados. Uno consistia en que dichos genes duplicados pasaron por un período de selección relajada que provoco una tasa evolutiva asimétrica para una de las copias, mientras que el otro sostenía que los genes duplicados experimentan restricciones de selección idénticas y no poseen mayor diversidad genética (Qi-long et al, 2012). Esto apoya que los genes duplicados pueden ser retenidos mediante un proceso de sub-funcionalización y/o relajación en ambas copias provenientes de un gen ancestral.
Neo-funcionalización y sub-funcionalización
Probablemente, en diversas etapas a lo largo de la evolución de metazoos, las secuencias que contenían cajas HMG se duplicaron, dejando una copia redundante libre de desarrollar una nueva función (neo-funcionalización) o perderse del genoma. De hecho, es posible que algunas de estas secuencias hayan reclutado dominios funcionales preexistentes y, por lo tanto, hayan formado proteínas-mosaico capaces de asumir rápidamente funciones nuevas. A diferencia de este proceso, la sub-funcionalización tiene lugar cuando ambos duplicados se mantienen debido a la división complementaria de funciones estructurales o reguladoras ancestrales entre las copias, de modo que la adición de sus funciones (a menudo la adición de sus sitios de expresión) proporciona al menos la expresión del gen pre-duplicado (Bowles et al, 2000; Force et al, 1999). Un modelo propuesto en evolución es el modelo de duplicación-degeneración-complementación (DDC), el cual predice que la probabilidad de conservación genética será mayor en genes complejos con mayor número de subfunciones, y sugiere que la partición de subfunciones ancestrales es un mecanismo importante que conduce a la preservación de múltiples copias. Esto significa que las subfunciones se mantendrían después de subsecuentes rondas de duplicación, y luego, la mayoría de las copias duplicadas se perderán durante el proceso de re-diploidización (Qi-long, et al, 2012; Chaboissier et al, 2004; Force et al, 1999).
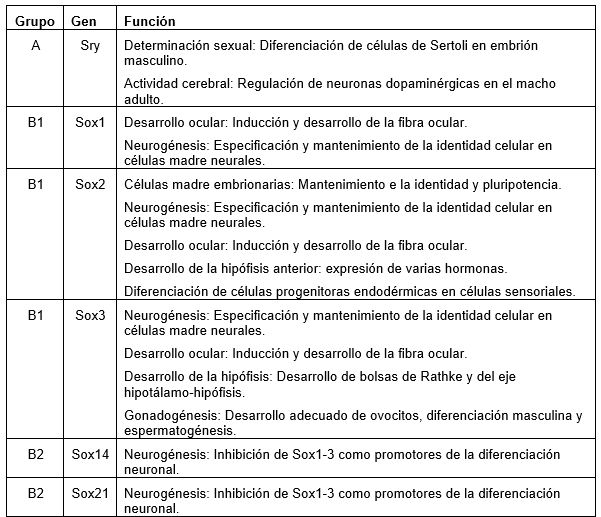
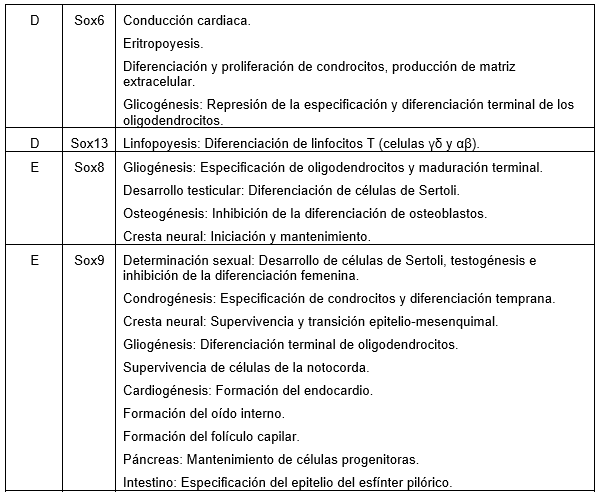
Todas las similitudes y diferencias surgidas después de los procesos de neo-funcionalización y sub-funcionalización entre los grupos de genes Sox también se manifiestan por medio de sus patrones de expresión dentro de varios tejidos. Por ejemplo, el subgrupo B está involucrado principalmente en el desarrollo neuronal (Sox1, Sox2, Sox3, Sox14) durante la diferenciación del sistema nervioso central (SNC) en animales como Drosophila, Xenopus, pollo y ratón. Por otro lado, Sox5, Sox6 y Sox9 se co-expresan durante la diferenciación de condrocitos en el hueso. Además, Sox9 es necesario y suficiente para la formación de testículos en ratones, mientras que Sox8 depende de la expresión de Sox9 para reforzar su función en la formación de testículos (Canning & Lovell-Badge, 2002; Popovic et al, 2009; Akiyama et al, 2002; Wright et al, 1995; Chaboissier et al, 2004; Berbejillo et al, 2012; Lefevbre et al, 2007) (Tabla1).
CONCLUSIONES
A pesar de toda la información presentada, no hay pistas puntuales que conduzcan a una filogenia definitivamente de la familia de genes Sox. Varios investigadores han identificado una gran cantidad de genes Sox y han intentado clasificarlos según la homología de sus cajas HMG, encontrando una relación entre su evolución y los eventos de duplicación que podrían haber tenido lugar en los primeros animales. A lo largo de esta revisión, hemos resaltado la importancia de la duplicación de genes como fuente de nuevos genes o funciones genéticas novedosas, porque este evento molecular podría haber constituido el mecanismo principal en la divergencia de los grupos Sox. En la actualidad, todavía se estudia la filogenia de esta familia de genes y la presencia de nuevos grupos de genes Sox que pueden aclarar su historia evolutiva. Todas las proteínas SOX presentan un dominio HMG que está altamente conservado, esto se debe a la función de unión al ADN, que es esencial para su rol como factor de transcripción. Los genes Sox podrían haber surgido en los primeros metazoos, después de una duplicación completa del genoma y duplicaciones en tándem específicas.
Tabla1: Funciones de los genes Sox (adaptado a partir de Lefebvre et al 2007; Schepers et al, 2002)


REFERENCIAS BIBLIOGRÁFICAS
1. Akiyama, H., Chaboissier, M.-C., Martin, J. F., Schedl, A., & de Crombrugghe, B. (2002). The transcription factor Sox9 has essential roles in successive steps of the chondrocyte differentiation pathway and is required for expression of Sox5 and Sox6. Genes & Development, 16(21), 2813–2828. https://doi.org/10.1101/gad.1017802
2. Argentaro, A., Olsson, J., Critcher, R., McDowall, S. G., & Harley, V. R. (2000). Genomic characterisation and fine mapping of the human SOX13 gene. Gene, 250(1–2), 181–189. https://doi.org/10.1016/s0378-1119(00)00157-8
3. Berbejillo, J., Martinez-Bengochea, A., Bedo, G., Brunet, F., Volff, J.-N., & Vizziano-Cantonnet, D. (2012). Expression and phylogeny of candidate genes for sex differentiation in a primitive fish species, the Siberian sturgeon, Acipenser baerii. Molecular Reproduction and Development, 79(8), 504–516. https://doi.org/10.1002/mrd.22053
4. Bowles, J., Schepers, G., & Koopman, P. (2000). Phylogeny of the SOX family of developmental transcription factors based on sequence and structural indicators. Developmental Biology, 227(2), 239–255. https://doi.org/10.1006/dbio.2000.9883
5. Breitling, R., & Gerber, J.-K. (2000). Origin of the paired domain. Development Genes and Evolution, 210(12), 644–650. https://doi.org/10.1007/s004270000106
6. Canning, C. A., & Lovell-Badge, R. (2002). Sry and sex determination: how lazy can it be? Trends in Genetics, 18(3), 111–113. https://doi.org/10.1016/S0168-9525(01)02615-4
7. Chaboissier, M.-C., Kobayashi, A., Vidal, V. I. P., Lützkendorf, S., van de Kant, H. J. G., Wegner, M., … Schedl, A. (2004). Functional analysis of Sox8 and Sox9 during sex determination in the mouse. Development (Cambridge, England), 131(9), 1891–1901. https://doi.org/10.1242/dev.01087
8. Chau, K. Y., Munshi, N., Keane-Myers, A., Cheung-Chau, K. W., Tai, A. K., Manfioletti, G., … Ono, S. J. (2000). The architectural transcription factor high mobility group I(Y) participates in photoreceptor-specific gene expression. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience, 20(19), 7317–7324.
9. Cock, J. Mark, Kristin Tessmar-Raible, Catherine Boyen & Frédérique Viard (2010). Introduction to Marine Genomics. Springer (PDF) . DOI: 10.1007/978-90-481-8639-6
10. Cremazy, F., Soullier, S., Berta, P., & Jay, P. (1998). Further complexity of the human SOX gene family revealed by the combined use of highly degenerate primers and nested PCR. FEBS Letters, 438(3), 311–314. https://doi.org/10.1016/S0014-5793(98)01294-0
11. Crow, K. D., & Wagner, G. P. (2006). What Is the Role of Genome Duplication in the Evolution of Complexity and Diversity? Molecular Biology and Evolution, 23(5), 887–892. https://doi.org/10.1093/molbev/msj083
12. Degnan, B. M., Vervoort, M., Larroux, C., & Richards, G. S. (2009). Early evolution of metazoan transcription factors. Current Opinion in Genetics & Development, 19(6), 591–599. https://doi.org/10.1016/j.gde.2009.09.008
13. Force, A., Lynch, M., Pickett, F. B., Amores, A., Yan, Y. L., & Postlethwait, J. (1999). Preservation of duplicate genes by complementary, degenerative mutations. Genetics, 151(4), 1531–1545.
14. Kaessmann, H. (2010). Origins, evolution, and phenotypic impact of new genes. Genome Research, 20(10), 1313–1326. https://doi.org/10.1101/gr.101386.109
15. King, N., Westbrook, M. J., Young, S. L., Kuo, A., Abedin, M., Chapman, J., … Rokhsar, D. (2008). The genome of the choanoflagellate Monosiga brevicollis and the origin of metazoans. Nature, 451(7180), 783–788. https://doi.org/10.1038/nature06617
16. Koopman, P., Schepers, G., Brenner, S., & Venkatesh, B. (2004). Origin and diversity of the SOX transcription factor gene family: genome-wide analysis in Fugu rubripes. Gene, 328, 177–186. https://doi.org/10.1016/j.gene.2003.12.008
17. Larroux, C., Fahey, B., Liubicich, D., Hinman, V. F., Gauthier, M., Gongora, M., … Degnan, B. M. (2006). Developmental expression of transcription factor genes in a demosponge: insights into the origin of metazoan multicellularity. Evolution & Development, 8(2), 150–173. https://doi.org/10.1111/j.1525-142X.2006.00086.x
18. Larroux, C., Luke, G. N., Koopman, P., Rokhsar, D. S., Shimeld, S. M., & Degnan, B. M. (2008). Genesis and expansion of metazoan transcription factor gene classes. Molecular Biology and Evolution, 25(5), 980–996. https://doi.org/10.1093/molbev/msn047
19. Lefebvre, V., Dumitriu, B., Penzo-Méndez, A., Han, Y., & Pallavi, B. (2007). Control of cell fate and differentiation by Sry-related high-mobility-group box (Sox) transcription factors. The International Journal of Biochemistry & Cell Biology, 39(12), 2195–2214. https://doi.org/10.1016/j.biocel.2007.05.019
20. McKimmie, C., Woerfel, G., & Russell, S. (2005). Conserved genomic organisation of Group B Sox genes in insects. BMC Genetics, 6, 26. https://doi.org/10.1186/1471-2156-6-26
21. Meyer, A., & Van de Peer, Y. (2005). From 2R to 3R: evidence for a fish-specific genome duplication (FSGD). BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology, 27(9), 937–945. https://doi.org/10.1002/bies.20293
22. Peer, Y. V. de, Maere, S., & Meyer, A. (2009). The evolutionary significance of ancient genome duplications. Nature Reviews Genetics, 10(10), 725–732. https://doi.org/10.1038/nrg2600
23. Popovic, J., & Stevanovic, M. (2009). Remarkable evolutionary conservation of SOX14 orthologues. Journal of Genetics, 88(1), 15–24. https://doi.org/10.1007/s12041-009-0003-4
24. Qi-long, C., Zi-jun, Q., #, Jian#, C., Sheng#, H. U., Hui, Z., … Wen-li, M. A. (2012). Isolation and sequencing of the HMG domains of fifteen Sox genes from Hyla sanchiangensis, and analysis of the evolutionary behaviors of Sox duplicated copies based on bioinformatics. African Journal of Microbiology Research. https://doi.org/10.5897/AJMR12.300
25. Schepers, G. E., Teasdale, R. D., & Koopman, P. (2002). Twenty pairs of sox: extent, homology, and nomenclature of the mouse and human sox transcription factor gene families. Developmental Cell, 3(2), 167–170. https://doi.org/10.1016/s1534-5807(02)00223-x
26. Srivastava, M., Simakov, O., Chapman, J., Fahey, B., Gauthier, M. E. A., Mitros, T., … Rokhsar, D. S. (2010). The Amphimedon queenslandica genome and the evolution of animal complexity. Nature, 466(7307), 720–726. https://doi.org/10.1038/nature09201
27. Voldoire, E. (2013). Duplication de génome et évolution de la famille Sox chez les poissons téléostéens (Phd thesis, Ecole normale supérieure de lyon - ENS LYON). Retrieved from https://tel.archives-ouvertes.fr/tel-01124192/document
28. Wegner, M. (1999). From head to toes: the multiple facets of Sox proteins. Nucleic Acids Research, 27(6), 1409–1420. https://doi.org/10.1093/nar/27.6.1409
29. Wright, E., Hargrave, M. R., Christiansen, J., Cooper, L., Kun, J., Evans, T., … Koopman, P. (1995). The Sry-related gene Sox9 is expressed during chondrogenesis in mouse embryos. Nature Genetics, 9(1), 15–20. https://doi.org/10.1038/ng0195-15