Resistencia extrema al cobre por un consorcio bacteriano conformado por Sulfobacillus spp. y Acidithiobacillus ferrooxidans
Extreme resistance to copper by a microbial consortium formed from Sulfobacillus spp., and Acidithiobacillus ferrooxidans
*Omar D. Pairazamán-Quiroz 1, José E. Neciosup-Quesñay 2, Yeni S. Valdez-Anco 3, Fabián A. Veliz-De La Vega 4, Paola J. Barrios-Cuevas 4, Christopher Araya-Rozas 4, Heber M. Robles-Castillo 5, Mario R. Esparza-Mantilla 4
1 Universidad Nacional de Cajamarca, Facultad de Ciencias de la Salud, Laboratorio de Microbiología. Cajamarca – Perú
2 Universidad Nacional Pedro Ruiz Gallo, Facultad de Ciencias Biológicas
3 Universidad Nacional Jorge Basadre Grohmann, Facultad de Ciencias Biológicas
4 Universidad de Antofagasta, Laboratorio de Biominería
5 Universidad Nacional de Trujillo. Laboratorio de Biotecnología
*Autor de correspondencia: opq2507@hotmail.com
Recibido: 26 de Marzo, 2019. Aceptado: 26 de Julio, 2019.
RESUMEN
La sensibilidad al cobre por parte de microorganismos biolixiviadores es uno de los principales problemas que enfrenta la minería para mejorar los procesos de biolixiviación. En tal sentido, nosotros evaluamos la resistencia al cobre de un consorcio microbiano conformado por Sulfobacillus spp., y Acidithiobacillus ferrooxidans, el cual se propagó, en bioreactores de tanque aireado y agitado de 1 L, los cuales contenían medio 9K suplementado con hierro y concentraciones crecientes de cobre (200 mM, 400 mM, 600 mM, 800 mM y 1,000 mM) a 30°C con un pH de 1.6 durante 96 horas. Se colectó una muestra de cada biorreactor cada 8 horas, para realizar análisis, microscópicos y moleculares, además el cultivo del consorcio en placa mostró una resistencia al cobre hasta 1,000 mM. Se observó que el consorcio microbiano puede crecer en presencia de cobre tanto en medio líquidos (biorreactores-9K+Cu: 200 mM-800mM) como en medios sólidos (agarosa-9K + Cu: 1,000mM). Nuestros resultados proponen que el consorcio conformado por Sulfobacillus spp., y Acidithiobacillus ferrooxidans, presenta resistencia extrema al cobre y podría ser aplicado bajo condiciones de mayor escalamiento en procesos de biolixiviación de minerales sulfuros donde el cobre se encuentra en concentraciones de hasta 1,000 mM.
Palabras clave: Acidithiobacillus ferrooxidans; consorcio microbiano; microorganismos biolixiviadores; Sulfobacillus spp, resistencia al cobre
ABSTRACT
The sensitivity to copper by bioleaching microorganism is one of the main problems faced by mining to improve bioleaching processes. In this sense, we evaluated the resistance to copper of a microbial consortium formed by Sulfobacillus spp., and Acidithiobacillus ferroxidans which was propagated, in aerated and agitated tank bioreactors of 1 L, which contained 9 K medium supplemented with iron and concentrations copper (200 mM, 400 mM, 600 mM, 800 mM and 1,000 mM) at 30 °C with a pH of 1.6 for 96 hours. A samples of each bioreactor was colleted every 8 hours, to perform cytometric microscope and molecular analysis, and plate count test showed a resistance to copper at 1,000 mM. It was observed that the microbial consortium can grow in the presence of copper both in liquid medium (bioreactors -9K + Cu: 200 mM – 800 mM) and in solid media (agarose – 9K + Cu: 1,000 mM). Our results propose that the consortium formed Sulfobacillus spp., and Acidithiobacillus ferrooxidans presents wxtreme resistance to copper and could be applied under higher scaling conditions in bioleaching processes of sulfide minerals where copper is found in concentrations up to 1 000 mM.
Keywords: Acidithiobacillus ferrooxidas; bioleaching microorganisms; microbial consortium; Sulfobacillus spp; resistance to copper.
1. INTRODUCCIÓN
Los microorganismos extremoacidófilos viven en condiciones que no son comunes para la mayoría de los seres vivos (pH < 2), donde están expuestos a la oxidación por metales pesados (Madigan et al., 2019). Dichos microorganismos pertenecen a especies de bacterias, arqueas y hongos, e incluso algunas protistas, las cuales se caracterizan por tener metabolismos quimiolitoautótrofos y quimiolito-organótrofos, además de ser aerobias y mesófilas, o bien termófilas (Madigan et al., 2019; Medina et al., 2012).
La biolixiviación se puede definir como un proceso natural de solubilización de metales, por disgregación de los minerales, generada por el ataque oxidante de moléculas liberadas por el metabolismo del hierro y el azufre en las bacterias extremoacidófilas (Watling, 2006). Este bioproceso ya es usado en países como Chile, donde la biominería es una estrategia eficiente frente a los métodos convencionales, utilizando cepas modificadas genéticamente para incrementar los rendimientos de extracción (Latorre et al., 2016).
En la biolixiviación del cobre, se disponen pilas de minerales, las cuales contienen sulfuros secundarios, donde se adicionan soluciones ácidas (Watling, 2006). Estas soluciones contienen diferentes concentraciones de cobre ya que constantemente están bañando las pilas, por lo que suelen clasificarse de tres formas: la solución rica en cobre o también llamada PLS (Pregnant Leach Solution), la solución intermedia de cobre o también llamada ILS (Intermediate leaching solution) y la solución de refino que contiene una baja concentración de cobre, debido a qué se obtiene después de la extracción del cobre (Morales, 2016).
Las soluciones PLS contienen alrededor de 100 mM de cobre, mientras que en las soluciones de refino se pueden encontrar concentraciones aproximadas a 19.2 mM de cobre (Morales, 2016); además, otras soluciones de biolixiviado presentan concentraciones en torno a 30.5 a 40 mM de cobre (Jia et al., 2016). Estas concentraciones son letales para procariotas no extremoacidófilas, porque el cobre tiene características antimicrobianas (Neciosup et al., 2015); sin embargo, empleando herramientas metagenómicas se han reportado la existencia de comunidades microbianas (Xie et al., 2007). Según Watling et al. (2014), bacterias de los géneros Acidithiobacillus, Leptospirillum, Ferriphilum y Acidiphilum, además de arqueas como Ferroplasma han sido identificadas en soluciones ILS (Demergasso et al., 2005). Según Jia et al. (2016), las soluciones PLS e ILS, con presencia de microorganismos, muestran una mejor oxidación del hierro en relación con la solución de refino, lo cual indica un metabolismo muy activo a pesar de las concentraciones de cobre.
Bacterias como Acidithiobacillus ferrooxidans son utilizadas en procesos de biolixiviación por su capacidad de crecer en presencia de cobre (Demergasso et al., 2005; Harvey y Crundwell, 1996; Rawlings y Johnson 2007), además de que subespecies de Sulfobacillus thermosulfidooxidans han sido reportadas como tolerantes a concentraciones elevadas de cobre (Watkin et al., 2009), pero su comportamiento como consorcio microbiano en respuesta al cobre no ha sido establecido.
Uno de los grandes desafíos es hacer constante la biolixiviación a escala industrial, y que las comunidades microbianas establecidas en las soluciones, obtengan resistencia al cobre. En este rumbo, esta investigación evaluó la resistencia de un consorcio microbiano expuesto a elevadas concentraciones de cobre como un modelo microbiológico de alta resistencia al cobre con potencial aplicación en la biolixiviación.
2. MATERIALES Y MÉTODOS.
2.1. Aislamiento, caracterización y propagación del consorcio microbiano.
El consorcio microbiano constituido por Sulfobacillus spp., y Acidithiobacillus ferrooxidans, se aisló desde un drenaje acido de mina (pH: 3.0) en el distrito de Sayapullo, (La Libertad, Perú), cuyas coordenadas son 7º35´34.46´´ S, 78º29´00.46´´ O, elevación: 2206 m.s.n.m. Este consorcio se mantuvo activo por repiques constantes en medio 9K-Fe hasta obtener un inoculo de 1.8 x 108 células/ml, usando recuento en cámara de neubauer.
2.2. Acondicionamiento y bioproceso
Cinco biorreactores (figura 1) tanque aireado agitado de 1.5 L (Miranda et al., 2006), contenían 250 mL del inóculo bacteriano (1 x 108 células / mL), 180 mL de medio 9K (10X) (Yates y Holmes (1987)), 75 mL de solución de Fe2SO4. 7H2O (10X), 270 mL de agua destilada estéril y 230 mL de CuSO4. 5H2O (utilizado para los biorreactores con sulfato de cobre y para el control fue reemplazado con agua destilada estéril) alcanzando un volumen total de 1 L. La solución de sulfato de cobre utilizada, fue preparada a partir de una solución madre (250 g / L) alcanzando una concentración de 880 mM de cobre (valor confirmado por espectrofotometría de absorción atómica), llevando a los 4 biorreactores a una concentración base de 200 mM de cobre, a partir de allí se adicionó sulfato de cobre en polvo, previos cálculos estequiométricos (59. 20 g, 118.50 g y 177.82 g) para alcanzar las concentraciones deseadas de 400, 600 y 800 mM de cobre. El H2SO4 se utilizó para reducir el pH hasta 1.5. Los 6 biorreactores fueron incubados a 30 ºC +/- 2ºC con aireación de 0.05 vvm, 180 rpm por 96 horas. El ensayo se realizó por triplicado.
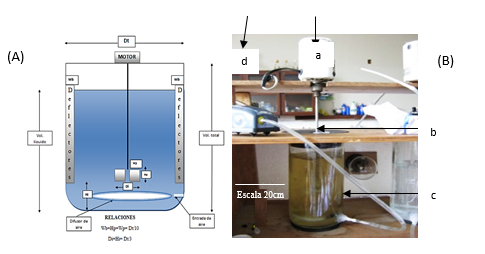
Figura 1. Biorreactor tanque aireado y agitado (Miranda et al., 2006), (A) diseño de biorreactor. (B) Biorreactor usado para bioproceso de resistencia al cobre, donde (a) motor a 180rpm, (b) eje en el que se encuentra adosada la turbina, (c) manguera 0.25mm diámetro, (d) bomba de aire (0.05vvm). Biorreactor diseñado para 1.5 L de capacidad.
2.3 Análisis y monitoreo del bioproceso.
Cada 8 horas hasta completar 96 horas se colectaron muestras de 1.0 ml por cada biorreactor y se analizó el número total de células en cámara de Neubauer (Madigan et al., 2019), se determinó la concentración de cobre al inicio y al final del bioproceso mediante espectrofotometría de absorción atómica (Allan, 1961).
Los datos del recuento celular se procesaron por análisis de varianza con un nivel de significancia de 95% y se determinó el grado de significancia entre el consorcio sin exposición (control) y el consorcio expuesto a concentraciones de 200, 400, 600 y 800 mM de cobre; además de determinar el grado de significancia entre el consorcio expuesto a 200, 400, 600 y 800 mM de cobre utilizando la prueba HSD de Tukey con el software SPSS v. 15.
2.4 Reactivación del consorcio y cultivo.
Al término del bioproceso, fueron tomados, 2.5 mL del contenido de cada biorreactor y se inoculó en un matraz de 500 mL, conteniendo 100 mL del medio 9K-Fe (Schrader y Holmes, 1988), incubándose en agitación constante de 120 rpm por 18 días a temperatura ambiente. Al término de este tiempo se concentraron por centrifugación a 8000 rpm, las biomasas celulares de 10 mL de cada consorcio, de estos concentrados fueron tomados 20 µL y adicionados sobre la superficie del medio 9K – Fe / Cu 1 000 mM /agarosa en placas de Petri siguiendo el protocolo de Yates y Holmes (1987).
2.5 Extracción de ADN genómico de los microorganismos resistentes al cobre.
Un inóculo de 100 mL (1 x 108 células / mL) obtenido del biorreactor de 800 mM de cobre, al término de los 18 días de reactivación, se sometió al sistema de filtración al vacío usando un filtro de 0.2 µm, dicho inóculo se lavó con 10 mL de agua HPLC libre de nucleasas a pH 2. La suspensión bacteriana resultante del lavado se colocó en tubos microtubos (1.5 ml) y se centrifugó a 10,000 rpm por 5 minutos. Este proceso se repitió dos veces y la biomasa bacteriana final se usó para la extracción del ADN total. Para ello, se utilizó el kit de extracción PowerSoil DNA Isolation Kit, MoBio Lac. Inc siguiendo las instrucciones del proveedor.
2.6 Amplificación del gen ARNr 16S.
El ADN total se usó como templado para amplificar por PCR la región del gen que codifica para el ARNr 16S, para ello se utilizó los oligonucleótidos 27-F (5´d AGAGTTTGATCMTGGCTCAG3´) y el 1492-R (5´d TACGGYTACCTTGTTACGACTT 3´) (Lane, 1991). La reacción de PCR tuvo, 8 µL de Power Mix; 2 µL ADN genómico a una concentración de 50 ng/µL. El Power Mix se formuló por 0.5 µL Green Buffer 5X, 13.5 µL de agua bidestilada estéril, 3.2 µL MgCl2 (50 mM), 0.6 µL de 27-F (10 mM), 0.6 µL de 1492-R (10 mM), 0.5 µL dNTPs (10 mM); y 0.02 µL de Taq Polimerasa (2.5 U). Las condiciones del PCR fueron: una desnaturalización a 95°C durante 5.00 minutos, una hibridación a 57°C durante 45 segundos, una extensión a 72°C durante 45 segundos, lo cual se repitió durante 36 ciclos, además de una amplificación final a 72 °C durante 5 minutos para finalmente almacenarse a 4 °C. El producto de PCR fue resuelto en un gel de agarosa al 1%, 100V por 30 minutos. Como marcador de peso molecular se utilizó 1 kb plus DNA Ladder (Promega®) y como revelador bromuro de etidio.
2.7 PCR Multiplex para microorganismos biolixiviantes.
Se realizó un PCR multiplex (De Wulf-Durand et al., 1997) utilizando los oligonucleótidos 170-F (5´-CAATCCCGCATACGTTCC-3´) y 606-R (5´-AAACCGCTACGTATCGCAC-3´) para detectar a Sulfobacillus sp., además de 458-F (5-GGGTGCTAATAWCGCCTGCTG-3´) y 1492-R (5´-TACGGYTACCTTGTTACGACTT-3´) para detectar a A. ferrooxidans. Para el PCR multiplex, se preparó el master mix, conteniendo: 603 µL de agua bidestilada estéril, 50 µL MgCl (50 mM), 250 µL buffer green 5X. El contenido del Master mix se dividió en dos microtubos de 1.5 mL de capacidad. El Mix taq contenía 250 uL de master mix más 7 uL de Taq ADN polimerasa. Por último el power mix contenía 653 uL de master mix y se le añadió 25 uL de dNTPs (10 Mm), además de 20 uL de cada oligonucleótido. La distribución en los tubos de PCR fue de 18.5µL de power mix, 1.5 µL de ADN templado (50 ng/ µL) y 5.0 µL de Mix Taq.
Las condiciones del PCR multiplex fueron: Una desnaturalización inicial a 94 °C durante 3 minutos, una amplificación a 72 °C durante 1 minuto, subsecuentemente, se realizó una amplificación final a 72 °C durante 7 minutos, para finalmente almacenarse a 4 °C. El producto de PCR fue resuelto en un gel de agarosa al 1 %, 100 V por 30 minutos. Como marcador de peso molecular se utilizó 1 kb plus DNA Ladder (Promega®). Los geles se revelaron con GelRed® Nucleic Acid Gel Stain (BIOTIUM).
3. RESULTADOS
3.1 Efecto del cobre sobre el crecimiento microbiano
En las primeras 8 horas post-inoculación se observó una disminución del número de células en los grupos con presencia de cobre. A partir de allí, hasta el final del bioproceso, el número de células varió en un rango de 7.87 x 107 hasta 6.65 x 107 (200 mM), de 6.51 x 107 hasta 5.40 x 107 (400 mM), 5.46 x 107 hasta 4.60 x 107(600 mM), 4.43 x 107 hasta 3.50 x 107 (800 mM). El grupo sin exposición al cobre aumentó su concentración celular durante las 96 horas (figura 2). El análisis de varianza ANOVA permitió determinar que existieron diferencias significativas entre el crecimiento del consorcio sin exposición al cobre y el consorcio expuesto a las concentraciones de 200, 400, 600 y 800 mM de cobre (P < 0.05). Para determinar si existe diferencia significativa entre los grupos expuestos al cobre se aplicó el análisis Tukey, determinándose que existe diferencia entre los grupos expuestos a 200 y 400 mM de cobre con los grupos 600 y 800 mM de cobre (p < 0,05); además se determinó que la velocidad específica de crecimiento del consorcio sin exposición al cobre fue 1.82 x 10-2 h-1.
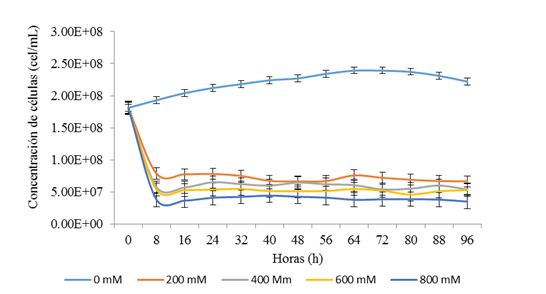
Figura 2. Crecimiento de un consorcio microbiano resistente al cobre conformado por Acidithiobacillus ferrooxidans y Sulfobacillus spp, expuesto a diferentes concentraciones de sulfato de cobre, durante 96 horas de incubación a 30°C +/- 2°C, en medio 9K Fe a pH 1,6, en condiciones de laboratorio.
3.2 Bioproceso y análisis microbiológicos.
En la figura 3, se muestran las imágenes de los biorreactores en el bioproceso y las características morfológicas de los microorganismos durante la exposición al cobre usando la tinción de Gram. Se observan bacterias con forma de bacilo y cocobacilos. La mayoría de células son Gram negativas, siendo visible en los grupos de 600 y 800 mM de cobre. Esta observación se realizó de los cultivos propagados terminada la experiencia.

Figura 3. Observación del bioproceso y análisis microbiológico en diferentes concentraciones de cobre. Todos los biorreactores tanque aireado y agitado contienen medio 9K-Fe a pH 1,6 agitados a 180 RPM, donde cada Biorreactor contiene el consorcio microbiano (A): sin cobre, (B):con 200 mM de cobre (12,7 g / L Cu), , (C): con 400 mM de cobre (25,4 g / L de Cu), ( D): con 600 mM de cobre (38,1 g /L de Cu),. (E): con 800 mM de cobre (50,8 g / L de Cu). En las microfotografías de la columna derecha se observa células bacterias teñidas con tinción de Gram, correspondientes a cada biorrector (A-E)
3.3 Electroforesis en geles de agarosa.
En la figura 4A, se muestra la amplificación de las secuencias del gen ARNr 16S para el dominio bacteria (1,500 pb). Este resultado indica la presencia de microorganismos del dominio bacteria, en los cuatro grupos del consorcio expuesto al cobre. En la figura 4B, se muestran tres bandas que corresponden a los amplificados de los oligonucleótidos para A. ferrooxidans y Sulfobacillus spp., con pesos moleculares de 1,000 pb y 500 pb, respectivamente. En el carril 2 y 3 se detectó la presencia de A. ferrooxidans y en el carril 3 solo Sulfobacillus sp.
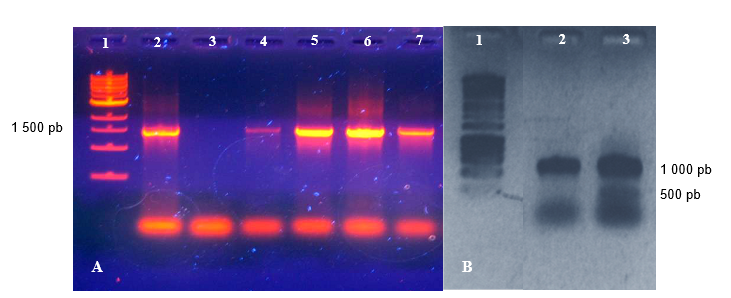
Figura 4. Electroforesis en gel de agarosa al 1%, para confirmar la presencia de bacterias extremoacidófilas. A: Productos de PCR para el dominio bacteria teñidos con Gel red (Biotium), utilizando oligonucleótidos 27-F, 1492-R; donde el carril 1 corresponde al marcador de peso molecular 1kb; carril 2, ADN de A. ferrooxidans (control positivo); carril 3 agua bidestilada estéril (control negativo); en los carriles 4, 5, 6 y 7 corresponden al consorcio expuesto a 200, 400, 600 y 800 mM de cobre. B: Productos de PCR multiplex teñidos con bromuro de etidio, donde el carril 1 es el marcador de peso molecular 1kb; en el carril 2 y 3 se observa la amplificación del gen ARNr de A. ferrooxidans y en el carril 3 se observa la amplificación de ARNr de Sulfobacillus sp.
3.4 Resistencia extrema al cobre (1,000 mM) en microorganismos extremoacidófilos
En la figura 5 se muestra el cultivo in vitro del consorcio que desarrolló en presencia de 1,000 mM de cobre en diferentes tiempos de crecimiento. La colonia mixta de bacterias Gram positivas y Gram negativas es convexa de color café claro y más oscuro en el centro, de borde redondeado. Además se muestra que la morfología celular bacteriana es preferentemente bacilar y cocobacilar.
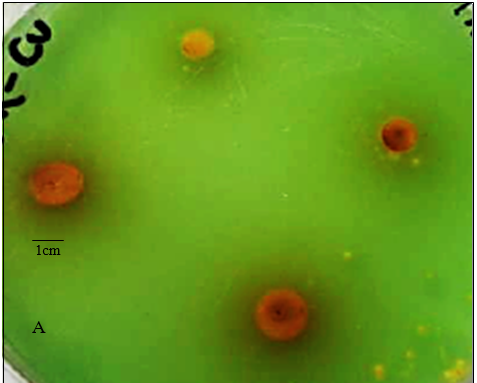

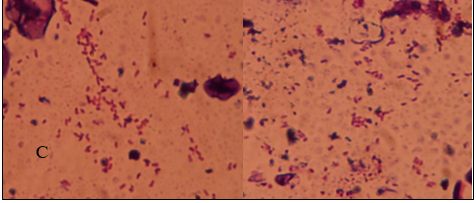
Figura 5. Análisis microbiológico del consorcio. A: Colonias resistentes a 1000 mM de cobre al cabo de 16 días de incubación, usando la técnica de siembra de microgota. B: vista panorámica de las colonias de color café oscuro en el centro y naranja oscuro en el borde; además se observa una zona oscura alrededor de la colonia. C: Análisis microscópico del consorcio, se observan a inmersión bacterias Gram positivas y Gram negativas.
4. DISCUSIÓN
La resistencia de los microorganismos al cobre, puede deberse a la activación de genes específicos mediante la regulación positiva que ejerce el cobre, para la traducción de proteínas transportadores como ATPasas tipo P (Dupont et al., 2010) y proteínas relacionadas a la resistencia, nodulación y división celular (RND) que pueden bombear el cobre desde el citoplasma al periplasma y luego hacia espacio extracelular (Li et al., 2015). Una característica notoria del consorcio bacteriano, es que la presencia de bacterias Gram negativas, explica uno de los mecanismos involucrados en la resistencia extrema al cobre, debido a la presencia de una membrana externa con mayor número de proteínas (RND) favoreciendo los sistemas de expulsión del cobre. Este mecanismo no está presente en la bacterias Gram positivas (Saier, 2003); además, existe evidencia que las bacterias utilizan polisacáridos como medida de protección frente a las altas concentraciones de metales pesados (Jachlewski et al., 2015).
La concentración de células en el consorcio, se ve disminuida por el efecto del cobre en las primeras 8 horas (Figura 2) y a partir de allí el consorcio comienza a mantenerse en una concentración celular constante. Esta acción del cobre sobre el crecimiento bacteriano fue investigada por Neciosup et al. (2015) al evaluar el crecimiento de bacterias patógenas intrahospitalarias en superficies de acero y cobre, siendo el cobre letal para Pseudomonas aeruginosa, Escherichia coli y Staphylococcus aureus, bacterias que son las principales causantes de infecciones intrahospitalarias y generalmente resistentes a antibióticos. Sin embargo las bacterias presentes en el consorcio crecen en ambientes ácidos con presencia de metales pesados y tienen mecanismos moleculares intrínsecos de resistencia que le permiten tolerar y mantenerse viables a pesar del estrés oxidativo que sufre la membrana celular, las proteínas y el ácido nucleico (San Miguel y Martín, 2009; Castillo et al., 2017).
La velocidad de crecimiento del consorcio sin exposición al cobre (1.82 x 10-2 h-1) en relación a la experiencia de Cabrera et al. (2005) fue menor tanto para el cultivo puro de Thiobacillus ferroxidans (8.6 x 10-2 células/ mL) y el cultivo mixto de Thiobacillus ferrooxidans e Acidiphilum sp (8.7 x 10-2 células/mL). Esta diferencia podría deberse a que nuestro trabajo utilizó 5 veces mayor volumen que la realizada por Cabrera et al. (2005) y que la toma de muestra en el biorreactor (1 L) podría no ser homogénea siendo una debilidad de la técnica de recuento celular usando la cámara de Neubauer, con volúmenes a mayor escala. Además este autor señala que la velocidad de crecimiento en presencia de cobre con una sola especie (Thiobacillus ferrooxidans) se ve más afectada que cuando se encuentra asociada (Thiobacillus ferrooxidans y Acidiphilium sp), por lo que la resistencia a mayores concentraciones de cobre estaría asociada a los mecanismos de interacción entre los integrantes del consorcio y sería una de las explicaciones por las cuales el consorcio es resistente a 800 mM de cobre en biorreactor de 1 L y a 1000 mM de cobre in vitro. Con relación a los grupos expuestos al cobre, existe un descenso en el número de células de forma gradual, sin embargo, se evidencia la resistencia del consorcio al mantener un número elevado de células durante 96 horas de bioproceso.
Después de reactivar el consorcio, se observaron microorganismos con morfología bacilar y cocobacilar, luego se realizó una tinción de Gram observándose presencia de bacterias Gram negativas en mayor proporción (figura 3). El análisis de PCR para el ARNr 16S específico para el dominio bacteria, es positivo en todos los grupos expuestos al cobre (figura 4), por ese motivo se afirma que todas las bacterias presentes en el consorcio en la etapa de reactivación, son resistentes a las diferentes concentraciones de cobre expuestas; además, en la figura 5, se muestra el resultado de la electroforesis de los productos de PCR multiplex detectando la presencia de, A. ferrooxidans y Sulfobacillus sp.
Estos resultados toman mayor solidez al enfocarse en A. ferrooxidans, la bacteria más estudiada en biominería ya que presenta por lo menos 10 genes relacionados con la homeostasis del cobre (Nies et al., 2003); además, la fase de latencia encontrada en nuestro trabajo (figura 2), puede ser explicada por la experiencia de Natarajan et al. (1994), cuando expuso a A. ferrooxidans a 314 mM de cobre, señalándose que las células podrían realizar algunos reajustes de material celular asociada con la membrana celular; sin embargo es preciso señalar que A. ferrooxidans adquiere resistencia cuando es expuesta consecutivamente al cobre (Navarro, 2009).
Según la literatura se conoce que el género Sulfobacillus tiene una especie S. thermosulfidooxidans que presenta resistencia al cobre hasta 786 mM (Watkin et al., 2009); sin embargo en esta investigación la cepa Sulfobacillus spp., presente en el consorcio es resistente hasta 1000 mM de cobre. Dentro de las especies de este género están S. thermosulfidooxidans, que es más activa a la oxidación de del Fe+2 y de sulfuros minerales, en comparación con S. acidophilus, que es más eficiente con la oxidación del azufre (Rodríguez et al., 2001), bajo esta perspectiva habrían pocas posibilidades que la especie de Sulfobacillus presente en el consorcio sea justamente ésta última. Cabe indicar que Demergasso et al. (2005), señaló que estas dos bacterias forman parte de la diversidad microbiana que se encuentran en la naturaleza en diferentes etapas de los procesos de biolixiviación, sumado a su alta resistencia al cobre hace que sea muy probable identificarlos luego de nuestra experiencia. Por lo tanto uno de los aportes de nuestro trabajo es confirmar que las bacterias A. ferrooxidans y Sulfobacillus spp., son resistentes al cobre entre 800 a 1,000 mM (figuras 4 y 5).
Se demostró que el PCR multiplex puede ser utilizado para la detección de microorganismos biolixiviantes, que generalmente se encuentran en medios extremos como aguas ácidas, relaves mineros, con elevadas concentraciones de metales pesados y también señalar que en el proceso de evaluación de este consorcio las bacterias estuvieron en soluciones elevadas de cobre, por ese motivo la técnica de PCR multiplex y el proceso de purificar ADN bacteriano libre de cobre para el PCR es reproducible para ser usada con microorganismos extremoacidófilos (Figura 4). Por tanto, la técnica de PCR multiplex en esta investigación debe ser aplicada como indicador de la presencia de bacterias resistentes al cobre, lo cual constituye una herramienta importante para evaluar bioprocesos en la industria minera, ambientes extremos como pasivos ambientales, drenajes ácidos así como durante los procesos de biolixiviación.
La generación de un cultivo mixto en medio 9K con hierro y cobre al 1,000 mM, en estado sólido, brinda un modelo para la detección, comparación y estudio de consorcios microbianos biolixiviantes resistentes al cobre, así como para el estudio in situ de las relaciones ecológicas entre dos o más procariotas frente a metales pesados. Una aplicación biotecnológica que surge como consecuencia de este cultivo mixto en estado sólido es la obtención de biomasa con características de resistencia al cobre y con metabolismo activo del hierro para su aplicación en tareas de biolixiviación. La ventaja de tener un cultivo en estado sólido es que se evita la formulación y preparación de grandes cantidades de medio líquido, reduciéndose los costos de insumos como agua, ácido sulfúrico, hierro y otras sales, así como, los costos de energía para el suministro de oxígeno y el mantenimiento de la temperatura, teniendo como consecuencia las reducción de los espacios útiles en el laboratorio y los costos de biomasa útil.
Para finalizar esta investigación, se demostró que los integrantes del consorcio conformado por A. ferrooxidans y Sulfobacillus sp, pueden permanecer viables a concentraciones de 800 mM de cobre en biorreactor de 1 L y 1,000 mM de cobre en medio de cultivo sólido, estos resultados nos permiten generar un modelo para el aprovechamiento de biotecnológico sobre estos microorganismos y poder seleccionar bacterias de acuerdo a su resistencia al cobre (figura 6). Además al contar con un grupo de bacterias adaptadas a elevadas concentraciones de cobre, se desprende su aprovechamiento a gran escala; es decir, su utilización en grandes volúmenes para el riego constante de soluciones ácidas en las pilas de biolixiviación, lográndose recuperar estos microorganismos a partir de las soluciones de PLS y Refino para luego volverlos a propagar de forma constante (figura 7).
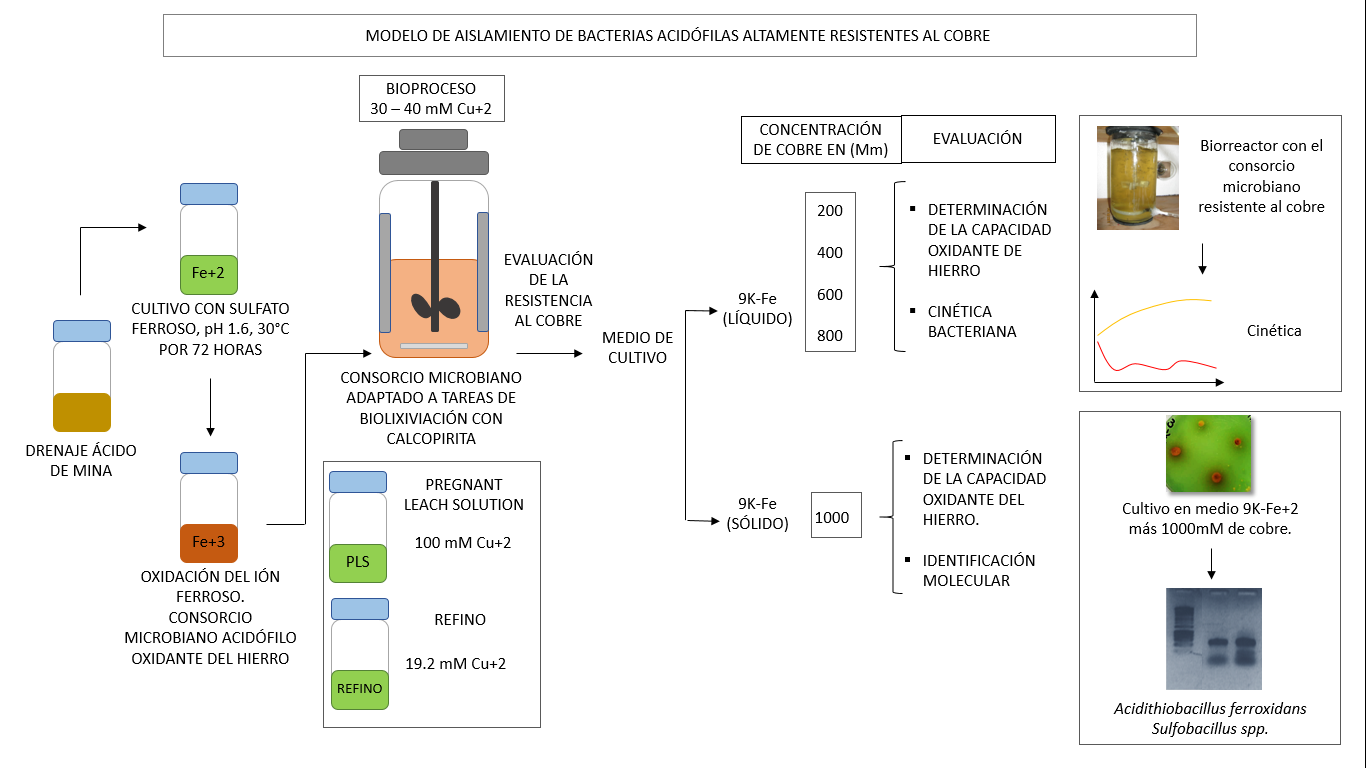
Figura 6. Modelo de aislamiento de bacterias acidófilas altamente resistente al cobre
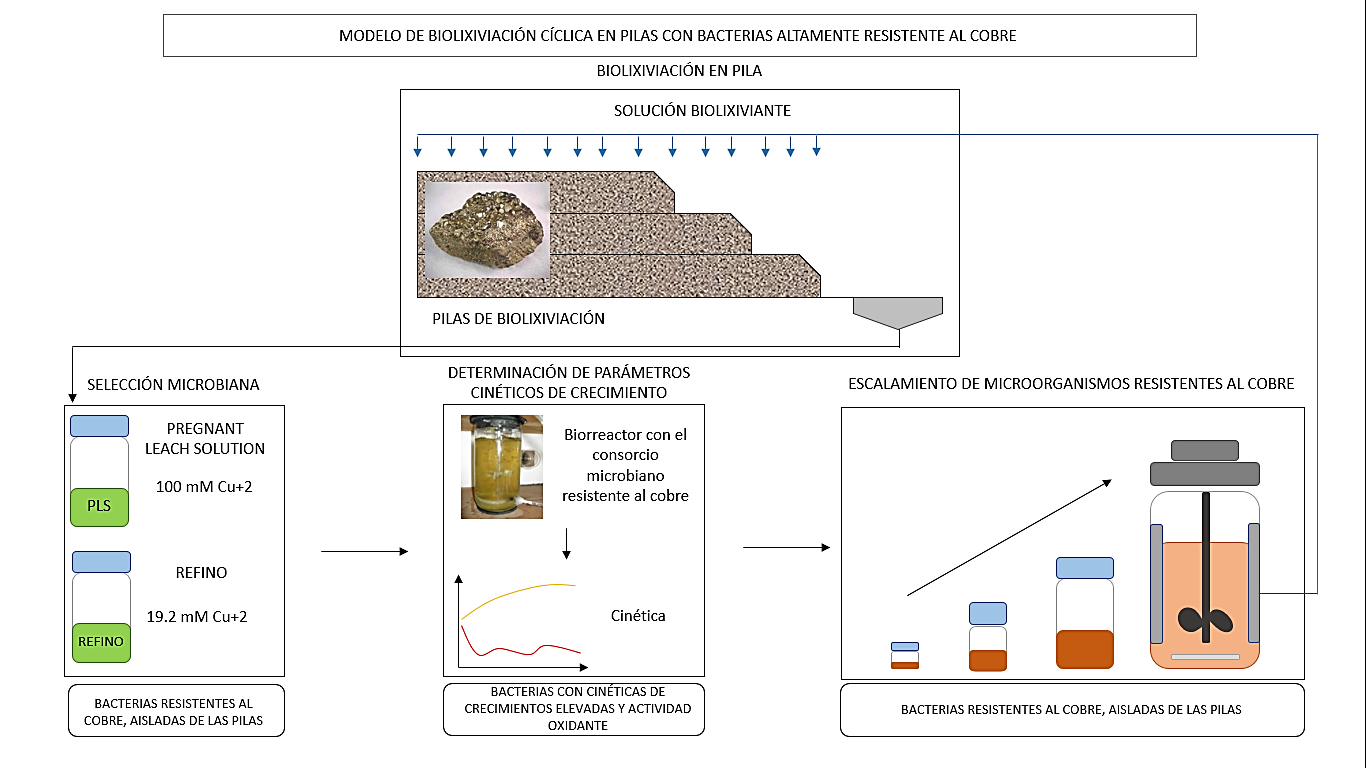
Figura 7. Modelo de Biolixiviación cíclica en pilas usando bacterias altamente resistentes al cobre
5. CONCLUSIONES
El consorcio conformado por A. ferrooxidans y Sulfobacillus sp, es resistente a altas concentraciones de cobre (1,000 mM) y constituye un modelo para estudios de alta resistencia al cobre puesto que puede ser usado en procesos de biolixiviacion con contenidos superiores a 1000 mM de cobre, además se determina que estas especies pueden ser un bioindicador de altas concentraciones de cobre en drenajes ácidos de minas, PLS y relaves en operaciones industriales mineras donde se procese minerales sulfuros de cobre.
6. FUENTES DE FINANCIAMIENTO
Fondo de Investigación para Tesis de Pregrado-UA 2015 MEM; ATI15-02/ ATI15-03 y Proyecto Semillero 5302 MEM de la Universidad de Antofagasta-Chile y fuentes propias de los autores.
7. AGRADECIMIENTOS
A todas las personas que contribuyeron de una u otra forma al desarrollo de este trabajo en los laboratorios de Biotecnología de la Facultad de Ciencias Biológicas de la Universidad Nacional de Trujillo-Perú y al Laboratorio de Biominería de la Facultad de Ciencias del Mar de la Universidad de Antofagasta.
8. REFERENCIAS BIBLIOGRÁFICAS
1. Madigan M., Brender K., Buckley D., Sattley W., y Stahl D. Brock Biology of Microorganisms. (2019). Edit. Pearson. Southern Illinois University Carbondate. 15ava Edición.
2. Medina S., Torres M., Durán Y., Ramírez R., Herrera J., y Ramírez P. (2012). Degradación de tiocianato por hongos aislados de ambientes mineros y evaluación de su capacidad degradativa. Revista Peruana de Biología. 19(1), 81-88.
3. Watling H. R. (2006). The bioleaching of sulphide minerals with emphasis on copper sulphides a review. Hydrometallurgy. 84 (1-2), 81-108. DOI: 10.1016/j.hydromet.2006.05.001
4. Latorre M., Cortés M. P., Travisany D., Di Genova A., Budinich M., Reyes-Jara A. y Cambiazo V. (2016). The bioleaching potential of a bacterial consortium. Bioresource technology. 218, 659-666. DOI: 10.1016/j.biortech.2016.07.012
5. Morales F. A. (2016). Evaluación técnico-económica de la implementación de un nuevo plan de riego de los sectores de lixiviación en la operación Mantoverde-Anglo American. Memoria para obtener el título de Ing Civil Químico. Datos tomados de Anglo American. Datos Clave Operación Mantoverde (www.angloamerican-chile.cl). Santiago de Chile. Chile.
6. Jia Y., Sun H., Chen D., Gao H., y Ruan R. (2016). Characterization of microbial community in industrial bioleaching heap of copper sulfide ore at Monywa mine, Myanmar. Hydrometallurgy 164, 355-361. DOI: 10.1016/j.hydromet.2016.07.007
7. Neciosup E., Vergara M., Pairazamán O., Apablaza M., y Esparza M. (2015). Cobre antimicrobiano contra patógenos intrahospitalarios en Perú. In Anales de la Facultad de Medicina. 76. 1. 9-14. DOI: 10.15381/anales.v76i1.11069
8. Xie X., Xiao S., He Z., Liu J., y Qiu G. (2007). Microbial populations in acid mineral bioleaching systems of Tong Shankou Copper Mine, China. Journal of applied microbiology. 103(4), 1227-1238. DOI: 10.1111/j.1365-2672.2007.03382.x
9. Watling H. R., Collinson D. M., Li J., Mutch L. A., Perrot F. A., Rea S. M., y Watkin E. L. J. (2014). Bioleaching of a low-grade copper ore, linking leach chemistry and microbiology. Minerals Engineering, 56, 35-44. DOI: 10.1016/j.mineng.2013.10.023
10. Demergasso C. S., Castillo D., y Casamayor E. O. (2005). Molecular characterization of microbial populations in a low-grade copper ore bioleaching test heap. Hydrometallurgy. 80(4), 241-253. DOI: 10.1016/j.hydromet.2005.07.013
11. Harvey P. I., y Crundwell F. K. (1996). The effect of As (III) on the growth of Thiobacillus ferrooxidans in an electrolytic cell under controlled redox potentials. Minerals Engineering, 9(10), 1059-1068. DOI: 10.1016/0892-6875(96)00098-2
12. Rawlings D. E., y Johnson D. B. (2007).The microbiology of biomining: development and optimization of mineral-oxidizing microbial consortia. Microbiology. 153(2), 315-324. DOI: 10.1099/mic.0.2006/001206-0
13. Watkin E. L. J., Keeling S. E., Perrot F. A., Shiers D. W., Palmer M. L., y Watling H. R. (2009). Metals tolerance in moderately thermophilic isolates from a spent copper sulfide heap, closely related to Acidithiobacillus caldus, Acidimicrobium ferrooxidans and Sulfobacillus thermosulfidooxidans. Journal of industrial microbiology & biotechnology. 36(3), 461. DOI: 10.1007/s10295-008-0508-5
14. Miranda H., Robles H., Villanueva L., y Rodríguez C. (2006). Biorreactores, Diseños y Aplicaciones. Sociedad Peruana de Biotecnología, Trujillo, Perú.
15. Allan J. E. (1961).The use of organic solvents in atomic absorption. Spectrophotometry. Spectrochimica Acta. 17(4), 467-473. DOI: 10.1016/0371-1951(61)80099-4
16. Schrader J. A., y Holmes D. S. (1988). Phenotypic switching of Thiobacillus ferrooxidans. Journal of bacteriology, 170(9), 3915-3923. DOI: 10.1128/jb.170.9.3915-3923.1988
17. Yates J. R., y Holmes D. S. (1987). Two families of repeated DNA sequences in Thiobacillus ferrooxidans. Journal of bacteriology, 169(5), 1861-1870. DOI: 10.1128/jb.169.5.1861-1870.1987
18. Lane D. J. (1991). 16S/23S rRNA sequencing. Nucleic acid techniques in bacterial systematics. 115-175.
19. De Wulf-Durand P., Bryant L. J., y Sly L. I. (1997). PCR-mediated detection of acidophilic, bioleaching-associated bacteria. Applied and environmental microbiology, 63(7), 2944-2948.
20. Dupont C. L., Butcher A., Valas R. E., Bourne P. E., y Caetano-Anollés G. (2010). History of biological metal utilization inferred through phylogenomic analysis of protein structures. Proceedings of the National Academy of Sciences, 107(23), 10567-10572. DOI: 10.1073/pnas.0912491107
21. Li X. Z., Plésiat P., y Nikaido H. (2015). The challenge of efflux-mediated antibiotic resistance in Gram-negative bacteria. Clinical microbiology reviews, 28(2), 337-418. DOI: 10.1128/CMR.00117-14
22. Saier Jr, M. H. (2003). Tracing pathways of transport protein evolution. Molecular microbiology. 48(5), 1145-1156. DOI: 10.1046/j.1365-2958.2003.03499.x
23. Jachlewski S., Jachlewski W. D., Linne U., Bräsen C., Wingender J., y Siebers B. (2015). Isolation of extracellular polymeric substances from biofilms of the thermoacidophilic archaeon Sulfolobus acidocaldarius. Frontiers in bioengineering and biotechnology. 3, 123. DOI: 10.3389/fbioe.2015.00123
24. San-Miguel A., y Martin-Gil F. J. (2009). Importancia de las especies reactivas al oxigeno (radicales libres) y los antioxidantes en clinica. Gaceta Médica de Bilbao, 106(3), 106-113. DOI: 10.1016/S0304-4858(09)74661-X
25. Castillo-Castañeda P. C., Gaxiola-Robles R., Labrada-Martagón V., Vargas B. A., Méndez-Rodríguez L. C., y Zenteno-Savín T. (2017) Oxidative damage to proteins related to metals and antioxidant defenses in breastmilk. Nutricion hospitalaria, 34(1), 59-64. DOI: 10.20960/nh.976
26. Cabrera G., Gomez J. M., y Cantero D. (2005). Kinetic study of ferrous sulphate oxidation of Acidithiobacillus ferrooxidans in the presence of heavy metal ions. Enzyme and Microbial Technology. 36(2-3), 301-306. DOI: 10.1016/j.enzmictec.2004.09.008
27. Nies D. H. (2003). Efflux-mediated heavy metal resistance in prokaryotes. FEMS microbiology reviews. 27(2-3), 313-339. DOI: 10.1016/S0168-6445(03)00048-2
28. Natarajan K. A., Sudeesha. K., y Rao, G. R. (1994). Stability of copper tolerance inThiobacillus ferrooxidans. Antonie van Leeuwenhoek. 66(4), 303-306. DOI: 10.1007/BF00882764
29. Navarro C. A., Orellana L. H., Mauriaca C., y Jerez C. A. (2009) Transcriptional and functional studies of Acidithiobacillus ferrooxidans genes related to survival in the presence of copper. Applied and Environmental Microbiology. 75(19), 6102-6109. DOI: 10.1128/AEM.00308-09
30. Rodríguez Y., Blázquez M. L., Ballester A., González F., y Muñoz J. A. (2001). La biolixiviación al comienzo del siglo XXI. Revista de metalurgia. 37(5), 616-627. DOI: 10.3989/revmetalm.2001.v37.i5.528